En protokoll for produksjon av Integrase dårlig Lentiviral vektorer for CRISPR/Cas9-mediert Gene Knockout i dele celler
Summary
Vi beskriver produksjon strategi integrase dårlig lentiviral vektorer (IDLVs) som kjøretøy for å levere CRISPR/Cas9 til cellene. Med en evne å megle rask og robust genet redigering i celler, IDLVs presenterer en tryggere og like effektivt vektor plattform for gen levering sammenlignet integrase-kompetent vektorer.
Abstract
Lentiviral vektorer er et ideelt valg for å levere gen-redigering komponenter til på grunn av deres evne til stabilt transducing et bredt spekter av celler og formidling av høye nivåer av genuttrykk. Men deres evne til å integrere i verten celle genomet øker risikoen for insertional genetisk virkning og dermed øker sikkerheten bekymringer og begrenser deres bruk i klinisk innstillinger. Videre levert vedvarende uttrykket av gen-redigering komponenter av disse integrasjon-kompetent lentiviral vektorer (ICLVs) øker sannsynligheten for promiskuøse genet målrette. Som et alternativ, har en ny generasjon integrase dårlig lentiviral vektorer (IDLVs) blitt utviklet som løser mange av disse bekymringene. Her produksjon protokollen av en ny og forbedret IDLV plattform for CRISPR-mediert genet redigering og listen trinnene involvert i rensing og konsentrasjonen av slike vektorer er beskrevet og signaltransduksjon og gen-redigering effektivitet ved hjelp HEK-293T cellene ble demonstrert. Denne protokollen er lett skalerbare og kan brukes til å generere høy titer IDLVs som kan transducing celler i vitro og i vivo. Dessuten, denne protokollen kan lett tilpasses for produksjon av ICLVs.
Introduction
Presis genet redigering utgjør hjørnesteinen i store biomedisinsk fremskritt som involverer utviklingen av romanen strategier for å takle genetiske sykdommer. I forkant av gen-redigering teknologiene er metoden stole på bruk av den clustered regularly –jegnterspaced short palindromic repeats (CRISPR) / Cas9 system som ble identifisert som en del av bakteriell immunitet mot invasjonen av viral genetiske materiale (omtalt i referanser1,2). En stor fordel av CRISPR/Cas9 over andre gen-redigeringsverktøy, som sink-finger nucleases (ZFNs) og transkripsjon aktivator som effektor nucleases (TALENs) (omtalt i referanse3), er den relative enkelheten av plasmider design og bygging av CRISPR komponenter, en funksjon som har drevet utvidelse av gen-redigering av noen få spesialiserte laboratories til en mye bredere forskningsmiljø. I tillegg har enkelheten til CRISPR/Cas9 programmering og dens kapasitet for multiplex målet anerkjennelse ytterligere fueled sin popularitet som en kostnadseffektiv og lett-å-bruke teknologi. Blant de ulike metodene tilgjengelig for forskere å levere slike gen-redigering komponenter til celler, fortsatt viral vektorer langt den mest populære og effektive systemet.
Lentiviral vektorer (LVs) har dukket opp som den ønsker å levere komponentene i CRISPR/Cas9 systemet i vivo for diverse programmer4,5,6,7. Flere viktige funksjoner gjør LVs et populært valg for denne prosessen inkluderer deres evne til å infisere både dele og ikke dele celler, lav immunogenisitet og minimal mobilnettet toksisitet (omtalt i referanse8). Som et resultat, har LV-mediert genterapi vært ansatt i behandling av infeksjonssykdommer, som HIV-1 og HBV HSV-1, og i korrigering av feil underliggende menneskelige arvelige sykdommer, som cystisk fibrose og neo-vaskulær macular degenerasjon 4 , 5 , 7 , 9 , 10 , 11. videre, LVs har endret effektivt for å utføre multiplex genet redigering på tydelige genomisk loci bruker en enkelt vektor systemet12.
Men den iboende egenskapen av LVs å integrere i vert genomet kan mutagene og ofte handicap deres nytte som transgene levering biler, spesielt i klinisk innstillinger. Videre siden stabilt integrert LVs uttrykke deres effekter av transgener på bærekraftig høye nivåer, er dette systemet dårlig tilpasset for levering av gen-redigering komponenter som CRISPR/Cas9; overuttrykte Cas9-guide RNA (gRNA) og lignende proteiner som ZFNs, er forbundet med forhøyede nivåer av off-målet effekter, blant annet uønsket mutasjoner13,14,15,16 , 17 og kan potensielt øke cytotoksisitet18. Derfor, for å oppnå presis gen-redigering med minimal off-målet effekter, er det viktig å design systemer som tillater for forbigående uttrykket av genet redigeringskomponenter.
De siste årene, en rekke levering plattformer er utviklet for å uttrykke transiently CRISPR/Cas9 i cellene16,19,20,21 (omtalt i referanse22). Disse omfatter metoder som bruker direkte innføre renset Cas9 sammen med riktig veiledning RNAs i celler, som viste seg å være mer effektiv på målrettet gen-redigering i forhold til plasmider-mediert transfection16. Studier har vist at ribonucleoprotein (RNP) komplekser bestående av guide RNA/Cas9 partikler er raskt snudd etter formidling DNA cleavage på sine mål, antyder at kortsiktige uttrykket av disse komponentene er tilstrekkelig for å oppnå robust genet redigering16. Tenkes, ikke integrert viral vektor plattformer som adeno-assosiert virus vektorer (AAVs) kan gi et levedyktig alternativ å levere gen-redigering maskiner til cellene. Dessverre AAV capsids har betydelig lavere emballasje kapasiteter enn LVs (< 5kb), som sterkt hindrer deres evne til å pakke multi-komponent CRISPR verktøysettet innenfor en enkelt vektor (omtalt i referanse8). Det er verdt å merke seg at tillegg av forbindelser som hemme histone deacetylases (f.eks, natrium butyrate23) eller hindre cellen syklus (f.eks, koffein24) har vist seg å øke lentiviral titers. Til tross for framskritt, er forbigående uttrykk systemer utviklet så langt fortsatt hindret av flere mangler, for eksempel lavere effektivitet, som fører til redusert viral titers, og lav signaltransduksjon effektiviteten av virus generert gjennom slike tilnærminger25.
Integrase dårlig lentiviral vektorer (IDLVs) representerer et stort fremskritt i utviklingen av genet levering biler, som de kombinerer emballasje evnen til LVs med fordelen av AAV-lignende episomal vedlikehold i celler. Disse funksjonene hjelper IDLVs i stor grad omgå de store problemene forbundet med å integrere vektorer, vis-à-vis kontinuerlig overuttrykte potensielt gentoksisk elementer og integrasjon-mediert genetisk virkning. Det ble tidligere vist at IDLVs kan endres med hell for å forbedre episomal gene expression26,27. Med hensyn til IDLV-mediert CRISPR/Cas9 levering begrenser lav produksjon titers og lavere uttrykk for episome-borne genomer i forhold til integrase-dyktige lentiviral systemer deres nytte som bona fide verktøy for å levere genomet-redigering transgene konstruksjoner. Vi har nylig vist at både transgene uttrykk og viral titers knyttet IDLV produksjon er betydelig forbedret ved inkludering av bindende områder for transkripsjon factor Sp1 i viral uttrykk kassett28. De endrede IDLVs støttet robust CRISPR-mediert genet redigering både i vitro (i HEK-293T celler) og i vivo (på etter mitotisk hjernen nevroner), mens inducing minimal off-målet mutasjoner sammenlignet med tilsvarende ICLV-mediert systemer28. Samlet, vi utviklet en roman, kompakt, alle-inne-ettall CRISPR toolkit gjennomført på en IDLV plattform og skissert ulike fordelene med å bruke slike en levering kjøretøy for forbedret genet redigering.
Her er produksjon protokollen av IDLV-CRISPR/Cas9 beskrevet, inkludert de ulike trinnene involvert i samlingen, renselse, konsentrasjon og titrering av IDLVs, samt strategier for å validere gen-redigering effekten av disse vektorer. Denne protokollen er lett skalerbare for å møte behovene til ulike etterforskere og er utformet for å kunne generere LV vektorer med titers i området fra 1 x 1010 transducing enheter (TU) / mL. Vektorer generert gjennom denne protokollen kan brukes til å effektivt infisere flere ulike celletyper, inkludert vanskelig å transduce embryonic stilk celler, blodkreft cellene (T-celler og makrofager) og kultivert og i vivo– injisert neurons. Videre er protokollen like godt egnet for produksjon av integrase-kompetent lentiviral vektorer i lignende mengder.
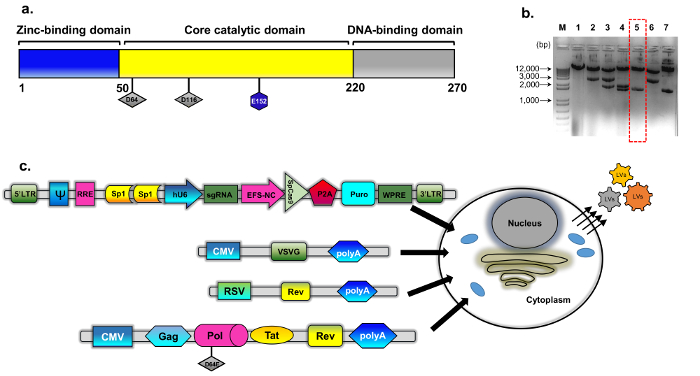
Figur 1: IDLV emballering. (a) skjematisk av wild type integrase protein (b) den endrede plasmider ble avledet fra psPAX2 (se metoder, plasmider konstruksjon for detaljer). Representant agarose gel bilde av kloner vist for muterte integrase kloner. DNA-prøver tilberedt med en standard plasmider DNA isolasjon mini kit ble analysert av fordøyelsen med EcoRV og SphI. Riktig fordøyd klone (nummer 5, stiplet rød boks) ble ytterligere bekreftet ved direkte (Sanger) sekvenser for D64E substitusjon i INT. Integrase-mangelfull emballasje kassetten ble kalt pBK43. (c) skjematisk av forbigående transfection protokollen ansatt til å generere IDLV-CRISPR/Cas9 vektorer, viser transfekterte 293T celler med VSV-G, emballasje og transgene kassetter (Sp1-CRISPR/Cas9 alt-i-ett plasmider). Virus partikler som bud ut fra cellemembranen inneholder den fulle RNA av vektoren (uttrykt fra transgene kassetten). Den andre generasjonen av IDLV-emballasje systemet ble brukt, som inkluderer regulatoriske proteiner Tat og rev Rev uttrykk er ytterligere supplert fra en egen kassett (RSV-REV-plasmider). ABBREV: LTR-lang-terminal gjenta, VSV-G, vesicular stomatitt virus G-protein, pCMV-cytomegalovirus promoter; Rous sarkomspesialitet virus (RSV) selskapet; RRE-(Rev svar Element). Andre regulerende elementer på uttrykk kassetten inkluderer Sp1 bindende områder, Rev Response element (RRE), hakkespett hepatitt Virus Posttranscriptional regulatorisk Element (WPRE), en kjerne-forlengelse faktor 1α promoter (EFS-NC), vektor emballasjen elementet ψ (psi), menneskelige Cytomegalovirus (hCMV) arrangøren og menneskelige U6 promoter. Klikk her for å se en større versjon av dette tallet.
Protocol
Representative Results
Discussion
IDLVs har begynt å dukke opp som bilen av valget for i vivo gen-redigering, spesielt i sammenheng med genetiske sykdommer, på grunn hovedsak til lav risiko for mutagenese forbundet med disse vektorer sammenlignet integrere levering plattformer22 , 28. i gjeldende manuskriptet, vi søkte på detaljer protokollen tilknyttet produksjonen av forbedret alt-i-ett IDLV-CRISPR/Cas9 systemet som ble nylig utviklet i vårt laboratorium28….
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi vil gjerne takke avdeling av Surgery, Duke University School of Medicine og Dekanus kontor for grunnleggende vitenskap, Duke University. Vi takker også medlemmer av Duke Viral vektor kjernen for kommentarer på manuskriptet. Plasmider pLenti CRISPRv2 var gave fra Feng Zhang (bred Institute). LV-pakkesystemet inkludert plasmider psPAX2, var VSV-G, pMD2.G og pRSV-Rev en slags gave fra Didier Trono (EPFL, Sveits). Økonomisk støtte til dette arbeidet ble levert av universitetet av South Carolina School Of Medicine, gi RDF18080-E202 (B.K).
Materials
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 – BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products – Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).

