התפוקה הגבוהה של DNA פלאגיסינג ומעבר באמצעות טכנולוגיית ננו-לחילוק אקוסטית
Summary
פרוטוקול זה מתאר את העברת הפלסטיות הגבוהה ביותר של תאי היונקים בצלחת 384-היטב באמצעות טכנולוגיית הפליטה האקוסטית של ה-droplet. הזמן גוזלת, שגיאות-DNA לחילוק ו ריבוב, אבל גם התרגום המקביל מגיב, הם תוכנה מונחה ומבוצעת על ידי מכשיר ננו. התאים הם הנזרע לאחר מכן בארות מלאות אלה.
Abstract
מעבר תאים, הכרחי למחקרים ביולוגיים רבים, דורש שליטה בפרמטרים רבים להשגת הישגים מדויקים ומוצלחים. בדרך כלל ביצוע בתפוקה נמוכה, זה יותר מדי זמן ושגיאות נוטה, אפילו יותר, כך כאשר ריבוב מספר פלמידים. פיתחנו שיטה קלה, מהירה ומדויקת לביצוע מעבר תאים בפריסת צלחת 384 באמצעות הוצאה אקוסטית של droplet (ADE). מכשיר ננו משמש במחקר זה מבוסס על טכנולוגיה זו ומאפשר משלוח מדויק ננונפח במהירות גבוהה מצלחת מקור היטב ליעד אחד. זה יכול לוותר על מולטיפלקס DNA ו-הזיהום מגיב לפי גיליון אלקטרוני מעוצב מראש. כאן אנו מציגים פרוטוקול אופטימלי כדי לבצע מבוסס אייד התפוקה הגבוהה בתפוקת האוויר הגבוה, מה שמאפשר להגיע ליעילות של עד 90% ו כמעט 100% הזיהום בניסויים הפנים. אנו מרחיבים את העבודה הראשונית על-ידי הצעת מאקרו ידידותי למשתמש המבוסס על גיליון אלקטרוני, מסוגל לנהל עד ארבע פלמידים/בארות מספרייה המכילה עד 1,536 שונים פלמידים, ויישום מדריך ליטוף מבוסס-לוח. המאקרו מעצב את התבניות הדרושות ללוחית המקור (ות) ויוצר את הקבצים המוכנים לשימוש עבור היישומים המבוססים על הננו-מנפק והלוח. פרוטוקול ארבעה שלבים החוצה כרוך i) מדלל לוותר עם מטפל הנוזל הקלאסי, ii) הפצה פלארמיד ו ריבוב, iii) מגיב החוצה על ידי הננו מנפק, ו iv) ציפוי תא על בארות מלאות. השליטה המבוססת על התוכנה של ריבוב הפלגיסינג והתרגום של ADE מאפשרת אפילו לא מומחים בתחום לבצע מעבר תאים אמין בדרך מהירה ובטוחה. שיטה זו מאפשרת זיהוי מהיר של הגדרות מיטביות עבור סוג תא נתון וניתן להשורבן לגישות בקנה מידה גבוה וידני. הפרוטוקול מקל על יישומים, כגון חלבון ORFeome אנושי (סט של מסגרות קריאה פתוח [ORFs] ביטוי הגנום) או CRISPR-Cas9-מבוסס על הפונקציה הגנטית אימות, באסטרטגיות סינון שאינם במאגר.
Introduction
השיטה המוצגת כאן מתארת בפרוטרוט כיצד לבצע בדיקת מידע והעברה של דנ א בתאי היונקים בתפוקה גבוהה באמצעות ננו-מתקן נוזלי מבוסס-אקוסטי בצלחת 384-באר, אפילו עבור שאינם מומחים בתחום. זו שיטה שפורסמה לאחרונה1 מאפשר לבצע ככל 384 באופן עצמאי מרבב ו-DNA בתנאי הזיהום בניסוי אחד, בתוך פחות מ 1 h. ניסויים בודדים או מעבר הצליחו, להגיע קרוב ל 100% זיהום בתוך אוכלוסיית התאים המזוהמים. פרוטוקול זה מאפשר העברה קלה יותר מכיוון שרוב השלבים המייקלים, הצורכים זמן ומועדים לשגיאות הם כעת מונחי תוכנה (ראה איור 1 עבור מבט כללי). מאמצים נוספים נעשו לפתח כלים ייעודיים להגברת קלות השימוש תוך הימנעות מטעויות אנוש במהלך התהליך הכולל ולקידום התפתחות מוצלחת גם עבור שאינם מומחים בתחום. הפרוטוקול המתואר כולל גיליון מאקרו “ידידותי למשתמש” שפיתחנו על מנת לנהל 384 מצבים בלתי תלויים עם אפשרויות ריבוב של עד ארבע פלמידים בכל באר. המאקרו מייצר באופן אוטומטי תבניות של לוחית המקור (s) כדי לטעון את ה-DNA הצפוי באמצעי האחסון להתחיל פתרונות המניה ואת הקבצים הדרושים כדי לכונן את התוכנה ננומנפק על העיצוב הניסיוני כי כבר inputted. כמו הידני מחלק של ה-DNA לוחית מקור 384-ובכן הוא מייגע ומועדים, אנו גם פיתח יישום ייעודי מבוסס לוח כדי להנחות את המשתמש בזמן לחלק פתרון DNA על פי התבנית.
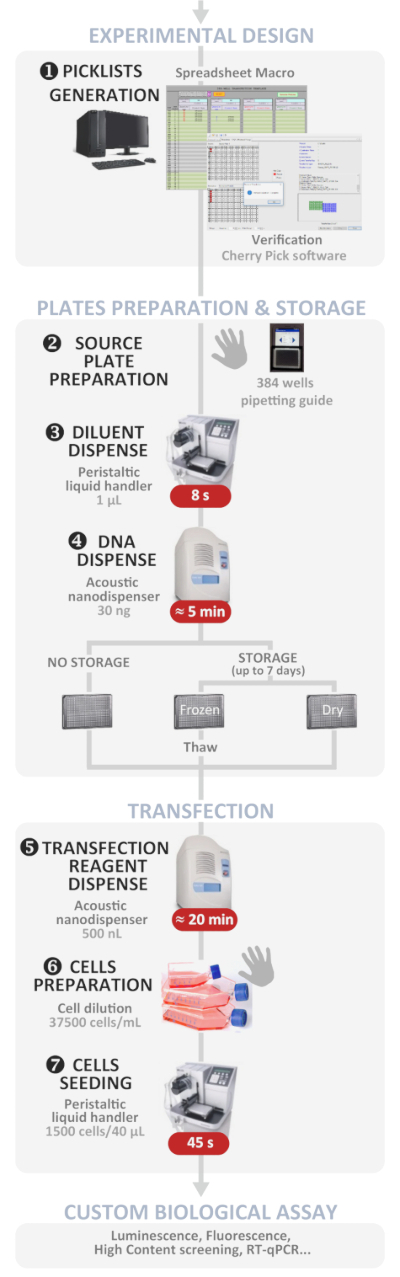
איור 1: זרימת עבודה ניסויית. ייצוג סכמטי של פרוטוקול העברת התפוקה האוטומטית האופטימלי האוטומטי (החל מהתכנון הנסיוני ועד לקבלת שיטת ביולוגית מותאמת אישית). שלבים ידניים מצוינים באמצעות סמל היד והזמן המשוער לכל שלב כתוב בתיבה אדומה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
רבים מבוססי תא ניסויים להתחיל עם התפתחות DNA פלמיד, וגם אם הרבה ריאגנטים ייעודי היו ועדיין מפותחים כדי לשפר את יעילות הטיפול ו/או להקל על ההליך, שנותר הרבה להיעשות2,3 , 4. DNA מעבר התא הזיהום כרוך כמה צעדים כדי להגיע ליעילות גבוהה, כגון ספיגה מורכבת ראשונית, בריחה אנדוזוממית, ו cytoplasmic הובלה לגרעין5,6. בנוסף סידן משקעים או טכניקות פיזיות כגון אלקטרופורציה או מיקרוהזרקה באמצעות מכשירים ייעודיים7, שיטות כימיות מודרניות התמקדו שיפור מסירת ה-DNA בזמן הורדת cytoxicity תא8, . בסדר, תשע השימוש בשומנים או פולימרים מיוצרים ליפובי מתחמים כמו, לאחרונה, מערכות כימיה פולימריים ליפוזוממית הפכה קלה ויעילה יותר10. למרות ההתפתחויות הללו, התפתחות התא עדיין דורש מיומנויות ספציפיות להתבצע באופן מדויק כמו רוב הפרוטוקולים הללו הפיזי או כימי מחייב מדענים להכין ידנית כל מצב תגובת דנ א, ולכן פוגע בתפוקה. כדי לעקוף בעיה זו, היפוך הפרוטוקולים הפוכים פותחו בעזרת ריאגנטים כימי העברת כימיקלים11,12,13, המאפשר למשתמש לבדוק או לשלב מספר פלמידים בצורה מהירה יותר. בפרוטוקולים אלה, חומצות גרעין מתחמים עם ריאגנטים החצייה נוצרות לפני זריעת התאים על התסביכים. עם זאת, פרוטוקולים אלה הפוכה עדיין מוגבלים על ידי טיפול ידני של פתרונות DNA ועל ידי שילוב של כל אחד מהתנאים העצמאיים. למרות שזה אפשרי לבצע אותם בפורמט הצלחת 96-באר, ההכנה DNA והיתר יהיה מייגע, וסביר להניח כי יהיו טעויות. כאשר כמויות שונות של מספר פלמיונים DNA נדרשים ומפולדלים אחד עם השני, הזיהום התא הופך אפילו קשה יותר להשיג יותר זמן רב, ושגיאות האדם להיות די בלתי נמנע. שינוי קנה מידה של עד לפורמט 384-באר בגישה הפוכה לאחור, למרות מספר התנאים הניתנים לשינוי בשיטת ה-DNA, הופך לאתגר בלתי אפשרי עקב הסיבות הבאות. i) כמויות ה-DNA, מגיב העברה, או התגובה תערובת אמצעי לניהול הם נמוכים מ-1 μL עבור כל באר. ii) ריבוב של פלמידים ל384 מצבים עצמאיים הופך למסובך ביותר. המסירה בכל אחד 384 בארות הוא גם iii) מאוד ארוך זמן ו iv) מועדת לשגיאות. ואכן, הפתרון הנכון בבארות הצפויות קשה לניהול מכיוון שאמצעי האחסון הנמוכים כבר אינם מאפשרים מעקב ויזואלי בין הבארות הריקות והמלאות. v) בסופו של דבר, יש סיכון גבוה לייבוש התערובת על ידי התאיידות לפני הוספת התאים בגלל הזמן הדרוש לביצוע הצעדים הדרושים לחילוק. לסיכום, הגורם המגביל להגדיר את ה-DNA התפוקה הגבוהה בשיטת הסינון מראה מופיע להיות המזעור של הטיפול, אשר מרמז על ריבוב בנפח נמוך וניהול כי לא ניתן לטפל באופן ידני יותר, אבל הם גם בקושי השגה ב דרך אמינה ע י מטפלים בנוזלים קלאסיים.
כהוכחה לקושי לספק מידע שכזה ולהשיג תפוקה גבוהה, רק נסיונות מעטים להפוך את החצייה לאוטומטית פורסמו עד כה: בתבנית צלחת 96-באר באמצעות מכשיר מסחרי טיפול נוזלי וסידן פוספט משקעים14 ו, לאחרונה, מגיב ליפולקס, ו שבב מיקרופלואידיג המאפשר 280 בלתי תלוי העברה של15 אבל דורש כישורים מיוחדים בתחום זה. שיטה נוספת, המאפשרת ריחוף נוזלי והובלת מניפולציה וערבוב נוזלים, נעשה שימוש כדי לבצע העברה של דנ א ב 24-כדי 96-צלחת לוחית בפורמטים16. למרות ריאלי, גישה זו סובלת תפוקה נמוכה מאוד כמו ערבוב של תאים עם התערובת DNA הזיהום דורש 60 s דגירה עבור כל נקודה אחת לפני זריעת. זה מרמז על משך של לפחות 96 דקות עבור צלחת מלאה 96-באר. יתר על כן, פרוטוקול זה הוא רחוק מלהיות קלה הקהל הכולל של הביולוגים כמו עבודה זו נעשתה עם מכשיר בבית מעוצב ומיוצר אשר כרגע לא זמין בשוק. להיפך, בשנים האחרונות, קל לשימוש מונחה תוכנה בטכנולוגיה מבוססת אקוסטי התפתחה עם מכשירי מנפק ננו נפח. באמצעות אנרגיה אקוסטית ממוקדת, התקנים אלה מאפשרים הוצאה בשליטה הדוקה של כרכים נוזליים קטנים מ 2.5 nL כדי 500 nL מלוחית המקור ליעד אחד17. טכנולוגיה זו, הנקראת לפליטה אקוסטית (ADE), היא בעלת יתרונות רבים: היא אוטומטית לחלוטין, ללא מגע, מדויקת, מדוייקת, מדויקת ומאוד מתאימה, והיא בעלת תפוקה גבוהה של18. המוקדש הראשון להעברת diמתיל סולפוקסיד (DMSO) פתרונות, הגדרות שופרו כדי לוותר על מאגרים מימית19. מכשירי ננו אקוסטי, לאחר מכן, נראה מתאים להפוך פרוטוקולים הפוכה תא החצייה והוא יכול לעקוף את רוב המגבלות הנ ל המדריך. מאחר שלא תוארו בעבר בטכנולוגיה זו, הערכנו לאחרונה את התאמתו של מערכת החילוק האקוסטית המבוססת לביצוע הפיכת תאים הפוכים.
ניצול תפוקת הננו וקלות השימוש, אנו אופטימיזציה פרוטוקול היפוך החוצה עבור תאי הלה על ידי החוצה בדיקה מספר פרמטרים שיכולים להשפיע על העברת דנ א על 384-ובכן, צלחת אחת, כלומר, סכום ה-dna סה כ ו מקור ה-DNA מתחיל ריכוז, מדלל נפח, מגיב העברה, ומספר תאים כפולה. הפרוטוקול המפותח חוסם את המגבלות הידניות שתוארו לעיל, ומציג מספר יתרונות על פני ניסיונות אחרים לשימוש אוטומטי. ראשית, זה מיניאטורי, ובכך מאפשר מגיב חסכונית הזיהום על ידי שמירת DNA ההכנות והזיהום התרגום. שנית, היא הרבה יותר גבוהה התפוקה והניתנת לקריאה מאשר הפרוטוקול הידני (אפילו למתחילים), כמו העברת הצלחת של 384 שלם-באר ניתן להשיג בפחות מ 1 h. לבסוף, זה מונחה תוכנה, המאפשר את השליטה של כמות ה-DNA מבוקר ואת ריבוב של מספר פלמידים. אכן, הודות לתוכנה ננו מנפק (טבלה של חומרים), המשתמש יכול להרחיב את תוכנית המחקר כדי לשלוט על אמצעי האחסון כדי להיות מוכן צלחת מקור מוגדר היטב ליעד אחד.
הפרוטוקול המוצג כאן מיועד בעיקר לאלה שיש להם גישה לננו-מנפק ורוצים להגדיר ניסויים בתפוקה גבוהה, אלא גם עבור אלה שרוצים למטב במהירות את הפרמטרים שלהם עבור סוג תא נתון על ידי החלת פרוטוקול זה כדי להצליב מספר פרמטרים בתפוקה גבוהה. אכן, הצגנו כי הפרמטרים אופטימיזציה מזוהה עם פרוטוקול זה ננו-סולם יכול להיות משתנה לניסויים בקנה מידה גדול וידני. בסופו של דבר, כמו מגיב החידוש המשמש בפרוטוקול הנוכחי מאפשר DNA או siRNA העברה על פי היצרן, הפרוטוקול הוא גם מעניין לאלה המכוונים לבצע גישות מערך עבור הבעות היתר של הגן או הנוק-אובר. לוחיות היעד המלאות מראש ב-DNA ניתן לשנות עד 7 ימים לפני השימוש בתוך שיטת החצייה ללא אובדן של יעילות, שהוא יתרון נוסף של הפרוטוקול הבא עבור סוג זה של יישום.
Protocol
Representative Results
Discussion
הקמתה והאופטימיזציה של שיטת העברה מדויקת של תפוקה גבוהה עבור קו תאים נתון מחייבת מדענים לעקוב אחר כמה פרמטרים מרכזיים המתוארים בסעיף זה. אנו מעודדים מאוד להתחיל עם הערכים המומלצים במהלך הפרוטוקול כמו הגדרות אלה אופטימיזציה עבור תאי הלה הוכיחו גם להיות יעיל עבור תאי HEK. עם זאת, ככל שהפרמטר?…
Declarações
The authors have nothing to disclose.
Acknowledgements
המחברים חשף קבלת התמיכה הפיננסית הבאה למחקר, מחבר ו/או פרסום מאמר זה: Inserm, אוניברסיטת ליל, מכון ליל-פסטר, Conseil Régional du Nord, ופרים-HCV1 ו-2 (שיא דה רצ’רצ’ה) למשל, סוכנות הידיעות הלאומית דה לה רצ’רצ’ה (ANR-10-04-01), הפדר (12001407 (ד-אל) ציוד לשעבר ביומד והקהילה האירופית (ERC-STG n ° 260901). המחברים רוצים להודות לד ר ש. מוראד, ד ר ב. וילרומאן, ד ר ר. פרירו-קלנט, וד ר ח. גראשט על הביקורת הקריטית והתיקונים של כתב היד.
Materials
| 384LDV Microplate | Labcyte | LP-0200 | |
| 384-well Microplate μClear Black | Greiner | 781906 | |
| Ampicilin | Sigma | A9393-5G | Selection antibiotic for bacteria transformed with ampicilin expressing vector |
| Android Tablet | Samsung | Galaxy Note 8 | used to guide the user while the source plate manual dispense |
| Aniospray Surf 29 | Anios | 2421073 | disinfectant to clean the MicroFlo head |
| Columbus software | Perkin Elmer | image analysis software | |
| Dulbecco's Modified Eagle Medium (DMEM), high glucose, GlutaMAX Supplement, pyruvate | Thermo Fisher Scientific | 10566032 | cell culture medium |
| Echo Cherry Pick 1.5.3 software | Labcyte | Software enabling ADE-based dispenses by the Echo550 device from a *.csv file; nanodispenser software | |
| Echo550 | Labcyte | ADE-based dispenser | |
| Fetal Bovine Serum | Thermo Fisher Scientific | 16000044 | to add in cell culture medium |
| Formalin solution, neutral buffered, 10% | Sigma-Aldrich | HT501128-4L | to fix cell |
| HeLa cells | ATCC | HeLa (ATCC® CCL-2™) | |
| Hoechst 33342, Trihydrochloride, Trihydrate | Thermo Fisher Scientific | H3570 | 10 mg/mL Solution in Water |
| INCell Analyzer 6000 | GE Healthcare | 29043323 | automated laser-based confocal imaging platform |
| LB medium | Thermoischer Scientific LB Broth Base (Lennox L Broth Base)®, powder |
12780052 | culture medium for bacteria growth |
| Lysis Buffer (A2) | Macherey-Nagel | 740912.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| MicroFlo 10µL cassette | Biotek Instruments Inc | 7170013 | to use with the Microflo Dispenser |
| MicroFlo 1μL cassette | Biotek Instruments Inc | 7170012 | to use with the Microflo Dispenser |
| MicroFlo Dispenser | Biotek Instruments Inc | 7171000 | peristaltic pump-based liquid handler device |
| Microvolume spectrophotometer | Denovix | DS-11 Spectrophotometer | Measure the DNA concentration of samples |
| mVenus plasmid | mVenus cDNA was cloned by enzymatic restriction digestion and ligation in Age1/BsrG1 sites of the tdTomato-N1 plasmid | Vector type: Mammalian Expression, Fusion Protein: mVenus | |
| Neutralization Buffer (A3) | Macherey-Nagel | 740913.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| NucleoSpin Plasmid kit | Macherey-Nagel | 740588.50 | used to prepare plasmid from bacterial culture |
| Optimal-Modified Eagle Medium (Opti-MEM) Medium | Thermo Fisher Scientific | 31985070 | |
| optional Wash bufferWash Buffer (A4) | Macherey-Nagel | 740914.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| orbital shaker | incubated large capacity shaker | 444-7084 | Used to grow bacteria under gentle agitation and 37°C |
| Penicillin-Streptomycin | Thermo Fisher Scientific | 15140122 | 10,000 U/mL |
| Phosphate-Buffered Saline | Thermo Fisher Scientific | 10010001 | |
| Plasmid mini-columns | Macherey-Nagel | 740499.250 | Silica membrane mini-column to prepare plasmid from bacterial culture |
| Resuspension Buffer (A1) | Macherey-Nagel | 740911.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| RNAse A | Macherey-Nagel | 740505 | Enzyme from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| tdTomato-N1 plasmid | Addgene | Plasmid #54642 | Vector type: Mammalian Expression, Fusion Protein: tdTomato |
| TransIT-X2 Dynamic Delivery System | Mirus Bio | MIR 6000 | |
| Wash Buffer (AW) | Macherey-Nagel | 740916.1 | Buffer from the NucleoSpin Plasmid kit used to prepare plasmid from bacterial culture |
| 3D printer | Creality | CR10S | used to print the plate adapter |
| Blender Software | https://www.blender.org/ Free software under GNU General Public License (GPL). |
version 2.79b | used to design the plate adapter |
Referências
- Colin, B., Deprez, B., Couturier, C. High-Throughput DNA Plasmid Transfection Using Acoustic Droplet Ejection Technology. SLAS Discovery: Advancing Life Sciences R & D. , 2472555218803064 (2018).
- Mirus Bio. . Optimising Transfection Performance. , (2019).
- Thermo Fisher Scientific. . Factors Influencing Transfection Efficiency | Thermo Fisher Scientific – FR. , (2019).
- Boussif, O., et al. A versatile vector for gene and oligonucleotide transfer into cells in culture and in vivo: polyethylenimine. Proceedings of the National Academy of Sciences of the United States of America. 92 (16), 7297-7301 (1995).
- Figueroa, E., et al. A mechanistic investigation exploring the differential transfection efficiencies between the easy-to-transfect SK-BR3 and difficult-to-transfect CT26 cell lines. Journal of Nanobiotechnology. 15 (1), 36 (2017).
- Kirchenbuechler, I., Kirchenbuechler, D., Elbaum, M. Correlation between cationic lipid-based transfection and cell division. Experimental Cell Research. 345 (1), 1-5 (2016).
- Zhang, Z., Qiu, S., Zhang, X., Chen, W. Optimized DNA electroporation for primary human T cell engineering. BMC Biotechnology. 18 (1), 4 (2018).
- Cao, D., et al. Transfection activity and the mechanism of pDNA-complexes based on the hybrid of low-generation PAMAM and branched PEI-1.8k. Molecular bioSystems. 9 (12), 3175-3186 (2013).
- Bos, A. B., et al. Development of a semi-automated high throughput transient transfection system. Journal of Biotechnology. 180, 10-16 (2014).
- Colosimo, A., et al. Transfer and expression of foreign genes in mammalian cells. BioTechniques. 29 (2), 314-318 (2000).
- Villa-Diaz, L. G., Garcia-Perez, J. L., Krebsbach, P. H. Enhanced transfection efficiency of human embryonic stem cells by the incorporation of DNA liposomes in extracellular matrix. Stem Cells and Development. 19 (12), 1949-1957 (2010).
- Sabatini, D. M. . Reverse transfection method. , WO2001020015A1 (2001).
- Raymond, C., et al. A simplified polyethylenimine-mediated transfection process for large-scale and high-throughput applications. Methods (San Diego, CA). 55 (1), 44-51 (2011).
- Junquera, E., Aicart, E. Recent progress in gene therapy to deliver nucleic acids with multivalent cationic vectors. Advances in Colloid and Interface Science. 233, 161-175 (2016).
- Woodruff, K., Maerkl, S. J. A High-Throughput Microfluidic Platform for Mammalian Cell Transfection and Culturing. Scientific Reports. 6, 23937 (2016).
- Vasileiou, T., Foresti, D., Bayram, A., Poulikakos, D., Ferrari, A. Toward Contactless Biology: Acoustophoretic DNA Transfection. Scientific Reports. 6, 20023 (2016).
- Hadimioglu, B., Stearns, R., Ellson, R. Moving Liquids with Sound: The Physics of Acoustic Droplet Ejection for Robust Laboratory Automation in Life Sciences. Journal of Laboratory Automation. 21 (1), 4-18 (2016).
- Grant, R. J., et al. Achieving accurate compound concentration in cell-based screening: validation of acoustic droplet ejection technology. Journal of Biomolecular Screening. 14 (5), 452-459 (2009).
- Sackmann, E. K., et al. Technologies That Enable Accurate and Precise Nano- to Milliliter-Scale Liquid Dispensing of Aqueous Reagents Using Acoustic Droplet Ejection. Journal of Laboratory Automation. 21 (1), 166-177 (2016).
- Day, R. N., Davidson, M. W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews. 38 (10), 2887-2921 (2009).
- Zielinski, D., Gordon, A., Zaks, B. L., Erlich, Y. iPipet: sample handling using a tablet. Nature Methods. 11 (8), 784-785 (2014).
- Brunner, S., et al. Cell cycle dependence of gene transfer by lipoplex, polyplex and recombinant adenovirus. Gene Therapy. 7 (5), 401-407 (2000).
- Nii, T., et al. Single-Cell-State Culture of Human Pluripotent Stem Cells Increases Transfection Efficiency. BioResearch Open Access. 5 (1), 127-136 (2016).
- Noonan, D. J., Henry, K., Twaroski, M. L. A High-Throughput Mammalian Cell-Based Transient Transfection Assay. Signal Transduction Protocols. 284, 051-066 (2004).
- . . Transfection | TransIT Transfection Reagents | Mirus Bio. , (2015).
- American Type Culture Collection. . General protocol for transfection of stem cells, primary cells, and continuous cell lines with ATCC TransfeX Transfection Reagent. , (2017).
- American Type Culture Collection. . Transfection Reagents for Nucleic Acid Transfer into ATCC Cells. , (2017).
- de Los Milagros Bassani Molinas, M., Beer, C., Hesse, F., Wirth, M., Wagner, R. Optimizing the transient transfection process of HEK-293 suspension cells for protein production by nucleotide ratio monitoring. Cytotechnology. 66 (3), 493-514 (2014).
- Promega. . FuGENE® 6 Transfection Reagent. , (2019).
- Olden, B. R., Cheng, Y., Yu, J. L., Pun, S. H. Cationic polymers for non-viral gene delivery to human T cells. Journal of Controlled Release: Official Journal of the Controlled Release Society. 282, 140-147 (2018).
- Park, E., Cho, H. B., Takimoto, K. Effective gene delivery into adipose-derived stem cells: transfection of cells in suspension with the use of a nuclear localization signal peptide-conjugated polyethylenimine. Cytotherapy. 17 (5), 536-542 (2015).
- Wood, R. W., Loomis, A. L. The physical and biological effects of high-frequency sound-waves of great intensity. The London, Edinburgh, and Dublin Philosophical Magazine and Journal of Science. 4 (22), 417-436 (1927).
- Mamat, U., et al. Eliminating Endotoxin at the Source – A Novel Competent Cell Line with Modified Lipopolysaccharide for Low-Endotoxin Plasmid Production. , (2014).
- Ivanova, N. V., Kuzmina, M. L. Protocols for dry DNA storage and shipment at room temperature. Molecular Ecology Resources. 13 (5), 890-898 (2013).
- Lesnick, J., Lejeune-Dodge, A., Ruppert, N., Jarman, C. . High-Precision Cell Dispensing with the Labcyte Echo® Liquid Handler. , (2017).
- Yang, X., et al. A public genome-scale lentiviral expression library of human ORFs. Nature Methods. 8 (8), 659-661 (2011).
- Peng, J., Zhou, Y., Zhu, S., Wei, W. High-throughput screens in mammalian cells using the CRISPR-Cas9 system. The FEBS journal. 282 (11), 2089-2096 (2015).

