Analyse de l’ARN de microdissection à capture laser pour analyse d’expression génique spécifique aux tissus spatiaux et temporels chez les plantes
Summary
Présenté ici est un protocole pour la microdésection laser-capture (LCM) des tissus végétaux. LCM est une technique microscopique pour isoler les zones de tissu d’une manière sans contamination. La procédure comprend la fixation des tissus, l’incorporation de paraffine, la sectionnement, l’extraction de LCM et d’ARN. L’ARN est utilisé dans l’analyse spécifique aux tissus en aval et résolue temporellement des transcriptomes.
Abstract
Le développement d’un organisme multicellulaire complexe est régi par des types de cellules distincts qui ont des profils transcriptionnels différents. Pour identifier les réseaux de régulation transcriptionnels qui régissent les processus de développement, il est nécessaire de mesurer les profils d’expression des gènes spatiaux et temporels de ces différents types de cellules. Par conséquent, il est essentiel de mieux comprendre le contrôle spatio-temporel de l’expression génique pour mieux comprendre comment les processus biologiques et développementaux sont réglementés. Ici, nous décrivons une méthode de microdissection de capture laser (LCM) pour isoler un petit nombre de cellules de trois organes d’embryon d’orge au cours d’un cours de temps pendant la germination suivie du profilage de transcription. La méthode consiste en la fixation des tissus, le traitement des tissus, l’incorporation de paraffine, la sectionnement, l’extraction de LCM et d’ARN suivie d’une PCR ou d’un RNA-seq en temps réel. Cette méthode nous a permis d’obtenir des profils spatiaux et temporels des transcriptomes d’organes de semence à partir de différents nombres de cellules (des dizaines à des centaines), fournissant une spécificité tissulaire beaucoup plus grande que les analyses typiques des tissus en vrac. À partir de ces données, nous avons pu définir et comparer les réseaux de réglementation transcriptionnels ainsi que prévoir les facteurs de transcription réglementaires des candidats pour les tissus individuels. La méthode doit s’appliquer à d’autres tissus végétaux avec un minimum d’optimisation.
Introduction
Le développement et la croissance des plantes impliquent l’action coordonnée des réseaux de régulation transcriptionnels au sein de différentes cellules qui existent dans un environnement cellulaire complexe. Pour comprendre l’activité de ces réseaux de réglementation, nous avons besoin de la connaissance de l’expression des gènes spatiaux et temporels dans différents types de cellules à travers les stades de développement. Cependant, les analyses de l’expression des gènes sont plus couramment effectuées dans des organes entiers ou des échantillons de tissus en vrac en raison du défi technique d’isoler et d’analyser un petit nombre de cellules. La méthode que nous décrivons ici a permis d’obtenir l’analyse spatiale et temporelle de transcritome tissulaire-spécifique en couplant LCM avec l’ARN-seq.
LCM a été développé il y a deux décennies par Emmert-Buck et ses collègues1. La technique a permis aux chercheurs d’isoler précisément les cellules individuelles ou les grappes de cellules de leur environnement en utilisant une visualisation microscopique directe et la manipulation avec un faisceau étroit laser1. Depuis lors, la méthode a été largement utilisée dans la biologie du cancer et la pathologie2,3. De nombreux groupes de recherche végétale ont également adapté LCM pour l’utilisation avec différentes espèces végétales et différents types de tissus4,5,6,7,8,9,10,11. Récemment, plusieurs articles ont également utilisé LCM sur les graines eudicot et monocoques pour étudier l’embryon, les endospermes et d’autres structures de semences pendant le développement des semences et la germination10,12,13. La plupart des autres méthodes d’isolement unicellulaire couramment utilisées telles que le micro-pipetage, le tri cellulaire, la séparation magnétique et les plates-formes microfluidiques dépendent de la digestion enzymatique ou de l’homogénéisation mécanique pour dissocier les cellules. Cela peut perturber l’expression des gènes, en introduisant des artefacts techniques qui confondent l’interprétation des données14,15. Ces méthodes exigent également une connaissance préalable des gènes marqueurs pour chaque type de cellule pour relier les cellules dissociées à leur emplacement spatial et au vrai type de cellule. Un autre groupe de techniques dépend de l’isolement basé sur l’affinité des structures subcellulaires au lieu de cellules entières, par exemple INTACT (Isolation des noyaux marqués dans les types de cellules) et TRAP (Transducing Ribosome Affinity Purification)16,17. Cependant, l’étiquetage et la purification des noyaux ou des ribosomes sont techniquement difficiles chez les espèces végétales qui n’ont pas de protocoles de transformation bien établis. LCM profite de la fixation rapide des tissus pour préserver les niveaux de transcription et l’identification histologique conventionnelle par visualisation directe des cellules dans leur contexte normal de tissu/organe, ce qui permet aux cellules discrètes d’être isolées dans un court laps de temps18,19.
Le protocole présenté ici est une méthode optimisée pour l’isolement de cellules spécifiques ou de types de cellules des sections tissulaires des graines de céréales, qui peuvent être appliquées à la plupart des cellules qui peuvent être identifiées histologiquement. LCM fournit une méthode sans contact d’isolement cellulaire, réduisant considérablement la contamination et augmentant l’intégrité de l’ARN récupéré. En outre, la méthode illustre la puissance du LCM sur les études à grande échelle du génome à partir de petites quantités de matériaux biologiques. Nous décrivons également l’amplification linéaire de l’ARN pour produire suffisamment de matériel d’entrée pour les analyses de transcription/transcription.
Il y a dix étapes principales dans ce protocole d’ARN-seq de LCM pour les transcriptomes spatiaux et temporels spécifiques aux tissus, y compris la fixation des échantillons de tissu, la déshydratation, l’infiltration de paraffine, l’incorporation, la section, le LCM, l’extraction d’ARN, l’amplification d’ARN, la quantification de l’ARN et le qRT-PCR et/ou l’ARN-seq (Figure 1).
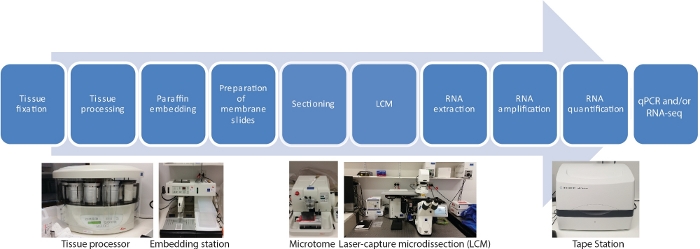
Figure 1 : Organigramme de LCM suivi de l’ARN-seq ou du qRT-PCR. LCM est une technique spatialement précise et sans contact pour recueillir les cellules à partir de sections de tissus fixes à l’aide d’un faisceau laser sous visualisation microscopique. Le processus commence par la fixation d’échantillons de tissus, suivi d’une déshydratation à l’aide d’une série de gradients d’éthanol et de xylène, et s’est terminé par une infiltration de paraffine. Le processus peut être entièrement automatisé à l’aide d’un processeur de tissus. Une fois que le tissu est infiltré avec de la paraffine, il est incorporé dans un moule avec de la paraffine en fusion à l’aide d’une station d’intégration. La section est effectuée à l’aide de microtomes réglés à l’épaisseur désirée. Les diapositives sont préparées et LCM effectuée immédiatement avant que l’ARN ne soit extrait des cellules capturées. L’extraction de l’ARN est suivie directement de deux cycles d’amplification de l’ARN avant qRT-PCR et/ou ARN-seq. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Protocol
Representative Results
Discussion
De nombreuses études d’expression génétique spécifiques aux tissus ont été limitées par la dissection à la main d’échantillons, qui prend beaucoup de temps, qui demande beaucoup de main-d’œuvre, présente un risque élevé de contamination et ne peut utiliser que des échantillons qu’un agent humain est suffisamment habile pour récolter. LCM est une technique précise et sans contact pour recueillir les cellules à partir de sections de tissus fixes à l’aide d’un faisceau laser actionné mécaniqu…
Declarações
The authors have nothing to disclose.
Acknowledgements
Ces travaux ont été appuyés par le Australian Research Council Centre of Excellence in Plant Energy Biology (CE140100008) à JW. M.G.L a reçu le soutien d’une subvention de départ de l’Université La Trobe. Nous remercions la Plate-forme de génomique la Trobe pour son soutien dans le séquençage à haut débit et l’analyse des données. Nous remercions le professeur agrégé Matthew Tucker pour les conseils d’experts sur l’établissement de LCM dans notre laboratoire.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Referências
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

