पौधों में स्थानिक और लौकिक ऊतक-विशिष्ट जीन अभिव्यक्ति विश्लेषण के लिए लेजर-कैप्चर माइक्रोडिसेक्शन आरएनए-अनुक्रमण
Summary
यहां प्रस्तुत पौधों के ऊतकों के लेजर-कैप्चर माइक्रोडिसेक्शन (एलसीएम) के लिए एक प्रोटोकॉल है। एलसीएम ऊतक के क्षेत्रों को संदूषण मुक्त तरीके से अलग करने के लिए एक सूक्ष्म तकनीक है। प्रक्रिया में ऊतक निर्धारण, पैराफिन एम्बेडिंग, सेक्शनिंग, एलसीएम और आरएनए निष्कर्षण शामिल हैं। आरएनए का उपयोग डाउनस्ट्रीम ऊतक-विशिष्ट, ट्रांसक्रिप्टोम के अस्थायी रूप से हल किए गए विश्लेषण में किया जाता है।
Abstract
एक जटिल बहुकोशिकीय जीव का विकास अलग-अलग कोशिका प्रकारों द्वारा नियंत्रित होता है जिनमें अलग-अलग ट्रांसक्रिप्शनल प्रोफाइल होते हैं। विकासात्मक प्रक्रियाओं को नियंत्रित करने वाले ट्रांसक्रिप्शनल नियामक नेटवर्कों की पहचान करने के लिए इन व्यक्तिगत सेल प्रकारों के स्थानिक और लौकिक जीन अभिव्यक्ति प्रोफाइल को मापना आवश्यक है। इसलिए, जैविक और विकासात्मक प्रक्रियाओं को विनियमित कैसे किया जाता है, इसकी समझ हासिल करने के लिए जीन अभिव्यक्ति के स्थानिक-लौकिक नियंत्रण में अंतर्दृष्टि आवश्यक है। यहां, हम अंकुरण के दौरान एक समय-पाठ्यक्रम पर तीन जौ भ्रूण अंगों से कोशिकाओं की छोटी संख्या को अलग करने के लिए एक लेजर-कैप्चर माइक्रोडिसेक्शन (एलसीएम) विधि का वर्णन करते हैं जिसके बाद ट्रांसक्रिप्ट प्रोफाइलिंग होती है। विधि ऊतक निर्धारण, ऊतक प्रसंस्करण, पैराफिन एम्बेडिंग, सेक्शनिंग, एलसीएम और आरएनए निष्कर्षण के बाद वास्तविक समय पीसीआर या आरएनए-एसईक्यू के होते हैं। इस विधि ने हमें कोशिकाओं की अलग-अलग संख्या (दसियों से सैकड़ों) से बीज अंग ट्रांसक्रिप्टोम के स्थानिक और लौकिक प्रोफाइल प्राप्त करने में सक्षम बनाया है, जो विशिष्ट थोक-ऊतक विश्लेषणों की तुलना में बहुत अधिक ऊतक-विशिष्टता प्रदान करता है। इन डेटा से हम प्रतिलेखन नियामक नेटवर्क को परिभाषित करने और तुलना करने में सक्षम थे और साथ ही व्यक्तिगत ऊतकों के लिए उम्मीदवार नियामक प्रतिलेखन कारकों की भविष्यवाणी करते थे। विधि न्यूनतम अनुकूलन के साथ अन्य पौधे ऊतकों पर लागू होनी चाहिए।
Introduction
संयंत्र विकास और विकास में विभिन्न कोशिकाओं के भीतर ट्रांसक्रिप्शनल नियामक नेटवर्क की समन्वित कार्रवाई शामिल है जो एक जटिल सेलुलर वातावरण में मौजूद हैं। इन विनियामक नेटवर्कों की गतिविधि को समझने के लिए, हमें विकासात्मक चरणों में विभिन्न सेल प्रकारों के भीतर स्थानिक और लौकिक जीन अभिव्यक्ति के ज्ञान की आवश्यकता होती है । हालांकि, जीन अभिव्यक्ति के विश्लेषण अधिक आमतौर पर कोशिकाओं की छोटी संख्या को अलग करने और विश्लेषण करने की तकनीकी चुनौती के कारण पूरे अंगों या थोक ऊतक नमूनों में आयोजित किए जाते हैं । हम यहां जिस विधि का वर्णन करते हैं, उसने आरएनए-एसईक्यू के साथ एलसीएम को युग्मन करके स्थानिक और लौकिक ऊतक-विशिष्ट ट्रांसक्रिप्टोम विश्लेषण प्राप्त करने की अनुमति दी है।
एलसीएम को दो दशक पहले Emmert-Buck और सहयोगियों द्वारा विकसित किया गया था1। इस तकनीक ने शोधकर्ताओं को सीधे सूक्ष्म दृश्य और संकीर्ण बीम लेजर 1 के साथ हेरफेर का उपयोग करके अपने पर्यावरण से एकल कोशिकाओं या कोशिकाओं के समूहों को ठीक से अलग करने में सक्षमबनाया। तब से इस विधि का व्यापक रूप से कैंसर जीव विज्ञान और पैथोलॉजी2,3 में उपयोग कियाजाता रहाहै . कई पादप अनुसंधान समूहों ने एलसीएम को विभिन्न पादप प्रजातियों और विभिन्न ऊतकों के प्रकार 4,,,5, 6 , 7,8,,,96,10,11के साथ उपयोगकेलिए भी अनुकूलित कियाहै। हाल ही में, बीज विकास और अंकुरण,,10, 12,13के दौरान भ्रूण, एंडोस्पर्म और अन्य बीज संरचनाओं का अध्ययन करने के लिए यूडिकॉट और मोनोकॉट बीजों पर कई पत्रों का भी उपयोग किया गया है।13 माइक्रो-पिपटिंग, सेल छंटाई, चुंबकीय पृथक्करण और माइक्रोफ्लुइडिक प्लेटफॉर्म जैसे अन्य आमतौर पर उपयोग किए जाने वाले अधिकांश एकल-कोशिका अलगाव विधियों को कोशिकाओं को अलग करने के लिए एंजाइमैटिक पाचन या यांत्रिक समरूपता पर निर्भर करता है। यह जीन अभिव्यक्ति को परेशान कर सकता है, तकनीकी कलाकृतियों को पेश कर सकता है जो डेटा व्याख्या14,,15को चकित करते हैं। इन तरीकों को भी प्रत्येक कोशिका प्रकार के लिए मार्कर जीन के पिछले ज्ञान की आवश्यकता को अपने स्थानिक स्थान और सही सेल प्रकार के लिए अलग कोशिकाओं से संबंधित है । तकनीकों का एक और समूह पूरी कोशिकाओं के बजाय उपकोशिकीय संरचनाओं के आत्मीयता-आधारित अलगाव पर निर्भर करता है, उदाहरण के लिए बरकरार (सेल प्रकारों में टैग किए गए नाभिक का अलगाव) और ट्रैप (राइबोसोम आत्मीयता शुद्धि का अनुवाद)16,,17। हालांकि, नाभिक या राइबोसोम्स की आत्मीयता लेबलिंग और शुद्धि तकनीकी रूप से पौधों की प्रजातियों में चुनौतीपूर्ण है जिनके पास अच्छी तरह से स्थापित परिवर्तन प्रोटोकॉल नहीं हैं। एलसीएम अपने सामान्य ऊतक/अंग संदर्भ के भीतर कोशिकाओं के प्रत्यक्ष दृश्य द्वारा ट्रांसक्रिप्ट स्तर और पारंपरिक हिस्टोलॉजिकल पहचान को संरक्षित करने के लिए त्वरित ऊतक निर्धारण का लाभ उठाता है, जो असतत कोशिकाओं को18,,19के समय की थोड़ी अवधि में अलग-थलग करने की अनुमति देता है ।
यहां प्रस्तुत प्रोटोकॉल अनाज बीजों के ऊतक वर्गों से विशिष्ट कोशिकाओं या कोशिका प्रकारों के अलगाव के लिए एक अनुकूलित विधि है, जिसे अधिकांश कोशिकाओं पर लागू किया जा सकता है जिन्हें हिस्टोलॉजिकल रूप से पहचाना जा सकता है। एलसीएम सेल अलगाव की एक संपर्क मुक्त विधि प्रदान करता है, जो संदूषण को कम करता है और बरामद आरएनए की अखंडता को बढ़ाता है। इसके अलावा, विधि जैविक सामग्रियों की छोटी मात्रा के साथ शुरू बड़े पैमाने पर जीनोम व्यापक अध्ययन पर एलसीएम की शक्ति दिखाता है । हम डाउनस्ट्रीम ट्रांसक्रिप्ट/ट्रांसक्रिप्टोम विश्लेषणों के लिए पर्याप्त इनपुट सामग्री उत्पन्न करने के लिए आरएनए के रैखिक प्रवर्धन का भी वर्णन करते हैं ।
स्थानिक और लौकिक ऊतक विशिष्ट प्रतिलिपि के लिए इस एलसीएम आरएनए-एसईक्यू प्रोटोकॉल में दस मुख्य कदम हैं, जिनमें ऊतक नमूनों का निर्धारण, निर्जलीकरण, पैराफिन घुसपैठ, एम्बेडिंग, सेक्शनिंग, एलसीएम, आरएनए निष्कर्षण, आरएनए प्रवर्धन, आरएनए क्वांटिफिकेशन और क्यूआरटी-पीसीआर और/या आरएनए-सेक्यू(चित्रा 1)शामिल हैं ।
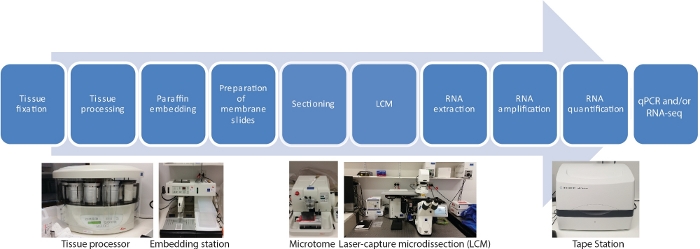
चित्रा 1: एलसीएम का फ्लोचार्ट इसके बाद आरएनए-सेक्यू या क्यूआरटी-पीसीआर। एलसीएम सूक्ष्म दृश्य के तहत लेजर बीम का उपयोग करके निश्चित ऊतक वर्गों से कोशिकाओं को इकट्ठा करने के लिए एक स्थानिक सटीक और संपर्क मुक्त तकनीक है। प्रक्रिया ऊतक नमूनों के निर्धारण के साथ शुरू होती है, जिसके बाद इथेनॉल और जाइलीन की ढाल श्रृंखला का उपयोग करके निर्जलीकरण होता है, और पैराफिन घुसपैठ के साथ समाप्त होता है। इस प्रक्रिया को पूरी तरह से एक ऊतक प्रोसेसर का उपयोग करके स्वचालित किया जा सकता है। एक बार जब ऊतक पैराफिन के साथ घुसपैठ हो जाती है, तो यह एम्बेडिंग स्टेशन का उपयोग करके पिघला हुआ पैराफिन के साथ एक मोल्ड में एम्बेडेड होता है। वांछित मोटाई के लिए माइक्रोटोम सेट का उपयोग करके सेक्शनिंग की जाती है। स्लाइड तैयार कर रहे है और एलसीएम तुरंत पहले आयोजित आरएनए कब्जा कर लिया कोशिकाओं से निकाला जाना है । आरएनए निष्कर्षण सीधे क्यूआरटी-पीसीआर और/या आरएनए-seq से पहले आरएनए प्रवर्धन के दो दौर से पीछा किया जाता है । कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
Protocol
Representative Results
Discussion
कई ऊतक विशिष्ट जीन अभिव्यक्ति अध्ययन नमूनों के हाथ विच्छेदन द्वारा सीमित किया गया है, जो समय लेने वाली है, श्रम गहन, संदूषण का एक उच्च जोखिम है और केवल नमूनों का उपयोग कर सकते है कि एक मानव ऑपरेटिव पर्याप…
Declarações
The authors have nothing to disclose.
Acknowledgements
इस काम को ऑस्ट्रेलियन रिसर्च काउंसिल सेंटर ऑफ एक्सीलेंस इन प्लांट एनर्जी बायोलॉजी (CE140100008) ने जेडब्ल्यू को सपोर्ट किया । M.G.L एक ला Trobe विश्वविद्यालय अनुदान शुरू द्वारा समर्थित था । हम ला ट्रोब जीनोमिक्स प्लेटफॉर्म को उच्च-थ्रूपुट अनुक्रमण और डेटा विश्लेषण में उनके समर्थन के लिए धन्यवाद देते हैं। हम अपनी प्रयोगशाला में एलसीएम की स्थापना पर विशेषज्ञ सलाह के लिए एसोसिएट प्रोफेसर मैथ्यू टकर को धन्यवाद देते हैं ।
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Referências
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

