레이저 캡처 미세 절 RNA-시퀀싱 용 공간 및 측두 조직 -특정 유전자 발현 분석
Summary
여기에 제시된 것은 식물 조직의 레이저 포획 미세 절(LCM)을 위한 프로토콜입니다. LCM은 오염없는 방식으로 조직의 영역을 격리하기위한 현미경 기술입니다. 절차는 조직 고정, 파라핀 포함, 단면, LCM 및 RNA 추출을 포함한다. RNA는 다운스트림 조직 특이적, 전사의 현세적으로 해결된 분석에서 사용된다.
Abstract
복잡한 다세포 유기체의 발달은 상이한 전사 단면도가 있는 명백한 세포 모형에 의해 지배됩니다. 발달 과정을 제어하는 전사 적 규제 네트워크를 식별하려면 이러한 개별 세포 유형의 공간 및 측두성 유전자 발현 프로파일을 측정할 필요가 있습니다. 따라서 유전자 발현의 현세-측량 제어에 대한 통찰력은 생물학적 및 발달 과정이 어떻게 규제되는지 이해하는 데 필수적입니다. 여기서는 발아 중에 시간 과정을 통해 3개의 보리 배아 기관에서 소수의 세포를 분리하는 레이저 포획 미세 절(LCM) 방법을 설명하고 전사체 프로파일링을 실시한다. 이 방법은 조직 고정, 조직 처리, 파라핀 포함, 단면, LCM 및 RNA 추출 다음으로 실시간 PCR 또는 RNA-seq로 구성됩니다. 이 방법을 통해 종자 장기 전사의 공간적 및 측두프로파일을 다양한 수의 세포(수십~수백 개)에서 얻을 수 있게 되어 일반적인 벌크 조직 분석보다 훨씬 더 큰 조직 특이성을 제공합니다. 이러한 데이터에서 우리는 전사 적 규제 네트워크를 정의하고 비교할 뿐만 아니라 개별 조직에 대한 후보 규제 전사 요인을 예측할 수 있었습니다. 이 방법은 최소한의 최적화를 가진 다른 플랜트 조직에 적용되어야 합니다.
Introduction
식물 개발 및 성장은 복잡한 세포 환경에 존재하는 다른 세포 내의 전사 규제 네트워크의 조정 된 작용을 포함한다. 이러한 규제 네트워크의 활동을 이해하기 위해, 우리는 발달 단계에 걸쳐 다른 세포 모형 내의 공간 및 측두성 유전자 발현의 지식을 요구합니다. 그러나, 유전자 발현의 분석은 세포의 작은 숫자를 격리하고 분석의 기술적 인 도전 때문에 전체 기관 또는 대량 조직 샘플에서 더 일반적으로 수행됩니다. 여기서 설명하는 방법은 LCM과 RNA-seq를 결합하여 공간 및 측두조직 특이적 전사 분석을 얻을 수 있게 하였다.
LCM은 에머트 벅과 동료에머트벅에 의해 이십 년 전에 개발되었다 1 . 이 기술을 통해 연구자들은 좁은 빔 레이저1을사용하여 직접 현미경 시각화 및 조작을 사용하여 단일 세포 또는 세포 클러스터를 환경으로부터 정확하게 분리할 수 있었습니다. 그 이후로 이 방법은 암 생물학 및 병리학2,,3에서널리 사용되고 있다. ,많은 식물 연구 그룹은 또한 다른 식물 종 및 다른 조직 유형,4,,5,,6,7,8,9,10,11과함께 사용하기 위해,LCM을적응시켰다. 최근에는, 몇몇 논문은 또한 종자 발달 및 발아 도중 배아, 내배식 및 그밖 종자 구조물을 공부하기 위하여 eudicot 및 단장화장 씨앗에LCM을 이용했습니다10,,12,13. 마이크로 파이펫팅, 세포 선별, 자기 분리 및 미세 유체 플랫폼과 같은 다른 일반적으로 사용되는 단일 세포 격리 방법의 대부분은 세포를 해리하기 위해 효소 소화 또는 기계적 균질화에 의존한다. 이는 데이터해석(14,15)을혼동하는 기술 적 유물을 도입하여 유전자발현을방해할 수 있다. 이러한 방법은 또한 해리된 세포를 공간 위치 및 진정한 세포 유형에 연관시키기 위해 각 세포 유형에 대한 마커 유전자에 대한 이전 지식을 요구합니다. 기술의 추가 그룹은 전체 세포 대신 세포 체형 구조의 친화성 기반 절연에 따라 달라집니다, 예를 들어 INTACT (세포 유형에서 태그 핵의 격리) 및 TRAP (리보솜 친화 정화 번역)16,,17. 그러나 핵 이나 리보좀의 선호도 라벨링 및 정제는 잘 확립 된 변형 프로토콜이없는 식물 종에서 기술적으로 도전적입니다. LCM은 짧은기간동안 이산 세포를 단기간에 격리할 수 있도록 하는 정상 조직/장기 문맥 내에서 세포의 직접 시각화에 의한 성적증명서 수준 및 종래의 조직학적 식별을 보존하기 위해 빠른 조직 고정을활용한다.19
여기에 제시된 프로토콜은 조직학적으로 식별될 수 있는 대부분의 세포에 적용될 수 있는 시리얼 씨앗의 조직 섹션에서 특정 세포 또는 세포 유형의 분리를 위한 최적화된 방법입니다. LCM은 접촉없는 세포 격리 방법을 제공하여 오염을 크게 줄이고 회수 된 RNA의 무결성을 증가시킵니다. 더욱이, 이 방법은 소량의 생물학적 물질로 시작하는 대규모 게놈 넓은 연구에 대한 LCM의 힘을 보여줍니다. 우리는 또한 다운스트림 전사/전사 분석을 위한 충분한 입력 물질을 생성하기 위한 RNA의 선형 증폭을 기술합니다.
이 LCM RNA-seq 프로토콜에는 조직 샘플, 탈수, 파라핀 침투, 임베딩, 단면, LCM, RNA 추출, RNA 증폭, RNA 정량화 및 qRT-PCR 및/또는 RNA-seq(그림1)의고정을 포함하는 공간 및 측두조직 특이적 전사에 대한 10가지 주요 단계가 있습니다.
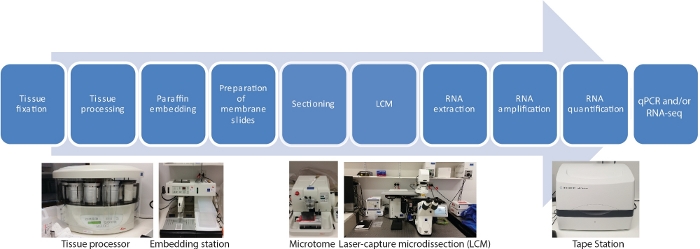
그림 1: LCM의 순서도는 RNA-seq 또는 qRT-PCR이 그 뒤를 따릅니다. LCM은 현미경 시각화 하에서 레이저 빔을 사용하여 고정 조직 섹션에서 세포를 수집하는 공간적으로 정확하고 접촉이 없는 기술입니다. 이 과정은 조직 샘플의 고정으로 시작하여 에탄올과 자일렌의 그라데이션 시리즈를 사용하여 탈수로 시작하여 파라핀 침투로 완성됩니다. 이 과정은 티슈 프로세서를 사용하여 완전히 자동화될 수 있습니다. 조직이 파라핀으로 침투하면 포함 스테이션을 사용하여 용융 파라핀이있는 금형에 내장됩니다. 절제는 원하는 두께로 설정된 마이크로토메를 사용하여 수행됩니다. 슬라이드는 RNA가 포획된 세포에서 추출되기 직전에 제조되고 LCM이 수행됩니다. RNA 추출은 qRT-PCR 및/또는 RNA-seq 이전에 RNA 증폭의 2라운드에 의해 직접 뒤따릅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
많은 조직 별 유전자 발현 연구는 시간이 많이 소요되는, 노동 집약적인 시료의 손 해부에 의해 제한되었으며, 오염위험이 높으며 인간 수술이 수확하기에 충분히 손재주적이라는 시료만 활용할 수 있습니다. LCM은 현미경 시각화 하에서 기계적으로 작동하는 레이저 빔을 사용하여 고정 조직 섹션에서 세포를 수집하는 정확하고 접촉없는 기술입니다.
좋은 샘플 준비는 LCM에 ?…
Declarações
The authors have nothing to disclose.
Acknowledgements
이 작품은 식물 에너지 생물학 (CE140100008)에서 JW에 우수의 호주 연구 위원회 센터에 의해 지원되었다. M.G.L은 라 트로브 대학 시작 보조금에 의해 지원되었다. 우리는 높은 처리량 시퀀싱 및 데이터 분석에 대한 지원에 대한 La Trobe 유전체학 플랫폼에 감사드립니다. 우리는 우리의 실험실에서 LCM을 설립에 대한 전문가의 조언을 준 교수 매튜 터커 에게 감사드립니다.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Referências
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

