Secuenciación de ARN-Secuenciación de ARN-Captura láser para Análisis de Expresión Genética Espacial y Temporal Específica de Tejidos en Plantas
Summary
Aquí se presenta un protocolo para la microdisección de captura láser (LCM) de tejidos vegetales. LCM es una técnica microscópica para aislar áreas de tejido de una manera libre de contaminación. El procedimiento incluye fijación de tejido, incrustación de parafina, seccionamiento, LCM y extracción de ARN. El ARN se utiliza en el análisis de transcriptomas de transcriptomas específicos del tejido aguas abajo y resueltos temporalmente.
Abstract
El desarrollo de un organismo multicelular complejo se rige por tipos celulares distintos que tienen diferentes perfiles transcripcionales. Para identificar las redes reguladoras transcripcionales que rigen los procesos de desarrollo es necesario medir los perfiles de expresión génica espacial y temporal de estos tipos de células individuales. Por lo tanto, la comprensión del control espacio-temporal de la expresión génica es esencial para comprender cómo se regulan los procesos biológicos y de desarrollo. Aquí, describimos un método de microdisección de captura láser (LCM) para aislar un pequeño número de células de tres órganos embrionarios de cebada durante un curso de tiempo durante la germinación seguido de perfiles de transcripción. El método consiste en fijación de tejidos, procesamiento de tejidos, incrustación de parafina, seccionamiento, LCM y extracción de ARN seguido de PCR en tiempo real o ARN-seq. Este método nos ha permitido obtener perfiles espaciales y temporales de transcriptomas de órganos de semillas de diferentes números de células (decenas a cientos), proporcionando una especificidad tisular mucho mayor que los análisis típicos de tejido a granel. A partir de estos datos pudimos definir y comparar redes reguladoras transcripcionales, así como predecir los factores de transcripción regulatorias candidatos para tejidos individuales. El método debe ser aplicable a otros tejidos vegetales con una optimización mínima.
Introduction
El desarrollo y crecimiento de las plantas implican la acción coordinada de las redes reguladoras transcripcionales dentro de diferentes células que existen en un entorno celular complejo. Para entender la actividad de estas redes reguladoras, requerimos el conocimiento de la expresión génica espacial y temporal dentro de diferentes tipos de células en todas las etapas del desarrollo. Sin embargo, los análisis de la expresión génica se llevan a cabo más comúnmente en órganos enteros o muestras de tejido a granel debido al desafío técnico de aislar y analizar un pequeño número de células. El método que describimos aquí ha permitido obtener análisis de transcriptomas espaciales y temporales específicos del tejido mediante el acoplamiento de LCM con RNA-seq.
LCM fue desarrollado hace dos décadas por Emmert-Buck y colegas1. La técnica permitió a los investigadores aislar con precisión células individuales o grupos de células de su entorno utilizando la visualización microscópica directa y la manipulación con un láser de haz estrecho1. Desde entonces el método ha sido ampliamente utilizado en la biología del cáncer y la patología2,3. Muchos grupos de investigación vegetal también han adaptado LCM para el uso con diferentes especies de plantas y diferentes tipos de tejidos4,,5,6,7,8,9,10,11. Recientemente, varios trabajos también han utilizado LCM sobre semillas eudicot y monocot para estudiar embriones, endospermas y otras estructuras de semillas durante el desarrollo de semillas y la germinación10,,12,,13. La mayoría de los otros métodos de aislamiento de una sola célula comúnmente utilizados, como micro-pipetting, clasificación celular, separación magnética y plataformas microfluídicas dependen de la digestión enzimática o homogeneización mecánica para disociar las células. Esto puede perturbar la expresión génica, introduciendo artefactos técnicos que confunda la interpretación de los datos14,15. Estos métodos también requieren el conocimiento previo de los genes marcadores para cada tipo de célula para relacionar las células disociadas con su ubicación espacial y el tipo de célula verdadero. Otro grupo de técnicas depende del aislamiento basado en afinidad de estructuras subcelulares en lugar de células enteras, por ejemplo INTACT (Aislamiento de núcleos etiquetados en tipos de celdas) y TRAP (Traducción de la purificación de afinidad ribosoma)16,17. Sin embargo, el etiquetado y la purificación de afinidad de núcleos o ribosomas son técnicamente desafiantes en especies vegetales que no tienen protocolos de transformación bien establecidos. LCM aprovecha la fijación rápida del tejido para preservar los niveles de transcripción y la identificación histológica convencional mediante la visualización directa de las células dentro de su contexto normal de tejido/órgano, lo que permite aislar las células discretas en un corto período de tiempo18,,19.
El protocolo presentado aquí es un método optimizado para el aislamiento de células o tipos celulares específicos de las secciones tisulares de las semillas de cereales, que se puede aplicar a la mayoría de las células que pueden ser identificadas histológicamente. LCM proporciona un método sin contacto de aislamiento celular, reduciendo en gran medida la contaminación y aumentando la integridad del ARN recuperado. Además, el método ilustra el poder de la MTC en estudios genómicos a gran escala a partir de pequeñas cantidades de materiales biológicos. También describimos la amplificación lineal del ARN para generar suficiente material de entrada para análisis de transcripción/transcriptoma posterior.
Hay diez pasos principales en este protocolo LCM RNA-seq para transcriptomas espaciales y temporales específicos de los tejidos, incluyendo la fijación de muestras de tejido, deshidratación, infiltración de parafina, incrustación, seccionamiento, LCM, extracción de ARN, amplificación de ARN, cuantificación de ARN y qRT-PCR y/o ARN-seq (Figura 1).
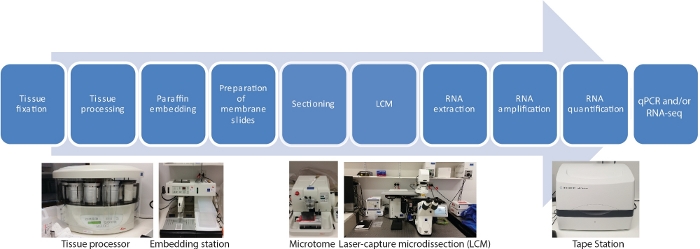
Figura 1: Diagrama de flujo de LCM seguido de RNA-seq o qRT-PCR. LCM es una técnica espacialmente precisa y libre de contacto para recoger células de secciones de tejido fijo utilizando un rayo láser bajo visualización microscópica. El proceso comienza con la fijación de muestras de tejido, seguido de deshidratación utilizando una serie de gradientes de etanol y xileno, y terminado con infiltración de parafina. El proceso se puede automatizar completamente mediante el uso de un procesador de tejidos. Una vez que el tejido se infiltra con parafina, se incrusta en un molde con parafina fundida utilizando una estación de incrustación. El seccionamiento se lleva a cabo utilizando microtomos ajustados al espesor deseado. Se preparan las diapositivas y se realiza LCM inmediatamente antes de que el ARN se extraiga de las células capturadas. La extracción de ARN es seguida directamente por dos rondas de amplificación de ARN antes de qRT-PCR y/o ARN-seq. Haga clic aquí para ver una versión más grande de esta figura.
Protocol
Representative Results
Discussion
Muchos estudios de expresión génica específicos de los tejidos se han visto limitados por la disección manual de muestras, que consume mucho tiempo, requiere mucho trabajo, tiene un alto riesgo de contaminación y sólo puede utilizar muestras de que un agente humano es lo suficientemente hábil para cosechar. LCM es una técnica precisa y sin contacto para recoger células de secciones de tejido fijo utilizando un rayo láser operado mecánicamente bajo visualización microscópica.
Una b…
Declarações
The authors have nothing to disclose.
Acknowledgements
Este trabajo fue apoyado por el Centro de Excelencia en Biología de la Energía Vegetal del Consejo Australiano de Investigación (CE140100008) a JW. M.G.L fue apoyado por una beca inicial de la Universidad La Trobe. Agradecemos a La Trobe Genomics Platform por su apoyo en la secuenciación de alto rendimiento y análisis de datos. Agradecemos al Profesor Asociado Matthew Tucker por el asesoramiento experto sobre el establecimiento de LCM en nuestro laboratorio.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Referências
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

