Bitkilerde Mekansal ve Zamansal Dokuya Özgü Gen Ekspresyonu Analizi için Lazer Yakalama Mikrodiszesi RNA-Dizileme
Summary
Burada sunulan bitki dokularının lazer yakalama mikrodisseksi (LCM) için bir protokoldür. LCM, doku alanlarını kontaminasyonsuz bir şekilde izole etmek için mikroskobik bir tekniktir. Prosedür doku fiksasyonu, parafin katıştırma, kesit, LCM ve RNA ekstraksiyon içerir. RNA, transkripsiyonların alt dokuya özgü, zamansal olarak çözümlenmiş analizinde kullanılır.
Abstract
Karmaşık çok hücreli bir organizmanın gelişimi farklı transkripsiyonel profillere sahip farklı hücre tipleri tarafından yönetilir. Gelişimsel süreçleri yöneten transkripsiyonel düzenleyici ağları belirlemek için bu bireysel hücre türlerinin mekansal ve zamansal gen ekspresyonu profillerini ölçmek gerekir. Bu nedenle, biyolojik ve gelişimsel süreçlerin nasıl düzenlendiğini anlamak için gen ekspresyonunun mekano-zamansal kontrolüne ilişkin içgörü esastır. Burada, çimlenme sırasında üç arpa embriyo organlarından az sayıda hücreyi izole etmek ve ardından transkript profilleme yöntemini lazer yakalama mikrodizesi (LCM) yöntemini tanımlıyoruz. Yöntem doku fiksasyonu, doku işleme, parafin katıştırma, kesit, LCM ve RNA ekstraksiyongerçek zamanlı PCR veya RNA-seq takip oluşur. Bu yöntem, tohum organ transkripsiyonlarının mekansal ve zamansal profillerini farklı sayıda hücreden (onlarca ila yüzlerce) elde etmemizi sağlayarak tipik toplu doku analizlerinden çok daha fazla doku özgüllüğü sağlamıştır. Bu verilerden transkripsiyonel düzenleyici ağları tanımlayabildik ve karşılaştırabildik ve bireysel dokular için aday düzenleyici transkripsiyon faktörlerini tahmin edebildik. Yöntem en az optimizasyon ile diğer bitki dokuları için geçerli olmalıdır.
Introduction
Bitki geliştirme ve büyüme karmaşık bir hücresel ortamda var olan farklı hücreler içinde transkripsiyonel düzenleyici ağların koordine eylem içerir. Bu düzenleyici ağların etkinliğini anlamak için, gelişim aşamalarında farklı hücre tipleri içinde mekansal ve zamansal gen ekspresyonu bilgisine ihtiyaç duyarız. Ancak, gen ekspresyonunun analizleri daha yaygın olarak tüm organlarda veya toplu doku örneklerinde, az sayıda hücrenin izole edilip analiz edilebis teknik zorluğu nedeniyle yapılır. Burada tanımladığımız yöntem, LCM’yi RNA-seq ile kaplayarak mekansal ve temporal dokuya özgü transkripsiyon analizinin elde edilmesine olanak sağlamıştır.
LCM emmert-Buck ve meslektaşları1tarafından yirmi yıl önce geliştirilmiştir. Bu teknik, araştırmacıların dar bir ışınlazeri1 ile doğrudan mikroskobik görüntüleme ve manipülasyon kullanarak tek hücreli veya hücre kümelerini çevrelerinden tam olarak izole etmelerini sağladı. O zamandan beri yöntem yaygın kanser biyolojisi ve patoloji2,3kullanılmaktadır . Birçok bitki araştırma grupları da farklı bitki türleri ve farklı doku türleri4,5,6,7,8,,9,10,11ile kullanım için LCM adapte var. Son zamanlarda, çeşitli kağıtları da embriyo, endosperm ve tohum gelişimi ve çimlenme sırasında diğer tohum yapıları incelemek için eudicot ve monocot tohumları üzerinde LCM kullandık10,12,13. Mikro-pipetleme, hücre ayrıştırma, manyetik ayırma ve mikroakışkan platformlar gibi yaygın olarak kullanılan diğer tek hücreli izolasyon yöntemlerinin çoğu hücreleri ayırmak için enzimatik sindirim veya mekanik homojenizasyon bağlıdır. Bu perturb gen ekspresyonu olabilir, bu veri yorumu 14 ,,15şaşırtan teknik eserler tanıtan .14 Bu yöntemler aynı zamanda her hücre tipi için işaretgenleri önceki bilgi gerektirir onların mekansal konumu ve gerçek hücre tipi ile ayrıştırılmış hücreleri ilişkilendirmek için. Tekniklerin bir başka grup tüm hücreler yerine hücre altı yapıların yakınlık tabanlı izolasyon bağlıdır, örneğin INTACT (Hücre Tiplerinde Etiketli Çekirdeklerin İzolasyon) ve TRAP (Ribozom Affinity Arınma Çevirme)16,17. Ancak, iyi kurulmuş dönüşüm protokolleri olmayan bitki türlerinde, çekirdek lerin veya ribozomların yakınlık etiketlemesi ve arınması teknik olarak zordur. LCM transkript düzeylerini korumak için hızlı doku fiksasyonu ve normal doku / organ bağlamında hücrelerin doğrudan görselleştirme ile konvansiyonel histolojik kimlik yararlanır, hangi kısa bir süre içinde izole edilmesine olanak sağlar18,19.
Burada sunulan protokol, histolojik olarak tanımlanabilen hücrelerin çoğuna uygulanabilen tahıl tohumlarının doku bölümlerinden belirli hücrelerin veya hücre tiplerinin izole edilmesi için optimize edilmiş bir yöntemdir. LCM, kontaminasyonu büyük ölçüde azaltarak ve kurtarılan RNA’nın bütünlüğünü artırarak temassız bir hücre yalıtımı yöntemi sağlar. Ayrıca, yöntem biyolojik malzemelerin küçük miktarlarda başlayan büyük ölçekli genom geniş çalışmalarda LCM gücünü göstermektedir. Ayrıca, downstream transkript/transkripsiyon analizleri için yeterli giriş materyali oluşturmak için RNA’nın doğrusal amplifikasyonuna neden olduğunu da açıklıyoruz.
Doku örneklerinin fiksasyonu, dehidratasyon, parafin infiltrasyonu, katıştırma, kesitleme, LCM, RNA ekstraksiyonu, RNA amplifikasyonu, RNA nicelleştirme ve qRT-PCR ve/veya RNA-seq dahil olmak üzere bu LCM RNA-seq protokolünde on ana adım vardır (Şekil 1).
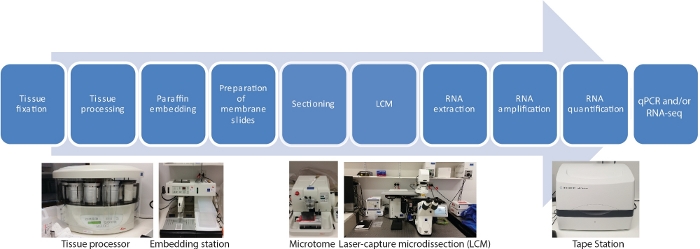
Şekil 1: LCM akış şeması ve ardından RNA-seq veya qRT-PCR. LCM mikroskobik görselleştirme altında bir lazer ışını kullanarak sabit doku bölümlerinden hücreleri toplamak için bir mekansal hassas ve temassız bir tekniktir. Süreç doku örneklerinin fiksasyonu ile başlar, etanol ve ksilen gradyan bir dizi kullanarak dehidratasyon takip, ve parafin infiltrasyon ile bitmiş. İşlem, bir doku işlemcisi kullanılarak tam otomatikhale alınabilir. Doku parafin ile infiltrasyon sonra, bir katışma istasyonu kullanarak erimiş parafin ile bir kalıp gömülüdür. Kesit istenilen kalınlıkta mikrotom seti kullanılarak yapılır. Slaytlar hazırlanır ve RNA yakalanan hücrelerden ayıklanacak hemen önce LCM yapılır. RNA ekstraksiyonu, qRT-PCR ve/veya RNA-seq’dan önce iki tur RNA amplifikasyonu ile doğrudan takip edilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Dokuya özgü gen ekspresyonu çalışmaları, zaman alan, emek yoğun olan örneklerin el diseksiyonu ile sınırlandırılmıştır, bu da yüksek kontaminasyon riskine sahiptir ve sadece bir insan operatifinin hasat için yeterince becerikli olduğu örneklerinden yararlanabilmiştir. LCM mikroskobik görselleştirme altında mekanik olarak işletilen lazer ışını kullanarak sabit doku bölümlerinden hücreleri toplamak için hassas ve temassız bir tekniktir.
İyi numune hazırlama LCM…
Declarações
The authors have nothing to disclose.
Acknowledgements
Bu çalışma Avustralya Araştırma Konseyi Bitki Enerji Biyolojisi Mükemmellik Merkezi (CE140100008) tarafından JW’ye desteklenmiştir. M.G.L, La Trobe Üniversitesi’nin başlangıç bursu ile desteklendi. La Trobe Genomik Platformu’na yüksek iş bilgisi sıralaması ve veri analizine verdikleri destekiçin teşekkür ederiz. Laboratuvarımızda LCM kurulması konusunda uzman tavsiyesi için Doçent Matthew Tucker’a teşekkür ederiz.
Materials
| Acetic acid 100 % ACS/R. | AnalaR NORMAPUR (BioStrategies) | VWRC20104.323 | |
| AdhesiveCap 200 opaque | Zeiss | 415190-9181-000 | |
| Clear base moulds 8 X 10 | Leica | 3803015 | |
| Diethyl pyrocarbonate | Sigma-Aldrich | 40718-25ML | |
| High Sensitivity RNA ScreenTape | Agilent | 5067-5579 | |
| Lowprofile disp.blades DB80LS | Leica | 14035843489 | |
| MembraneSlide 1.0 PEN | Zeiss | 415190-9041-000 | |
| MessageAmp II aRNA Amplification Kit | Ambion (ThermoFisher) | AMB17515 | |
| On-Column DNase I Digestion Set | Sigma-Aldrich | DNASE70 | |
| Ovation RNA-Seq System V2 | NuGen (Integrated Science) | 7102-08 | |
| Paraffin (Surgipath Paraplast) | Leica | 39601006 | |
| PicoPure RNA Isolation Kit | ABI (ThermoFisher) | KIT0214 | |
| RNaseZap RNase Decontamination Solution | Ambion (ThermoFisher) | AM9780 | |
| Xylene | AnalaR NORMAPUR (BioStrategies) | VWRC28975.360 | |
| Leica Benchtop Tissue Processor | Leica Biosystems | TP1020 | |
| Leica Heated Paraffin Embedding Module | Leica Biosystems | EG1150H | |
| Leica Cold Plate | Leica Biosystems | EG1150C | |
| Safemate Class 2 Biological Safety Cabinets | LAF Technologies Pty Ltd | Safemate 1.5 | |
| Leica Fully Automated Rotary Microtome | Leica Biosystems | RM2265 | with PALMRobo v 4.6 software |
| Zeiss PALM MicroBeam LCM system | Zeiss miscroscopy | ||
| TapeStation | Agilent | TapeStation 2200 |
Referências
- Emmert-Buck, M. R., et al. Laser capture microdissection. Science. 274 (5289), 998-1001 (1996).
- Alevizos, I., et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene. 20 (43), 6196-6204 (2001).
- Cong, P., et al. In situ localization of follicular lymphoma: description and analysis by laser capture microdissection. Blood, The Journal of the American Society of Hematology. 99 (9), 3376-3382 (2002).
- Blokhina, O., et al. Laser capture microdissection protocol for xylem tissues of woody plants. Frontiers in Plant Science. 7, 1965 (2017).
- Casson, S., Spencer, M., Walker, K., Lindsey, K. Laser capture microdissection for the analysis of gene expression during embryogenesis of Arabidopsis. The Plant Journal. 42 (1), 111-123 (2005).
- Chen, Z., et al. LCM-seq reveals the crucial role of LsSOC1 in heat-promoted bolting of lettuce (Lactuca sativa L.). The Plant Journal. 95 (3), 516-528 (2018).
- Jiao, Y., et al. A transcriptome atlas of rice cell types uncovers cellular, functional and developmental hierarchies. Nature Genetics. 41 (2), 258-263 (2009).
- Kivivirta, K., et al. A protocol for laser microdissection (LMD) followed by transcriptome analysis of plant reproductive tissue in phylogenetically distant angiosperms. Plant Methods. 15 (1), 1-11 (2019).
- Li, P., et al. The developmental dynamics of the maize leaf transcriptome. Nature Genetics. 42 (12), 1060-1067 (2010).
- Liew, L. C., et al. Temporal tissue-specific regulation of transcriptomes during barley (Hordeum vulgare) seed germination. The Plant Journal. 101 (3), 700-715 (2020).
- Matas, A. J., et al. Tissue-and cell-type specific transcriptome profiling of expanding tomato fruit provides insights into metabolic and regulatory specialization and cuticle formation. The Plant Cell. 23 (11), 3893-3910 (2011).
- Sakai, K., et al. Combining laser-assisted microdissection (LAM) and RNA-seq allows to perform a comprehensive transcriptomic analysis of epidermal cells of Arabidopsis embryo. Plant Methods. 14 (1), 10 (2018).
- Zhan, J., et al. RNA Sequencing of Laser-Capture Microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation. The Plant Cell. 27 (3), 513-531 (2015).
- Hwang, B., Lee, J. H., Bang, D. Single-cell RNA sequencing technologies and bioinformatics pipelines. Experimental & Molecular Medicine. 50 (8), 1-14 (2018).
- Zeb, Q., Wang, C., Shafiq, S., Liu, L. . Single-Cell Omics. , 101-135 (2019).
- Deal, R. B., Henikoff, S. The INTACT method for cell type-specific gene expression and chromatin profiling in Arabidopsis thaliana. Nature Protocols. 6 (1), 56 (2011).
- Heiman, M., Kulicke, R., Fenster, R. J., Greengard, P., Heintz, N. Cell type-specific mRNA purification by translating ribosome affinity purification (TRAP). Nature Protocols. 9 (6), 1282 (2014).
- Bevilacqua, C., Ducos, B. Laser microdissection: A powerful tool for genomics at cell level. Molecular Aspects of Medicine. 59, 5-27 (2018).
- Nelson, T., Tausta, S. L., Gandotra, N., Liu, T. Laser microdissection of plant tissue: what you see is what you get. Annual Reviews in Plant Biology. 57, 181-201 (2006).
- Day, R. C., Grossniklaus, U., Macknight, R. C. Be more specific! Laser-assisted microdissection of plant cells. Trends in Plant Science. 10 (8), 397-406 (2005).
- Takahashi, H., et al. A method for obtaining high quality RNA from paraffin sections of plant tissues by laser microdissection. Journal of Plant Research. 123 (6), 807-813 (2010).
- Schroeder, A., et al. The RIN: an RNA integrity number for assigning integrity values to RNA measurements. BMC Molecular Biology. 7 (1), 3 (2006).
- Ferreira, E. N., et al. Linear mRNA amplification approach for RNAseq from limited amount of RNA. Gene. 564 (2), 220-227 (2015).
- Schneider, J., et al. Systematic analysis of T7 RNA polymerase based in vitro linear RNA amplification for use in microarray experiments. BMC Genomics. 5 (1), 29 (2004).
- Shanker, S., et al. Evaluation of commercially available RNA amplification kits for RNA sequencing using very low input amounts of total RNA. Journal of Biomolecular Techniques. 26 (1), 4 (2015).
- Bhattacherjee, V., et al. Laser capture microdissection of fluorescently labeled embryonic cranial neural crest cells. Genesis. 39 (1), 58-64 (2004).
- Clément-Ziza, M., Munnich, A., Lyonnet, S., Jaubert, F., Besmond, C. Stabilization of RNA during laser capture microdissection by performing experiments under argon atmosphere or using ethanol as a solvent in staining solutions. RNA. 14 (12), 2698-2704 (2008).
- Blokhina, O., et al. Parenchymal Cells Contribute to Lignification of Tracheids in Developing Xylem of Norway Spruce. Plant Physiology. 181 (4), 1552-1572 (2019).
- Schad, M., Lipton, M. S., Giavalisco, P., Smith, R. D., Kehr, J. Evaluation of two-dimensional electrophoresis and liquid chromatography-tandem mass spectrometry for tissue-specific protein profiling of laser-microdissected plant samples. Electrophoresis. 26 (14), 2729-2738 (2005).
- Schad, M., Mungur, R., Fiehn, O., Kehr, J. Metabolic profiling of laser microdissected vascular bundles of Arabidopsis thaliana. Plant Methods. 1 (1), 2 (2005).
- Latrasse, D., et al. The quest for epigenetic regulation underlying unisexual flower development in Cucumis melo. Epigenetics & Chromatin. 10 (1), 22 (2017).
- Turco, G. M., et al. DNA methylation and gene expression regulation associated with vascularization in Sorghum bicolor. The New Phytologist. 214 (3), 1213-1229 (2017).
- Gomez, S. K., Harrison, M. J. Laser microdissection and its application to analyze gene expression in arbuscular mycorrhizal symbiosis. Pest Management Science: Formerly Pesticide Science. 65 (5), 504-511 (2009).
- Roux, B., et al. An integrated analysis of plant and bacterial gene expression in symbiotic root nodules using laser-capture microdissection coupled to RNA sequencing. The Plant Journal. 77 (6), 817-837 (2014).
- Tang, W., Coughlan, S., Crane, E., Beatty, M., Duvick, J. The application of laser microdissection to in planta gene expression profiling of the maize anthracnose stalk rot fungus Colletotrichum graminicola. Molecular Plant-Microbe Interactions. 19 (11), 1240-1250 (2006).

