Live imaging met hoge doorvoer van microkolonnies om heterogeniteit in groei en genexpressie te meten
Summary
Gistgroei fenotypes worden nauwkeurig gemeten door middel van zeer parallelle time-lapse beeldvorming van geïmmobiliseerde cellen die uitgroeien tot microkolonnies. Tegelijkertijd kunnen stresstolerantie, eiwitexpressie en eiwitlokalisatie worden gemonitord, waardoor geïntegreerde datasets worden gegenereerd om te bestuderen hoe omgevings- en genetische verschillen, evenals genexpressie heterogeniteit tussen isogene cellen, de groei moduleren.
Abstract
Nauwkeurige metingen van heterogeniteit tussen en binnen de stam in microbiële groeisnelheden zijn essentieel voor het begrijpen van genetische en omgevingsinputs in stresstolerantie, pathogeniteit en andere belangrijke componenten van fitness. Dit manuscript beschrijft een microscoop-gebaseerde test die ongeveer 105 Saccharomyces cerevisiae microkolonnies per experiment volgt. Na geautomatiseerde time-lapse beeldvorming van gist geïmmobiliseerd in een multiwell plaat, microkolonisatie groeisnelheden kunnen eenvoudig worden geanalyseerd met aangepaste beeldanalyse software. Voor elke microkolonie kunnen ook expressie en lokalisatie van fluorescerende eiwitten en overleving van acute stress worden gecontroleerd. Deze test maakt een nauwkeurige schatting van de gemiddelde groeipercentages van stammen mogelijk, evenals een uitgebreide meting van heterogeniteit in groei, genexpressie en stresstolerantie binnen klonale populaties.
Introduction
Groeifenotypes dragen kritisch bij aan de gistconditie. Natuurlijke selectie kan efficiënt onderscheid maken tussen afstammingen met groeipercentages die verschillen door de inverse van de effectieve populatiegrootte, die meer dan 108 individuen kan overschrijden1. Bovendien is variabiliteit van groeipercentages tussen individuen binnen een populatie een evolutionair relevante parameter , aangezien deze kan dienen als basis voor overlevingsstrategieën zoals bet hedging2,3,4,5,6. Daarom zijn assays die zeer nauwkeurige metingen van groeifenotypes en hun distributies mogelijk maken, cruciaal voor de studie van micro-organismen. De hier beschreven microkoloniale groeitest kan individuele groeisnelheidsmetingen genereren voor ~ 105 microkolonnies per experiment. Deze test biedt daarom een krachtig protocol om gist evolutionaire genetica en genomica te bestuderen. Het leent zich bijzonder goed om te testen hoe variabiliteit binnen populaties van genetisch identieke enkelvoudige cellen wordt gegenereerd, gehandhaafd en bijdraagt tot populatiegeschiktheid7,8,9,10.
De hier beschreven methode (figuur 1) maakt gebruik van periodiek vastgelegde, low-vergroting brightfield beelden van cellen die groeien in vloeibare media op een 96- of 384-put glasbodemplaat om de groei tot microkolonnies te volgen. De cellen hechten zich aan de lectineconcanavalin A, die de bodem van de microscoopplaat bedekt en tweedimensionale kolonies vormt. Omdat de microkolonnies in een monolaag groeien, is het microkoloniale gebied sterk gecorreleerd met celnummer7. Daarom kunnen nauwkeurige schattingen van de groeisnelheid en vertragingstijd van microkolonen worden gegenereerd met aangepaste beeldanalysesoftware die de veranderingssnelheid van het gebied van elke microkolonisatie bijhoudt. Bovendien kan de experimentele opstelling de overvloed en zelfs de subcellulaire lokalisaties van fluorescerend gelabelde eiwitten, uitgedrukt in deze microkolonnies, monitoren. Downstream verwerking van gegevens van deze microkoloniale groeitest kan worden bereikt door aangepaste analyse of door bestaande beeldanalysesoftware, zoals Processing Images Easily (PIE)11, een algoritme voor robuuste koloniegebiedherkenning en groeianalyse met hoge doorvoer van low-magnification, brightfield-afbeeldingen, die beschikbaar is via GitHub12.
Omdat groeisnelheidsschattingen afgeleid van de microkoloniale-groeitest worden gegenereerd uit een groot aantal metingen met één kolonie, zijn ze uiterst nauwkeurig, met standaardfouten die enkele ordes van grootte kleiner zijn dan de schattingen zelf voor een experiment van redelijke omvang. Daarom is de kracht van de test om groeisnelheidsverschillen tussen verschillende genotypen, behandelingen of omgevingsomstandigheden te detecteren hoog. Het multiwell-plaatformaat maakt het mogelijk om tal van verschillende omgevings- en genotypecombinaties in één experiment te vergelijken. Als stammen constitutief verschillende fluorescerende markers uitdrukken, kunnen ze in dezelfde put worden gemengd en worden onderscheiden door daaropvolgende beeldanalyse, wat het vermogen verder zou kunnen vergroten door een goede gegevensnormalisatie mogelijk te maken.
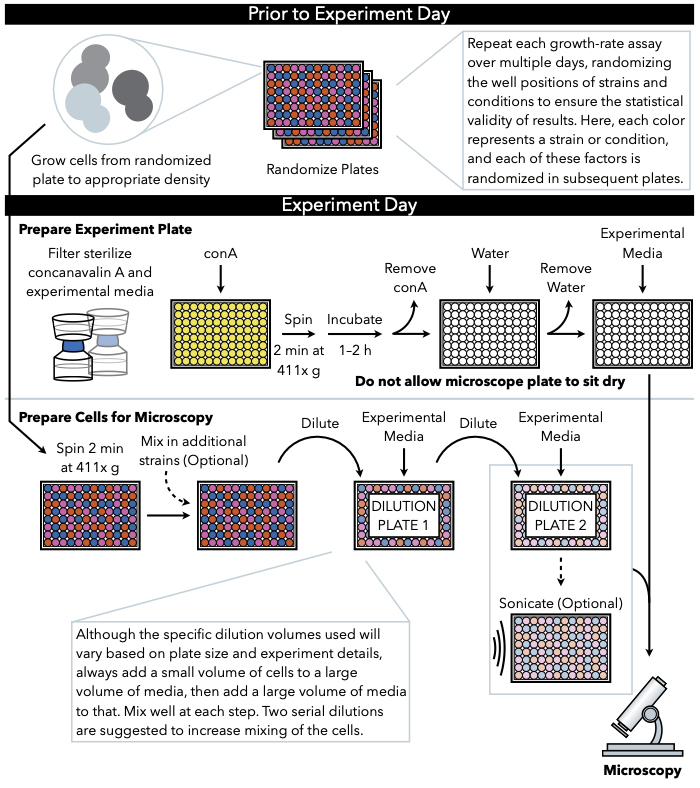
Figuur 1: Schematische weergave van het protocol. Dit protocol volgt twee belangrijke stappen, namelijk de voorbereiding van de experimentele plaat en de voorbereiding van de cellen op beeld. Randomisatie van platen en groei van cellen moeten worden uitgevoerd vóór en voorafgaand aan de experimentdag. Herhaalde menging van cellen bij elke stap tijdens verdunning is noodzakelijk in de stappen tot het plateren, en daarom wordt het voorbereiden van de experimentele plaat eerst aanbevolen, zodat deze onmiddellijk na voltooiing van de celverdunning klaar is voor plating. Klik hier om een grotere versie van deze afbeelding te bekijken.
Protocol
Representative Results
Discussion
Het hier beschreven protocol is een veelzijdige test waarmee celgroei en genexpressie tegelijkertijd kunnen worden gecontroleerd op het niveau van individuele microkolonnies. Het combineren van deze twee modaliteiten levert unieke biologische inzichten op. Eerder werk heeft deze test bijvoorbeeld gebruikt om een negatieve correlatie aan te tonen tussen de expressie van het TSL1-gen en de microkoloniale groeisnelheid in isogene wildtypecellen door beide gelijktijdig7,<sup class=…
Declarações
The authors have nothing to disclose.
Acknowledgements
We danken Naomi Ziv, Sasha Levy en Shuang Li voor hun bijdragen aan de ontwikkeling van dit protocol, David Gresham voor gedeelde apparatuur en Marissa Knoll voor hulp bij videoproductie. Dit werk werd ondersteund door de National Institutes of Health grant R35GM118170.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Referências
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genética. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genética. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genética. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

