성장과 유전자 발현의 이질성을 측정하는 마이크로콜로니의 높은 처리량 라이브 이미징
Summary
효모 성장 표현형은 마이크로콜로 성장하는 고정 된 세포의 매우 병렬 시간 경과 이미징을 통해 정확하게 측정됩니다. 동시에, 스트레스 내성, 단백질 발현 및 단백질 국소화를 모니터링할 수 있고, 통합된 데이터 세트를 생성하여 환경 및 유전적 차이뿐만 아니라 동종 세포 간의 유전자 발현 이질성, 변조 성장을 연구할 수 있습니다.
Abstract
미생물 성장 속도에서 스트레인 내이질성의 정확한 측정은 유전적 및 환경 입력을 스트레스 내성, 병원성 및 기타 피트니스 구성 요소로 이해하는 데 필수적입니다. 이 원고는 실험 당 약 105 Saccharomyces cerevisiae 미세 식민지를 추적하는 현미경 기반 분석서를 설명합니다. 멀티웰 플레이트에 고정된 효모의 자동 타임랩스 이미징 후 맞춤형 이미지 분석 소프트웨어로 미세 콜로니 성장률을 쉽게 분석할 수 있습니다. 각 미세 식민지에 대 한, 발현 및 형광 단백질의 국소화 및 급성 스트레스의 생존또한 모니터링 될 수 있습니다. 이 분석법은 균주의 평균 성장률의 정확한 추정뿐만 아니라 클로나 집단 내에서 성장, 유전자 발현 및 스트레스 내성의 이질성의 포괄적 인 측정을 허용합니다.
Introduction
성장 표현형은 효모 피트니스에 매우 기여합니다. 자연 선택은 108명을 초과할 수 있는 유효 인구 규모의 역에 따라 성장률이 다른 혈통을 효율적으로 구별할 수있다. 더욱이, 인구 내개인의 성장률의 변동성은 2,3,4,5,6과같은 생존 전략의 기초역할을 할 수 있기 때문에 진화적으로 관련된 매개 변수이다. 따라서, 성장 표현형및 그 분포의 고도로 정확한 측정을 허용하는 아스약은 미생물의 연구에 중추적인 이다. 여기에 설명된 미세 식민지 성장 분석은 실험 당 ~105 마이크로 콜로니에 대한 개별 성장 속도 측정을 생성 할 수 있습니다. 이 분석법은 따라서 효모 진화 유전학 및 유전체학을 연구하기 위하여 강력한 프로토콜을 제공합니다. 그것은 유전적으로 동일한 단일 세포의 인구 내의 가변성이 생성되고, 유지되고, 인구 적합성7,8,9,10에기여하는 방법을 테스트하는 데 특히 잘 빌려준다.
여기서 설명된 방법은(도 1)을주기적으로 캡처한, 96-384웰 유리 바닥 플레이트에서 액체 미디어에서 성장하는 세포의 저배율 밝은 필드 이미지를 사용하여 마이크로콜로니로의 성장을 추적한다. 세포는 현미경 판의 바닥을 코팅하고 2 차원 식민지를 형성하는 렉틴 concanavalin A를 부착합니다. 마이크로콜로니가 단층에서 자라기 때문에 마이크로콜로니 영역은 세포 번호7과매우 상관관계가 있다. 따라서, 마이크로콜로니 의 성장 속도와 지연 시간의 정확한 추정은 각 마이크로 콜로니의 영역의 변화의 속도를 추적하는 사용자 정의 이미지 분석 소프트웨어로 생성 될 수있다. 더욱이, 실험용 설정은 이러한 마이크로콜로니에서 발현된 형광 표지 단백질의 풍부하고 세포전 소세포 국소화를 모니터링할 수 있다. 이러한 마이크로콜로니 성장 분석에서 데이터의 다운스트림 처리는 맞춤 분석 또는 기존 이미지 분석 소프트웨어(예: 쉽게(PIE)11,GitHub12를통해 사용할 수 있는 저배율, 브라이트필드 이미지로부터의 견고한 콜로니 영역 인식 및 고처리량 성장 분석을 위한 알고리즘으로 달성될 수 있다.
마이크로콜로니-성장 분석에서 파생된 성장률 추정치는 많은 수의 단일 콜로니 측정에서 생성되기 때문에, 표준 오류로 합리적으로 크기의 실험을 위한 추정치 자체보다 몇 배 더 작은 크기로 매우 정확합니다. 따라서 상이한 유전자형, 치료 또는 환경 조건 간의 성장률 차이를 검출하는 분석의 힘이 높다. 멀티웰 플레이트 포맷은 단일 실험에서 수많은 다른 환경과 유전자형 조합을 비교할 수 있게 해줍니다. 균주가 다른 형광 마커를 구성적으로 표현하는 경우, 동일한 우물로 혼합되고 후속 이미지 분석에 의해 구별될 수 있으며, 이는 잘 함으로써 잘 데이터 정규화를 허용함으로써 전력을 더욱 증가시킬 수 있습니다.
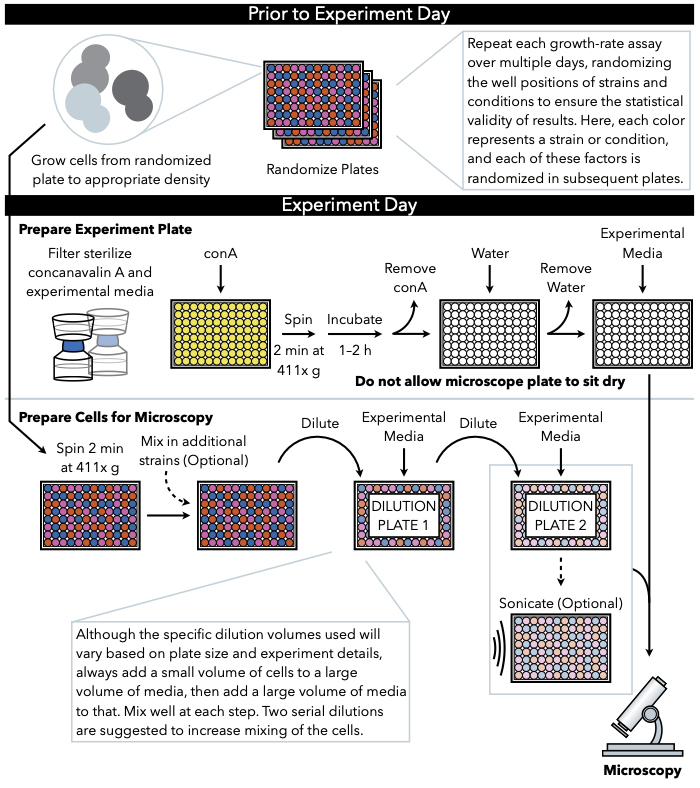
그림 1: 프로토콜의 회로도 표현. 이 프로토콜은 실험 판의 준비및 화상에 세포의 준비인 두 가지 주요 단계를 따릅니다. 플레이트의 무작위화와 세포의 성장은 실험의 날까지 전에 실시되어야한다. 희석 하는 동안 각 단계에서 세포의 반복 혼합 도금 까지 단계에서 필수적 이다, 따라서 먼저 실험 판을 준비 하는 것이 좋습니다 세포 희석의 완료 시 즉시 도금에 대 한 준비가. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
여기서 설명된 프로토콜은 세포 성장 및 유전자 발현이 개별 마이크로콜로니의 수준에서 동시에 모니터링될 수 있도록 하는 다목적 분석이다. 이 두 가지 양식이 결합하면 고유한 생물학적 통찰력을 얻을 수 있습니다. 예를 들어, 이전 연구는 이 분석을 사용하여 TSL1 유전자의 발현과 동종 야생형 세포에서 마이크로콜로니 성장률 사이의 부정적인 상관관계를 동시에7,…
Declarações
The authors have nothing to disclose.
Acknowledgements
나오미 지브, 사샤 레비, 슈앙 리(Shuang Li)가 이 프로토콜 개발에 기여한 것에 대해 감사를 표하고, 데이비드 그레샴(David Gresham)은 공유 장비를 제공했으며, 마리사 크놀이 비디오 제작에 도움을 준 것에 대해 감사드립니다. 이 작품은 건강 보조금 R35GM118170의 국가 학회에 의해 지원되었다.
Materials
| General Materials | |||
| 500 mL Bottletop Filter .22 µm PES Sterilizing, Low Protein Binding, w/45mm Neck | Fisher | CLS431154 | used to filter the media |
| BD Falcon*Tissue Culture Plates, microtest u-bottom | Fisher | 08-772-54 | 96-well culture tubes used to freeze cells, pre-grow cells, and dilutions |
| BD Syringes without Needle, 50 mL | Fisher | 13-689-8 | Used to filter the Concanavalin A |
| Costar Sterile Disposable Reagent Reservoirs | Fisher | 07-200-127 | reagent reservoirs used to pipette solutions with multichannel pipette |
| Costar Thermowell Aluminum Sealing Tape | Fisher | 07-200-684 | 96-well plate seal for pre-growth and freezing |
| lint and static free Kimwipes | Fisher | 06-666A | lint and static free wipes to keep microscope plate bottom free of debris and scratches |
| Nalgene Syringe Filters | ThermoFisher Scientific | 199-2020 | 0.2 μm pore size, 25 mm diameter; used to filter concanavalin A solution |
| Media Components | |||
| Minimal chemically defined media (MD; 2% glucose) | alternative microscopy media used for yeast pre-growth and growth during microscopy | ||
| Synthetic Complete Media (SC; 2% glucose) | microscopy media used for yeast pre-growth and growth during microscopy | ||
| Yeast extract-peptone-dextrose (YEPD; 2% glucose) medium | cell growth prior to freezing down randomized plates | ||
| Microscopy Materials | |||
| Breathe-Easy sealing membrane | Millipore Sigma | Z380059-1PAK | breathable membranes used to seal plate during microscopy experiment. At this stage breathable membranes are reccomended because they prevent condensation in the wells and allow for better microscopy images |
| Brooks 96-well flat clear glass bottom microscope plate | Dot Scientific | MGB096-1-2-LG-L | microscope plate |
| Concanavalin A from canavalia ensiformis (Jack Bean), lyophilized powder | Millipore Sigma | 45-C2010-1G | Make 5x concanavalin A solution and freeze 5ml of 5x concanavalin A in 50 mL conical tubes at -80 °C |
| Strains Used | |||
| MAH.5, MAH.96, MAH.52, MAH.66, MAH.11, MAH.58, MAH.135, MAH.15, MAH.44, MAH.132 | Haploid mutation accumulation strains in a laboratory background, described in Hall and Joseph 2010 | ||
| EP026.2A-2C | Progeny of the ancestral Hall and Joseph 2010 mutation accumulation strain, transformed with YFR054cΔ::Scw11P::GFP | ||
| Equipment | |||
| Misonix Sonicator S-4000 with 96-pin attachment | Sonicator https://www.labx.com/item/misonix-inc-s-4000-sonicator/4771281 | ||
| Nikon Eclipse Ti-E with Perfect Focus System | Inverted microscope with automated stage and autofocus system |
Referências
- Geiler-Samerotte, K. A., Hashimoto, T., Dion, M. F., Budnik, B. A., Airoldi, E. M., Drummond, D. A. Quantifying condition-dependent intracellular protein levels enables high-precision fitness estimates. PloS one. 8 (9), 75320 (2013).
- Kussell, E., Leibler, S. Phenotypic diversity, population growth, and information in fluctuating environments. Science. 309 (5743), 2075-2078 (2005).
- Thattai, M., van Oudenaarden, A. Stochastic gene expression in fluctuating environments. Genética. 167 (1), 523-530 (2004).
- King, O. D., Masel, J. The evolution of bet-hedging adaptations to rare scenarios. Theoretical population biology. 72 (4), 560-575 (2007).
- Acar, M., Mettetal, J. T., van Oudenaarden, A. Stochastic switching as a survival strategy in fluctuating environments. Nature genetics. 40 (4), 471-475 (2008).
- Avery, S. V. Microbial cell individuality and the underlying sources of heterogeneity. Nature reviews. Microbiology. 4 (8), 577-587 (2006).
- Levy, S. F., Ziv, N., Siegal, M. L. Bet hedging in yeast by heterogeneous, age-correlated expression of a stress protectant. PLoS biology. 10 (5), 1001325 (2012).
- van Dijk, D., et al. Slow-growing cells within isogenic populations have increased RNA polymerase error rates and DNA damage. Nature communications. 6, 7972 (2015).
- Ziv, N., Shuster, B. M., Siegal, M. L., Gresham, D. Resolving the Complex Genetic Basis of Phenotypic Variation and Variability of Cellular Growth. Genética. 206 (3), 1645-1657 (2017).
- Li, S., Giardina, D. M., Siegal, M. L. Control of nongenetic heterogeneity in growth rate and stress tolerance of Saccharomyces cerevisiae by cyclic AMP-regulated transcription factors. PLoS genetics. 14 (11), 1007744 (2018).
- Plavskin, Y., Li, S., Ziv, N., Levy, S. F., Siegal, M. L. Robust colony recognition for high-throughput growth analysis from suboptimal low-magnification brightfield micrographs. bioRxiv. , (2018).
- Ziv, N., Siegal, M. L., Gresham, D. Genetic and nongenetic determinants of cell growth variation assessed by high-throughput microscopy. Molecular biology and evolution. 30 (12), 2568-2578 (2013).
- Hall, D. W., Joseph, S. B. A high frequency of beneficial mutations across multiple fitness components in Saccharomyces cerevisiae. Genética. 185 (4), 1397-1409 (2010).
- Saleemuddin, M., Husain, Q. Concanavalin A: a useful ligand for glycoenzyme immobilization–a review. Enzyme and microbial technology. 13 (4), 290-295 (1991).
- Geiler-Samerotte, K. A., Bauer, C. R., Li, S., Ziv, N., Gresham, D., Siegal, M. L. The details in the distributions: why and how to study phenotypic variability. Current opinion in biotechnology. 24 (4), 752-759 (2013).
- Nakagawa, S., Schielzeth, H. Repeatability for Gaussian and non-Gaussian data: a practical guide for biologists. Biological reviews of the Cambridge Philosophical Society. 85 (4), 935-956 (2010).
- Bolker, J. A. Exemplary and surrogate models: two modes of representation in biology. Perspectives in biology and medicine. 52 (4), 485-499 (2009).

