Udforskning af biomolekylær interaktion mellem den molekylære chaperon Hsp90 og dens klientproteinkinase Cdc37 ved hjælp af felteffektbiosensingteknologi
Summary
Field-effect biosensing (FEB) er en etiketfri teknik til påvisning af biomolekylære interaktioner. Den måler den elektriske strøm gennem grafenbiosensoren, som bindingsmålene er immobiliseret til. FEB-teknologien blev brugt til at evaluere biomolekylære interaktioner mellem Hsp90 og Cdc37, og der blev påvist en stærk interaktion mellem de to proteiner.
Abstract
Biomolekylære interaktioner spiller alsidige roller i adskillige cellulære processer ved at regulere og koordinere funktionelt relevante biologiske begivenheder. Biomolekyler såsom proteiner, kulhydrater, vitaminer, fedtsyrer, nukleinsyrer og enzymer er grundlæggende byggesten i levende væsener; de samles i komplekse netværk i biosystemer for at synkronisere et utal af livsbegivenheder. Proteiner bruger typisk komplekse interaktomnetværk til at udføre deres funktioner; derfor er det obligatorisk at evaluere sådanne interaktioner for at opklare deres betydning i celler på både cellulært og organismeniveau. Mod dette mål introducerer vi en hurtigt voksende teknologi, field-effect biosensing (FEB), til at bestemme specifikke biomolekylære interaktioner. FEB er en stationær, etiketfri og pålidelig biomolekylær detektionsteknik til bestemmelse af specifikke interaktioner og bruger elektronisk baserede biosensorer af høj kvalitet. FEB-teknologien kan overvåge interaktioner i det nanomolære område på grund af de biokompatible nanomaterialer, der anvendes på dens biosensoroverflade. Som et bevis på konceptet blev protein-protein-interaktionen (PPI) mellem varmechokprotein 90 (Hsp90) og celledelingscyklus 37 (Cdc37) belyst. Hsp90 er en ATP-afhængig molekylær chaperon, der spiller en væsentlig rolle i foldning, stabilitet, modning og kvalitetskontrol af mange proteiner og derved regulerer flere vitale cellulære funktioner. Cdc37 betragtes som en proteinkinase-specifik molekylær chaperon, da den specifikt genkender og rekrutterer proteinkinaser til Hsp90 for at regulere deres nedstrøms signaltransduktionsveje. Som sådan betragtes Cdc37 som en co-chaperone af Hsp90. Chaperon-kinasevejen (Hsp90 / Cdc37-komplekset) er hyperaktiveret i flere maligniteter, der fremmer cellulær vækst; derfor er det et potentielt mål for kræftbehandling. Denne undersøgelse viser effektiviteten af FEB-teknologien ved hjælp af Hsp90 / Cdc37-modelsystemet. FEB påviste en stærk PPI mellem de to proteiner (KD-værdier på 0,014 μM, 0,053 μM og 0,072 μM i tre uafhængige eksperimenter). Sammenfattende er FEB en etiketfri og omkostningseffektiv PPI-detektionsplatform, der tilbyder hurtige og nøjagtige målinger.
Introduction
Biomolekylære interaktioner:
Proteiner er væsentlige dele af organismer og deltager i adskillige molekylære veje såsom cellemetabolisme, cellestruktur, cellesignalering, immunresponser, celleadhæsion og meget mere. Mens nogle proteiner udfører deres funktion(er) uafhængigt, interagerer de fleste proteiner med andre proteiner ved hjælp af en bindende grænseflade til at koordinere korrekt biologisk aktivitet1.
Biomolekylære interaktioner kan hovedsageligt klassificeres ud fra de forskellige strukturelle og funktionelle egenskaber ved involverede proteiner2, for eksempel baseret på proteinoverfladerne, den komplekse stabilitet eller persistensen af interaktioner3. Identifikation af essentielle proteiner og deres roller i biomolekylære interaktioner er afgørende for at forstå biokemiske mekanismer på molekylært niveau4. I øjeblikket er der forskellige tilgange til at detektere disse interaktioner5: in vitro6, in silico7, i levende celler8, ex vivo9 og in vivo10, hvor hver har sine egne styrker og svagheder.
In vivo-assays udføres ved hjælp af hele dyret som et eksperimentelt værktøj11, ogt ex vivo-assays udføres på vævsekstrakter eller hele organer (f.eks. Hjerte, hjerne, lever) i et kontrolleret eksternt miljø ved at give minimale ændringer i naturlige forhold. Den mest almindelige anvendelse af in vivo– og ex vivo-undersøgelser er at evaluere farmakokinetikken, farmakodynamikken og toksicitetsvirkningerne af potentielle farmakologiske agenser før humane forsøg ved at sikre deres samlede sikkerhed og virkning12.
Biomolekylære interaktioner kan også påvises i levende celler. Billeddannelse af levende celler giver os mulighed for at observere dynamiske interaktioner, når de udfører reaktionerne fra en bestemt biokemisk vej13. Desuden kan detektionsteknikker, såsom bioluminescens eller fluorescensresonansenergioverførsel, give information om, hvor og hvornår disse interaktioner forekommer i cellen14. Selvom detektion i levende celler tilbyder vigtige detaljer, er disse detektionsmetoder afhængige af optik og etiketter, som muligvis ikke afspejler den oprindelige biologi; de er også mindre kontrollerede end in vitro-metoder og kræver specialiseret ekspertise for at udføre15.
In silico-beregningsmetoderne anvendes primært til storstilet screening af målmolekyler før in vitro-forsøgene . Beregningsmæssige forudsigelsesmetoder, computerbaserede databaser, molekylær docking, kvantitative struktur-aktivitetsforhold og andre molekylære dynamiksimuleringsmetoder er blandt de veletablerede i silico-værktøjer 16. Sammenlignet med besværlige eksperimentelle teknikker kan in silico-værktøjerne let lave forudsigelser med høj følsomhed, men med reduceret nøjagtighed i prædiktiv ydeevne17.
In vitro-assays udføres med mikroorganismer eller biologiske molekyler uden for deres standard biologiske kontekst. At skildre biomolekylære interaktioner gennem in vitro-metoder er afgørende for at forstå proteinfunktioner og biologien bag det komplekse netværk af cellefunktion. Den foretrukne analysemetode vælges i henhold til proteinets iboende egenskaber, kinetiske værdier og interaktionsmåden og intensiteten18,19.
Hsp90/Cdc37-interaktionen:
Chaperon-kinase-vejen, der forbinder Hsp90 og Cdc37, er et lovende terapeutisk mål i tumorbiologi20. Hsp90 spiller en central rolle i cellecykluskontrol, proteinsamling, celleoverlevelse og signalveje. Proteiner, der er afhængige af Hsp90 for deres funktioner, leveres til Hsp90 til kompleksnering gennem en co-chaperon, såsom Cdc37. Hsp90/Cdc37-komplekset styrer foldningen af de fleste proteinkinaser og fungerer som et knudepunkt for en lang række intracellulære signalnetværk21. Det er et lovende antitumormål på grund af dets forhøjede ekspression i forskellige maligniteter, herunder akut myeloblastisk leukæmi, multipelt myelom og hepatocellulært carcinom22,23.
Almindeligt anvendt in vitro biomolekylære interaktionsdetekteringsteknikker
Co-immunoprecipitation (co-IP) er en teknik, der er afhængig af antigen-antistofs specificitet for at identificere biologisk relevante interaktioner24. Den primære ulempe ved denne metode er dens manglende evne til at detektere interaktioner med lav affinitet og kinetiske værdier24. Biofysiske metoder såsom isotermisk titreringskalorimetri (ITC), overfladeplasmonresonans (SPR), biolagsinterferometri (BLI) og FEB-teknologi foretrækkes til at bestemme de kinetiske værdier.
ITC er en biofysisk detektionsmetode baseret på bestemmelse af bindingsenergi sammen med en komplet termodynamikanalyse til karakterisering af biomolekylære interaktioner25. Den primære fordel ved ITC er, at det ikke kræver nogen mærkning eller fiksering af målproteinet. De største vanskeligheder, som ITC støder på, er den høje koncentration af målprotein, der kræves til et eksperiment, og vanskeligheden ved at analysere ikke-kovalente komplekser på grund af små bindende entalpier26. Både SPR og BLI er etiketfrie biofysiske teknikker, der er afhængige af immobilisering af målmolekylet på sensoroverfladen efterfulgt af efterfølgende injektioner af analytten over det immobiliserede mål27,28. I SPR måles ændringer i brydningsindekset under biomolekylære interaktioner27; i BLI registreres interferensen i reflekteret lys i realtid som en ændring i bølgelængden som en funktion af tid28. Både SPR og BLI deler fælles fordele ved at tilbyde høj specificitet, følsomhed og detektionsfunktioner29. I begge metoder immobiliseres målproteinet på biosensoroverflader, og der kan derfor være et vist tab af målets oprindelige konformation, hvilket gør det vanskeligt at skelne mellem specifikke vs. ikke-specifikke interaktioner30. BLI bruger dyre engangsfiberoptiske biosensorer til at immobilisere målet, og er derfor en dyr teknik31. Sammenlignet med disse veletablerede biomolekylære detektionsværktøjer tilbyder FEB-teknologien en pålidelig og etiketfri platform ved at bruge lave nanomolære koncentrationer til biomolekylær detektion i realtid med kinetisk karakterisering. FEB-teknologien overvinder også de boblende udfordringer, som ITC står over for, og er mere omkostningseffektiv sammenlignet med SPR eller BLI.
De felteffekttransistorbaserede biosensorer (FET) er et voksende felt til påvisning af biomolekylære interaktioner ved at tilbyde forskellige biomedicinske applikationer. I FET-systemet immobiliseres målene til biosensorchipsene, og interaktioner detekteres ved ændringer i konduktans32. Det unikke træk, der skal tages i betragtning ved udviklingen af en effektiv elektronisk biosensor, er de fysisk-kemiske egenskaber såsom den halvledende karakter og kemiske stabilitet af det belægningsmateriale, der anvendes til fremstilling af sensoroverfladen33. Konventionelle materialer som silicium, der anvendes til FET, har begrænset sensorernes følsomhed, fordi det kræver oxidlag klemt inde mellem transistorkanalen og et specifikt miljø for korrekt funktion34. Desuden er siliciumtransistorer følsomme over for miljøer med højt saltindhold, hvilket gør det svært at måle biologiske interaktioner i deres naturlige miljø. Den grafenbaserede biosensor præsenteres som et alternativ, da den tilbyder fremragende kemisk stabilitet og elektrisk felt. Da grafen er et enkelt atomlag af kulstof, er det både ekstremt følsomt som en halvleder og kemisk kompatibelt med biologiske opløsninger; begge disse kvaliteter er ønskelige for at generere kompatible elektroniske biosensorer35. Det bemærkelsesværdige ultrahøje belastningspotentiale i biomolekyler, der tilbydes af grafenbelagte biosensorer, fører til udviklingen af grafenbaserede biosensorer FEB-teknologi.
Princippet om FEB-teknologi: FEB er en etiketfri biomolekylær detektionsteknik, der måler den elektriske strøm gennem grafenbiosensoren, som bindingsmålene immobiliseres til. Interaktioner mellem det immobiliserede protein og analytten resulterer i ændringer i strømmen, der overvåges i realtid, hvilket muliggør nøjagtige kinetiske målinger36.
Instrumentering: FEB-systemet består af en grafenfelteffekttransistor (gFET) sensorchip og en elektronisk læser, der anvender en konstant spænding gennem hele eksperimentet (figur 1). Analytten påføres i opløsning på målproteinet immobiliseret på biosensoroverfladen. Når en interaktion opstår, måles og registreres en ændring i strømmen i realtid. Efterhånden som analytkoncentrationen stiger, vil fraktionen af bundet analyt også stige, hvilket forårsager højere vekslinger i strømmen. Ved hjælp af den automatiserede analysesoftware, der følger med instrumentet (Table of Materials), måles og registreres I-Response i form af biosensingenheder (BU)37. I-Response defineres som ændringen i strømmen (I) gennem biosensorchippen målt i realtid ved interaktionen mellem det immobiliserede mål og analytten. DEN FEB automatiserede analysesoftware kan analysere både I-Response og C-Response til dynamiske interaktionshændelser, hvor C-Response registrerer ændringerne i kapacitansen (C). Variationerne i både I-Response og C-Response svarer direkte til brøkdelen af bundet analyt og kan analyseres yderligere for at generere KD-værdier . Den automatiserede analysesoftwares standardpræference er I-Response.
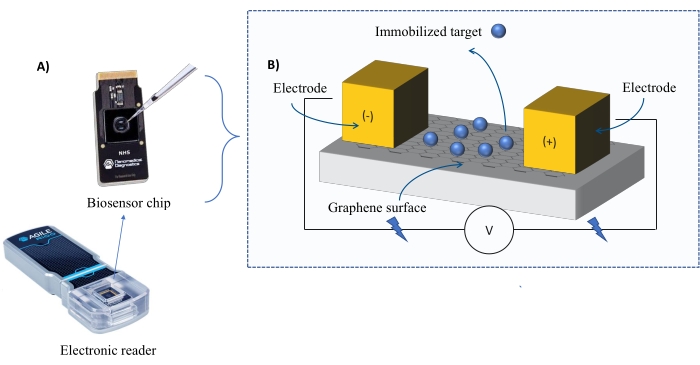
Figur 1: Oversigt over forsøgsopstillingen. (A) Grafenbaseret chip og elektronisk læser. (B) En oversigt over chipkomponenterne. Chippen er fastgjort til to elektroder, der leverer strøm til systemet. Overfladen af chippen er dækket af grafen, som, når den aktiveres, kan binde målet. Klik her for at se en større version af denne figur.
Metodik:
Indledningsvis indsættes den aktiverede biosensorchip i FEB-enheden (figur 1) efterfulgt af udførelsen af nedenstående trin: (1) Kalibrering: Eksperimentet starter med systemkalibrering ved anvendelse af 1x fosfatbufret saltvand (PBS; pH = 7,4) for at skabe baseline-ligevægtsresponsen. (2) Tilknytning: Analytten indføres i chippen, og I-Response overvåges, indtil bindingsmætningen er nået. (3) Dissociation: Analytten dissocieres ved hjælp af 1x PBS. (4) Regenerering: Rester af analytten fjernes ved hjælp af 1x PBS. (5) Vask: I alt fem vaske udføres ved hjælp af 1x PBS til grundig fjernelse af de bundne og ubundne analytter fra chippen.
Analyse:
Dataanalyse udføres ved hjælp af den fuldautomatiske software, der følger med instrumentet. Den automatiserede analysesoftware genererer et Hill fit-plot med en KD-værdi . Hill fit-plottet beskriver en analyts tilknytning til målproteinet som en funktion af analytkoncentrationer. Koncentrationen, ved hvilken der opnås et halvt maksimalt respons, er proportional med KD-værdien . En lav KD-værdi repræsenterer høj bindingsaffinitet og omvendt.
For at validere de data, der er opnået fra FEB-eksperimentet, ekstraheres I-Responses fra hvert udlæsningspunkt for hver analytkoncentration ved hjælp af datagennemgangs-/eksportsoftwaren og kan eksporteres til anden statistisk analysesoftware (se materialetabellen) som forklaret nedenfor.
Protocol
Representative Results
Discussion
I denne undersøgelse blev muligheden for at anvende FEB-teknologien (en kinetisk karakteriseringsmetode i realtid) evalueret for at bestemme den biomolekylære interaktion mellem Hsp90 og Cdc37. Det indledende sonderende eksperiment (første eksperiment) foreslog, at valg af de korrekte analytkoncentrationer er en kritisk del af eksperimentet, og at eksperimentet skulle designes ved at inkludere koncentrationspunkter over og under KD-værdien , som blev forudsagt ud fra de tilgængelige data i litteraturen.</…
Declarações
The authors have nothing to disclose.
Acknowledgements
Denne forskning blev støttet af en bevilling fra Binational Science Foundation (BSF) til S.K.S. og N.Q.
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
Referências
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Pesquisa do Câncer. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

