Utforska biomolekylär interaktion mellan molekylär chaperon Hsp90 och dess klientproteinkinas Cdc37 med hjälp av fälteffektbiosensingteknik
Summary
Field-effect biosensing (FEB) är en etikettfri teknik för att detektera biomolekylära interaktioner. Den mäter den elektriska strömmen genom grafenbiosensorn till vilken bindningsmålen är immobiliserade. FEB-tekniken användes för att utvärdera biomolekylära interaktioner mellan Hsp90 och Cdc37 och en stark interaktion mellan de två proteinerna detekterades.
Abstract
Biomolekylära interaktioner spelar mångsidiga roller i många cellulära processer genom att reglera och samordna funktionellt relevanta biologiska händelser. Biomolekyler som proteiner, kolhydrater, vitaminer, fettsyror, nukleinsyror och enzymer är grundläggande byggstenar i levande varelser; de samlas i komplexa nätverk i biosystem för att synkronisera en myriad av livshändelser. Proteiner använder vanligtvis komplexa interaktomnätverk för att utföra sina funktioner; därför är det obligatoriskt att utvärdera sådana interaktioner för att avslöja deras betydelse i celler på både cellulär och organismnivå. Mot detta mål introducerar vi en snabbt framväxande teknik, field-effect biosensing (FEB), för att bestämma specifika biomolekylära interaktioner. FEB är en bänkskiva, etikettfri och pålitlig biomolekylär detektionsteknik för att bestämma specifika interaktioner och använder högkvalitativa elektroniska baserade biosensorer. FEB-tekniken kan övervaka interaktioner i nanomolärt område på grund av de biokompatibla nanomaterial som används på dess biosensoryta. Som ett bevis på konceptet belyses protein-proteininteraktionen (PPI) mellan värmechockprotein 90 (Hsp90) och celldelningscykel 37 (Cdc37). Hsp90 är en ATP-beroende molekylär chaperon som spelar en viktig roll i vikning, stabilitet, mognad och kvalitetskontroll av många proteiner och därigenom reglerar flera vitala cellulära funktioner. Cdc37 betraktas som en proteinkinasspecifik molekylär chaperon, eftersom den specifikt känner igen och rekryterar proteinkinaser till Hsp90 för att reglera deras nedströms signaltransduktionsvägar. Som sådan anses Cdc37 vara en co-chaperone av Hsp90. Chaperon-kinasvägen (Hsp90 / Cdc37-komplexet) är hyperaktiverad vid flera maligniteter som främjar cellulär tillväxt; därför är det ett potentiellt mål för cancerbehandling. Föreliggande studie visar effektiviteten hos FEB-tekniken med hjälp av modellsystemet Hsp90/Cdc37. FEB detekterade en stark PPI mellan de två proteinerna (KD-värden på 0,014 μM, 0,053 μM och 0,072 μM i tre oberoende experiment). Sammanfattningsvis är FEB en etikettfri och kostnadseffektiv PPI-detekteringsplattform, som erbjuder snabba och exakta mätningar.
Introduction
Biomolekylära interaktioner:
Proteiner är väsentliga delar av organismer och deltar i många molekylära vägar som cellmetabolism, cellstruktur, cellsignalering, immunsvar, cellvidhäftning och mer. Medan vissa proteiner utför sin funktion (er) oberoende, interagerar de flesta proteiner med andra proteiner med hjälp av ett bindande gränssnitt för att samordna korrekt biologisk aktivitet1.
Biomolekylära interaktioner kan huvudsakligen klassificeras baserat på de distinkta strukturella och funktionella egenskaperna hos involverade proteiner2, till exempel baserat på proteinytorna, den komplexa stabiliteten eller persistensen av interaktioner3. Att identifiera essentiella proteiner och deras roller i biomolekylära interaktioner är avgörande för att förstå biokemiska mekanismer på molekylär nivå4. För närvarande finns det olika metoder för att upptäcka dessa interaktioner5: in vitro6, in silico7, i levande celler8, ex vivo9 och in vivo10 där var och en har sina egna styrkor och svagheter.
In vivo-analyserna utförs med hela djuret som ett experimentellt verktyg11, ocht he ex vivo-analyser utförs på vävnadsextrakt eller hela organ (t.ex. hjärta, hjärna, lever) i en kontrollerad yttre miljö genom att ge minimala förändringar under naturliga förhållanden. Den vanligaste tillämpningen av in vivo – och ex vivo-studier är att utvärdera farmakokinetiken, farmakodynamiken och toxicitetseffekterna av potentiella farmakologiska agens före försök på människa genom att säkerställa deras övergripande säkerhet och effekt12.
Biomolekylära interaktioner kan också detekteras inom levande celler. Avbildning av levande celler tillåter oss att observera dynamiska interaktioner när de utför reaktionerna hos en viss biokemisk väg13. Dessutom kan detektionstekniker, såsom bioluminescens eller fluorescensresonansenergiöverföring, ge information om var och när dessa interaktioner inträffar i cellen14. Även om detektion i levande celler erbjuder viktiga detaljer, är dessa detektionsmetoder beroende av optik och etiketter, vilket kanske inte återspeglar den ursprungliga biologin; De är också mindre kontrollerade än in vitro-metoder och kräver specialiserad expertis för att utföra15.
Beräkningsmetoderna in silico används främst för storskalig screening av målmolekyler före in vitro-experimenten . Beräkningsprediktionsmetoder, datorbaserade databaser, molekylär dockning, kvantitativa struktur-aktivitetsrelationer och andra molekylära dynamiksimuleringsmetoder är bland de väletablerade i silicoverktyg 16. Jämfört med mödosamma experimentella tekniker kan in silico-verktygen enkelt göra förutsägelser med hög känslighet, men med minskad noggrannhet i prediktiv prestanda17.
In vitro-analyser utförs med mikroorganismer eller biologiska molekyler utanför deras biologiska standardsammanhang. Att skildra biomolekylära interaktioner genom in vitro-metoder är avgörande för att förstå proteinfunktioner och biologin bakom det komplexa nätverket av cellfunktion. Den föredragna analysmetoden väljs utifrån proteinets inneboende egenskaper, kinetiska värden och interaktionsläget och intensiteten18,19.
Hsp90/Cdc37-interaktionen:
Chaperon-kinasvägen, som förbinder Hsp90 och Cdc37, är ett lovande terapeutiskt mål inom tumörbiologi20. Hsp90 spelar en central roll i cellcykelkontroll, proteinmontering, cellöverlevnad och signalvägar. Proteiner som är beroende av Hsp90 för sina funktioner levereras till Hsp90 för komplexering genom en co-chaperon, såsom Cdc37. Hsp90 / Cdc37-komplexet styr vikningen av de flesta proteinkinaser och fungerar som ett nav för en mängd intracellulära signalnätverk21. Det är ett lovande antitumörmål på grund av dess förhöjda uttryck i olika maligniteter, inklusive akut myeloblastisk leukemi, multipelt myelom och hepatocellulärt karcinom22,23.
Vanliga in vitro biomolekylära interaktionsdetekteringstekniker
Co-immunoprecipitation (co-IP) är en teknik som bygger på antigen-antikroppsspecificitet för att identifiera biologiskt relevanta interaktioner24. Den primära nackdelen med denna metod är dess oförmåga att detektera interaktioner med låg affinitet och kinetiska värden24. Biofysiska metoder som isotermisk titreringskalorimetri (ITC), ytplasmonresonans (SPR), biolagerinterferometri (BLI) och FEB-teknik föredras för att bestämma de kinetiska värdena.
ITC är en biofysisk detektionsmetod baserad på bestämning av bindande energi tillsammans med en fullständig termodynamikanalys för att karakterisera biomolekylära interaktioner25. Den främsta fördelen med ITC är att det inte kräver någon märkning eller fixering av målproteinet. De största svårigheterna som ITC stöter på är den höga koncentrationen av målprotein som krävs för ett experiment och svårigheten att analysera icke-kovalenta komplex på grund av små bindande entalpier26. Både SPR och BLI är etikettfria biofysiska tekniker som bygger på immobiliseringen av målmolekylen på sensorytan, följt av efterföljande injektioner av analyten över det immobiliserade målet27,28. I SPR mäts förändringar i brytningsindexet under biomolekylära interaktioner27; i BLI registreras interferensen i reflekterat ljus i realtid som en förändring i våglängden som en funktion av tid28. Både SPR och BLI delar gemensamma fördelar med att erbjuda hög specificitet, känslighet och detekteringsfunktioner29. I båda metoderna immobiliseras målproteinet på biosensorytor, och därför kan det finnas en viss förlust av målets ursprungliga konformation, vilket gör det svårt att skilja mellan specifika kontra icke-specifika interaktioner30. BLI använder dyra fiberoptiska engångsbiosensorer för att immobilisera målet och är därför en kostsam teknik31. Jämfört med dessa väletablerade biomolekylära detektionsverktyg erbjuder FEB-tekniken en pålitlig och etikettfri plattform genom att använda låga nanomolära koncentrationer för biomolekylär detektion i realtid med kinetisk karakterisering. FEB-tekniken övervinner också de bubblande utmaningarna i ITC och är mer kostnadseffektiv jämfört med SPR eller BLI.
De fälteffekttransistorbaserade biosensorerna (FET) är ett framväxande fält för att detektera biomolekylära interaktioner genom att erbjuda olika biomedicinska tillämpningar. I FET-systemet immobiliseras målen till biosensorchipsen och interaktioner detekteras genom förändringar i konduktans32. Den unika egenskapen som ska beaktas vid utvecklingen av en effektiv elektronisk biosensor är de fysikalisk-kemiska egenskaperna såsom den halvledande naturen och den kemiska stabiliteten hos beläggningsmaterialet som används för att tillverka sensorytan33. Konventionella material som kisel som används för FET har begränsat sensorernas känslighet eftersom det kräver oxidskikt inklämda mellan transistorkanalen och en specifik miljö för korrekt funktion34. Dessutom är kiseltransistorer känsliga för miljöer med högt salt, vilket gör det svårt att mäta biologiska interaktioner i sin naturliga miljö. Den grafenbaserade biosensorn presenteras som ett alternativ eftersom den erbjuder utmärkt kemisk stabilitet och elektriskt fält. Eftersom grafen är ett enda atomlager av kol är det både extremt känsligt som halvledare och kemiskt kompatibelt med biologiska lösningar; båda dessa egenskaper är önskvärda för att generera kompatibla elektroniska biosensorer35. Den anmärkningsvärda ultrahöga belastningspotentialen hos biomolekyler som erbjuds av grafenbelagda biosensorer leder till utvecklingen av grafenbaserade biosensorer FEB-teknik.
Principen för FEB-teknik: FEB är en etikettfri biomolekylär detektionsteknik som mäter den elektriska strömmen genom grafenbiosensorn till vilken bindningsmålen är immobiliserade. Interaktioner mellan det immobiliserade proteinet och analyten resulterar i förändringar i ström som övervakas i realtid, vilket möjliggör noggranna kinetiska mätningar36.
Instrumentering: FEB-systemet består av ett grafenfälteffekttransistor (gFET) sensorchip och en elektronisk läsare som tillämpar en konstant spänning under hela experimentet (figur 1). Analyten appliceras i lösning på målproteinet immobiliserat på biosensorytan. När en interaktion inträffar mäts och registreras en förändring i strömmen i realtid. När analytkoncentrationen ökar kommer fraktionen av bunden analyt också att öka, vilket orsakar högre växlingar i strömmen. Med hjälp av den automatiserade analysprogramvara som medföljer instrumentet (Table of Materials) mäts och registreras I-Response i termer av biosensingenheter (BU)37. I-Response definieras som förändringen i strömmen (I) genom biosensorchipet mätt i realtid vid interaktionen mellan det immobiliserade målet och analyten. Den automatiserade analysprogramvaran FEB kan analysera både I-Response och C-Response på dynamiska interaktionshändelser, där C-Response registrerar förändringarna i kapacitansen (C). Variationerna i både I-Response och C-Response motsvarar direkt fraktionen av bunden analyt och kan analyseras ytterligare för att generera KD-värden . Standardinställningen för den automatiserade analysprogramvaran är I-Response.
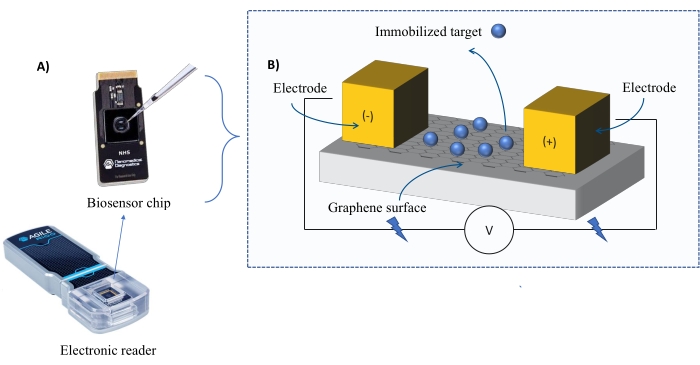
Figur 1: Översikt över den experimentella installationen. (A) Grafenbaserat chip och en elektronisk läsare. (B) En översikt över chipkomponenterna. Chipet är fäst vid två elektroder som levererar ström till systemet. Chipets yta är täckt med grafen, som när den aktiveras kan binda målet. Klicka här för att se en större version av denna figur.
Metodik:
Initialt sätts det aktiverade biosensorchipet in i FEB-enheten (figur 1) följt av utförandet av stegen som beskrivs nedan: (1) Kalibrering: Experimentet börjar med systemkalibrering med 1x fosfatbuffrad saltlösning (PBS; pH = 7.4) för att skapa baslinjens jämviktsrespons. (2) Förening: Analyten införs i chipet och I-responsen övervakas tills bindande mättnad uppnås. (3) Dissociation: Analyten dissocieras med 1x PBS. (4) Regenerering: Rester av analyten avlägsnas med 1x PBS. (5) Tvätt: Totalt fem tvättar utförs med 1x PBS för noggrant avlägsnande av de bundna och obundna analyterna från chipet.
Analys:
Dataanalys utförs med hjälp av den helautomatiska programvaran som medföljer instrumentet. Den automatiserade analysprogramvaran genererar en Hill-passformsplot med ett KD-värde . Hill fit plot beskriver föreningen av en analyt till målproteinet som en funktion av analytkoncentrationer. Koncentrationen vid vilken ett halvmaxiellt svar uppnås är proportionell mot KD-värdet . Ett lågt KD-värde representerar hög bindningsaffinitet och vice versa.
För att validera data som erhållits från FEB-experimentet extraheras I-svar från varje avläsningspunkt för varje analytkoncentration med hjälp av programvaran för datagranskning / export och kan exporteras till annan statistisk analysprogramvara (se materialtabell) som förklaras nedan.
Protocol
Representative Results
Discussion
I denna studie utvärderades möjligheten att använda FEB-tekniken (en kinetisk karakteriseringsmetod i realtid) för att bestämma den biomolekylära interaktionen mellan Hsp90 och Cdc37. Det första undersökande experimentet (första experimentet) föreslog att valet av rätt analytkoncentrationer är en kritisk del av experimentet och att experimentet bör utformas genom att inkludera koncentrationspunkter över och under KD-värdet , vilket förutspåddes baserat på de data som finns tillgängliga i lit…
Declarações
The authors have nothing to disclose.
Acknowledgements
Denna forskning stöddes av ett bidrag från Binational Science Foundation (BSF) till S.K.S. och N.Q.
Materials
| Automated analysis software | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
Referred to in the text as the automated analysis software supplied with the instrument. Generates automated analysis. |
| COOH-BPU (Biosensing Processing Unit) | Agile plus software, Cardea (Nanomed) | NA CAS number: NA |
biosensor chip |
| Data review software | Datalign 1.0, Cardea (Nanomed) | NA CAS number: NA |
Referred to as the supplied data review software in the text. Supplied with the instrument and allows to review and export the information data points. |
| Dialysis bag | CelluSep, Membrane filtration products | T2-10-15 CAS number: NA |
T2 tubings (6,000-8,000 MWCO), (10 mm fw, 6.4mm Ø, 0.32ml/cm, 15m) |
| EDC (1-Ethyl-3-(3-dimethylamino propyl) carbodiimide) | Cardea (Nanomed) | EDC160322-02 CAS number: 25952-53-8 |
White powder |
| ITC (Isothermal titration calorimetry) system | Microcal-PEAQ-ITC (Malvern, United Kingdom) | NA CAS number: NA |
|
| MES (2-(N-morpholino) ethane sulfonic acid) buffer | Merck | M3671-50G CAS number: 4432-31-9 |
White powder |
| NHS (N-Hydroxysulfosuccinimide) chips | Cardea (Nanomed) | NA CAS number: NA |
Graphene-based chip |
| PBS (Phosphate-buffered saline) X 10 | Bio-Lab | 001623237500 CAS number: 7758-11-4 |
Liquid transparent solution |
| Pipete | Thermo Scientific | 11855231 CAS number: NA |
Finnpipette F3 5-50 µL, yellow |
| Quench 1 (3.9 mM amino-PEG5-alcohol in 1 X PBS) | Cardea (Nanomed) | 0105-001-002-001 CAS number: NA |
Liquid, transparent solution |
| Quench 2 (1 M ethanolamine (pH=8.5)) | Cardea (Nanomed) | 0105-001-003-001 CAS number: NA |
Liquid, transparent solution |
| Recombinant protein Cdc37 | Abcam | ab256157 CAS number: NA |
|
| Recombinant protein Hsp90 beta | Abcam | ab80033 CAS number: NA |
|
| Spreadsheet | Excel, Microsoft office | NA CAS number: NA |
|
| Statistical software | GraphPad, Prism | NA CAS number: NA |
Referred to as the other statistical software. Sigma plot, phyton or other statistical programes may also be used |
| Sulfo-NHS | Cardea (Nanomed) | NHS160321-07 CAS number: 106627-54-7 |
White powder |
| Tips | Alex red | LC 1093-800-000 CAS number: NA |
Tip 1-200 µl, in bulk, 1,000 pcs |
Referências
- Tuncbag, N., Gursoy, A., Guney, E., Nussinov, R., Keskin, O. Architectures and functional coverage of protein-protein interfaces. Journal of Molecular Biology. 381 (3), 785-802 (2008).
- Berggård, T., Linse, S., James, P. Methods for the detection and analysis of protein–protein interactions. Proteomics. 7 (16), 2833-2842 (2007).
- Magliery, T. J., et al. Detecting protein-protein interactions with a green fluorescent protein fragment reassembly trap: Scope and mechanism. Journal of the American Chemical Society. 127 (1), 146-157 (2005).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Nguyen, T. N., Goodrich, J. A. Protein-protein interaction assays: Eliminating false positive interactions. Nature Methods. 3 (2), 135-139 (2006).
- Fernández-Suárez, M., Chen, T. S., Ting, A. Y. Protein-protein interaction detection in vitro and in cells by proximity biotinylation. Journal of the American Chemical Society. 130 (29), 9251-9253 (2008).
- Jiang, M., Niu, C., Cao, J., Ni, D. -. A., Chu, Z. In silico-prediction of protein–protein interactions network about MAPKs and PP2Cs reveals a novel docking site variants in Brachypodium distachyon. Scientific Reports. 8 (1), 15083 (2018).
- Yazawa, M., Sadaghiani, A. M., Hsueh, B., Dolmetsch, R. E. Induction of protein-protein interactions in live cells using light. Nature Biotechnology. 27 (10), 941-945 (2009).
- Wang, W., Goodman, M. T. Antioxidant property of dietary phenolic agents in a human LDL-oxidation ex vivo model: Interaction of protein binding activity. Nutrition Research. 19 (2), 191-202 (1999).
- Xing, S., Wallmeroth, N., Berendzen, K. W., Grefen, C. Techniques for the analysis of protein-protein interactions in vivo. Plant Physiology. 171 (2), 727-758 (2016).
- Qvit, N., Disatnik, M. -. H., Sho, E., Mochly-Rosen, D. Selective phosphorylation inhibitor of delta protein kinase C–Pyruvate dehydrogenase kinase protein–protein interactions: Application for myocardial injury in vivo. Journal of the American Chemical Society. 138 (24), 7626-7635 (2016).
- Alam, M. N., Bristi, N. J., Rafiquzzaman, M. Review on in vivo and in vitro methods evaluation of antioxidant activity. Saudi Pharmaceutical Journal. 21 (2), 143-152 (2013).
- Paulmurugan, R., Gambhir, S. S. Novel fusion protein approach for efficient high-throughput screening of small molecule–mediating protein-protein interactions in cells and living animals. Pesquisa do Câncer. 65 (16), 7413-7420 (2005).
- Boute, N., Jockers, R., Issad, T. The use of resonance energy transfer in high-throughput screening: BRET versus FRET. Trends in Pharmacological Sciences. 23 (8), 351-354 (2002).
- Deriziotis, P., Graham, S. A., Estruch, S. B., Fisher, S. E. Investigating protein-protein interactions in live cells using bioluminescence resonance energy transfer. Journal of Visualized Experiments: JoVE. (87), e51438 (2014).
- Ekins, S., Mestres, J., Testa, B. In silico pharmacology for drug discovery: Methods for virtual ligand screening and profiling. British Journal of Pharmacology. 152 (1), 9-20 (2007).
- Valerio, L. G. Application of advanced in silico methods for predictive modeling and information integration. Expert Opinion on Drug Metabolism & Toxicology. 8 (4), 395-398 (2012).
- Piehler, J. New methodologies for measuring protein interactions in vivo and in vitro. Current Opinion in Structural Biology. 15 (1), 4-14 (2005).
- Ideker, T., Sharan, R. Protein networks in disease. Genome Research. 18 (4), 644-652 (2008).
- Lu, H., et al. Recent advances in the development of protein–protein interactions modulators: mechanisms and clinical trials. Signal Transduction and Targeted Therapy. 5 (1), 213 (2020).
- Jarosz, D. Hsp90: A global regulator of the genotype-to-phenotype map in cancers. Advances in Cancer Research. 129, 225-247 (2016).
- Johnson, V. A., Singh, E. K., Nazarova, L. A., Alexander, L. D., McAlpine, S. R. Macrocyclic inhibitors of Hsp90. Current Topics in Medicinal Chemistry. 10 (14), 1380-1402 (2010).
- Mahalingam, D., et al. Targeting HSP90 for cancer therapy. British Journal of Cancer. 100 (10), 1523-1529 (2009).
- Stewart, A., Fisher, R. A. Co-Immunoprecipitation: Isolation of protein signaling complexes from native tissues. Methods in Cell Biology. 112, 33-54 (2012).
- Pierce, M. M., Raman, C. S., Nall, B. T. Isothermal titration calorimetry of protein-protein interactions. Methods. 19 (2), 213-221 (1999).
- Paketurytė, V., et al. Inhibitor binding to carbonic anhydrases by isothermal titration calorimetry. Carbonic Anhydrase as Drug Target. , 79-95 (2019).
- Grote, J., Dankbar, N., Gedig, E., Koenig, S. Surface plasmon resonance/mass spectrometry interface. Analytical Chemistry. 77 (4), 1157-1162 (2005).
- Kumaraswamy, S., Tobias, R. Label-free kinetic analysis of an antibody–antigen interaction using biolayer interferometry. Methods in Molecular Biology. , 165-182 (2015).
- Wallner, J., Lhota, G., Jeschek, D., Mader, A., Vorauer-Uhl, K. Application of bio-layer interferometry for the analysis of protein/liposome interactions. Journal of Pharmaceutical and Biomedical Analysis. 72, 150-154 (2013).
- Singh, A. N., Ramadan, K., Singh, S. Experimental methods to study the kinetics of protein–protein interactions. Advances in Protein Molecular and Structural Biology Methods. , 115-124 (2022).
- Frenzel, D., Willbold, D. Kinetic titration series with biolayer interferometry. PLoS One. 9 (9), 106882 (2014).
- Vu, C. -. A., Chen, W. -. Y. Field-effect transistor biosensors for biomedical applications: Recent advances and future prospects. Sensors. 19 (19), 4214 (2019).
- Bergveld, P. A critical evaluation of direct electrical protein detection methods. Biosensors & Bioelectronics. 6 (1), 55-72 (1991).
- Lowe, B. M., Sun, K., Zeimpekis, I., Skylaris, C. K., Green, N. G. Field-effect sensors – from pH sensing to biosensing: sensitivity enhancement using streptavidin–biotin as a model system. The Analyst. 142 (22), 4173-4200 (2017).
- Goldsmith, B. R., et al. Digital biosensing by foundry-fabricated graphene sensors. Scientific Reports. 9 (1), 434 (2019).
- Afsahi, S., et al. Novel graphene-based biosensor for early detection of Zika virus infection. Biosensors and Bioelectronics. 100, 85-88 (2018).
- Afsahi, S. J., et al. Towards novel graphene-enabled diagnostic assays with improved signal-to-noise ratio. MRS Advances. 2 (60), 3733-3739 (2017).
- Roe, S. M., et al. The mechanism of Hsp90 regulation by the protein kinase-specific cochaperone p50cdc37. Cell. 116 (1), 87-98 (2004).
- Gaiser, A. M., Kretzschmar, A., Richter, K. Cdc37-Hsp90 complexes are responsive to nucleotide-induced conformational changes and binding of further cofactors. The Journal of Biological Chemistry. 285 (52), 40921-40932 (2010).
- Popescu, A. I., Găzdaru, D. M., Chilom, C. G., Bacalum, M. Biophysical interactions: Their paramount importance for life. Romanian Reports in Physics. 65 (3), 1063-1077 (2013).
- Surya, S., Abhilash, J., Geethanandan, K., Sadasivan, C., Haridas, M. A profile of protein-protein interaction: Crystal structure of a lectin-lectin complex. International Journal of Biological Macromolecules. 87, 529-536 (2016).
- Velazquez-Campoy, A., Freire, E. ITC in the post-genomic era…? Priceless. Biophysical Chemistry. 115 (23), 115-124 (2005).
- Concepcion, J., et al. Label-free detection of biomolecular interactions using bioLayer interferometry for kinetic characterization. Combinatorial Chemistry & High Throughput Screening. 12 (8), 791-800 (2009).
- Helmerhorst, E., Chandler, D. J., Nussio, M., Mamotte, C. D. Real-time and label-free bio-sensing of molecular interactions by surface plasmon resonance: A laboratory medicine perspective. The Clinical Biochemist. Reviews. 33 (4), 161-173 (2012).
- Jacob, N. T., et al. Synthetic molecules for disruption of the MYC protein-protein interface. Bioorganic & Medicinal Chemistry. 26 (14), 4234-4239 (2018).

