RPA-CRISPR/Cas12a-SPM ve Derin Öğrenmeye Dayalı DNA Virüs Tespit Sistemi
Summary
DNA virüslerinin eser tespiti için rekombinaz polimeraz amplifikasyonunu bir CRISPR/Cas12a sistemi ile birleştiren bir protokol sunuyoruz ve hasta başı DNA virüsü tespiti için yapay zeka destekli bir sınıflandırma ile taşınabilir akıllı telefon mikroskobu oluşturuyoruz.
Abstract
DNA virüslerinin eser tespiti için rekombinaz polimeraz amplifikasyonu (RPA) ve CRISPR/Cas12a sistemini birleştiren hızlı, uygulaması kolay, son derece hassas, diziye özgü ve bakım noktası (POC) DNA virüsü tespit sistemini rapor ediyoruz. Hedef DNA, RPA ve CRISPR/Cas12a tarafından ayrı ayrı amplifiye edilir ve tanınır, bu da Cas12a’nın florofor söndürücü etiketli bir DNA raportörünü parçalayan ve floresansı genelleştiren kollateral bölünme aktivitesini tetikler. POC tespiti için taşınabilir akıllı telefon mikroskobu, floresan görüntüler çekmek üzere üretilmiştir. Ayrıca, pozitif veya negatif numunelerin ikili sınıflandırması için derin öğrenme modelleri, yüksek doğruluk elde ederek sistem içinde konuşlandırılır. Kurbağa virüsü 3 (FV3, cins Ranavirus, Iridoviridae familyası) bu DNA virüsü POC tespit sistemi için bir örnek olarak test edilmiştir ve tespit sınırları (LoD) 40 dakika içinde 10’ye ulaşabilir. Yetenekli operatörler ve hantal cihazlar olmadan, yapay zeka (AI) destekli sınıflandırmaya sahip taşınabilir ve minyatür RPA-CRISPR/Cas12a-SPM, POC DNA virüsü tespiti için büyük potansiyel gösterir ve bu tür virüslerin yayılmasını önlemeye yardımcı olabilir.
Introduction
Son yıllarda, 20141 ve 20182’de Ebola virüsü hastalığı (EVH) salgını, 2015’te Orta Doğu Solunum Sendromu (MERS)3, 2015’te Zika virüsü hastalığı salgını4, şiddetli akut solunum sendromu koronavirüs 2’nin neden olduğu Koronavirüs hastalığı 2019 (COVID-19) dahil olmak üzere farklı virüslerin neden olduğu bulaşıcı hastalık salgınları sık sık meydana gelmiştir.5 ve 2022’de Maymun Çiçeği virüsünün (MKPV) neden olduğu devam eden Maymun Çiçeği (MKPV)6‘dir. Salgın bulaşıcı hastalıkların bu ani salgınları çok sayıda ölüme neden olmakta ve büyük ekonomik kayıplar ve sosyal huzursuzluk getirmektedir. Enfeksiyonu hızlı bir şekilde teşhis etmek ve virüsün daha fazla yayılmasını önlemek için acilen hızlı ve doğru bir tespit sistemi gereklidir.
Son zamanlarda, kümelenmiş düzenli aralıklı kısa palindromik tekrarlar (CRISPR) ve CRISPR ile ilişkili (Cas) proteinler dünya çapında ilgi görmüş ve nükleik asit tespitinde umut verici sonuçlar göstermiştir 7,8,9,10,11,12,13,14,15 . CRISPR RNA (crRNA) tarafından yönlendirilen CRISPR/Cas12a proteini, hedef DNA’ya bağlanır ve onu parçalar. Bu aktivite, trans-bölünme olarak bilinen spesifik olmayan tek sarmallı DNA’nın (ssDNA) salınmasına yol açar ve nükleik asit tespiti için algılama sinyalini geliştirmek için kullanılabilir. Polimeraz zincir reaksiyonu (PCR), kantitatif gerçek zamanlı PCR (qPCR) ve enzime bağlı immünosorbent testi (ELISA) gibi bazı geleneksel tespit yöntemleri, bakım noktası (POC) tespiti için karmaşık, zaman alıcı ve maliyetlidir. Önceki çalışmamız, CRISPR / Cas12a teknolojisine dayalı Afrika domuz ateşi virüsü (ASFV) için otomatik, entegre ve uygun maliyetli bir tespit sistemini başarıyla geliştirdi. Bu sistemde, amplifikasyona ihtiyaç duymadan 2 saatlik bir zaman dilimi içinde 1 pM’lik bir algılama limitine ulaştık. CRISPR/Cas12a sistemi ve rekombinaz polimeraz amplifikasyonu (RPA), eser DNA tespiti için duyarlılığı ve özgüllüğü geliştirmek üzere birleştirilmiştir. Diğer izotermal amplifikasyon teknikleriyle karşılaştırıldığında, RPA’nın tasarımı basittir ve karmaşık sıcaklık kontrol ekipmanı olmadan daha kısa bir reaksiyon süresine sahip olduğu için kullanımı kolaydır.
Patojenlerin POC tespiti için, sonuç okumaları 16,17,18 için akıllı telefon mikroskobu (SPM), el tipi florimetre veya yanal akış şeritleri gibi aletler geliştirilmiştir. SPM, görüntüleri bir kamera aracılığıyla yakalar ve hızlı veri analizi için bazı mobil uygulamalara yükler. Bu tür bir mikroskopi, yüksek hassasiyete sahip taşınabilir, ucuz ve minyatür bir sinyal toplama sistemi yapar ve H5N1, Zika virüsü ve SARS-CoV-219,20 gibi patojenleri tespit etmede avantajlar göstermiştir. Bu nedenle, hedef DNA virüsünün RPA-CRISPR/Cas12a tespiti tarafından tetiklenen floresan sinyallerini yakalamak için taşınabilir bir SPM oluşturuyoruz. Bir florofor ve bir söndürücüyü birbirine bağlayan ssDNA raportör probu, CRISPR / Cas12a hedef DNA virüsünü tanıdığında parçalanacak ve florofor tarafından yayılan floresan SPM tarafından yakalanabilir.
SPM21’den gelen floresan görüntülerinden sonuç bilgilerini elde etmek için genellikle kullanılan profesyonel yazılımla karşılaştırıldığında, bazı uzmanlar, floresan görüntüleri22 elde ettikten sonra virüs DNA konsantrasyonlarını ölçmek için makine öğrenimi ve derin öğrenmeyi kullanır, bu da daha fazla zaman alıcıdır. Tıbbi görüntülerin sınıflandırılması söz konusu olduğunda, geleneksel sinir ağları (CNN’ler) genellikle ham pikselli görüntülerden uçtan uca bir şekilde özellikleri öğrenmek için kullanılır 23,24,25,26. AlexNet, DenseNet-121 ve EfficientNet-B7 gibi popüler CNN tabanlı derin öğrenme modelleri bu alanda başarıyla uygulanmıştır27,28. Bununla birlikte, belirli alanlarda büyük veri setleri elde etmek zor olabilir ve bu da transfer öğrenmeyi gerektirir29,30. Bu yaklaşım, büyük bir veri kümesine sahip bir derin öğrenme modelini önceden eğitir ve önceden eğitilmiş model, küçük bir veri kümesine sahip yeni bir görev için başlangıç noktası olarak kullanılır. Bu teknik, büyük veri kümelerine olan ihtiyacı azaltabilir, aşırı öğrenmeyle mücadele edebilir ve eğitim süresini azaltabilir31. Burada, pozitif ve negatif numunelerin floresan görüntülerinin ikili sınıflandırması için transfer öğrenmeli derin öğrenme modellerini kullanıyoruz.
Bu yöntemde, DNA virüslerinin iz tespiti için RPA ve CRISPR/Cas12a sistemini birleştiriyoruz. Hedef DNA, RPA ve CRISPR/Cas12a tarafından ayrı ayrı amplifiye edilir ve tanınır, bu da Cas12a’nın florofor söndürücü etiketli bir DNA raportörünü parçalayan ve floresansı genelleştiren kollateral bölünme aktivitesini tetikler. POC tespiti için floresan görüntüleri almak ve ikili sınıflandırma için derin öğrenme modelleri geliştirmek için taşınabilir bir SPM oluşturuyoruz. Yerleşik POC algılama sisteminin şeması Şekil 1’de gösterilmiştir. Yetenekli operatörler ve hantal cihazlar olmadan, yapay zeka (AI) destekli sınıflandırmaya sahip RPA-CRISPR/Cas12a-SPM, POC DNA virüsü tespiti için büyük potansiyel gösterir.
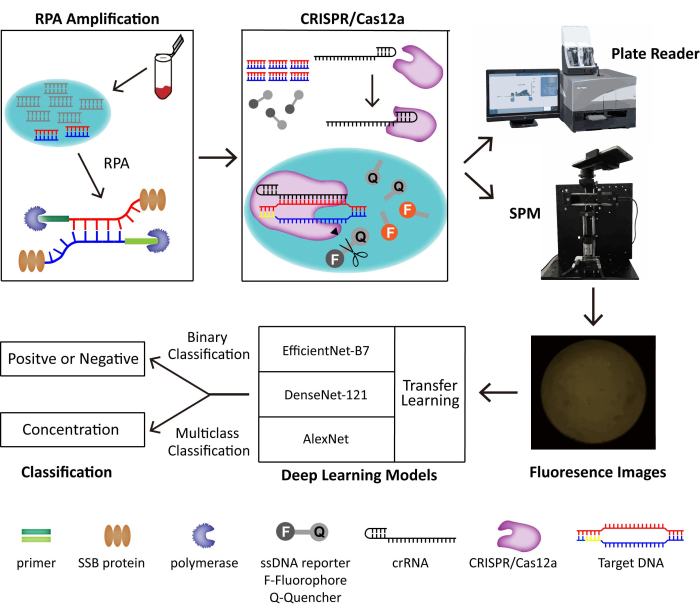
Şekil 1: RPA-CRISPR/Cas12-SPM algılama sisteminin şeması ve toplanan görüntüler için yapay zeka sınıflandırması. Hayvansal kaynaklı örneklerin nükleik asitleri PINDBK tarafından salınır. Virüsün Hedef DNA’sı, RPA-CRISPR/Cas12a sistemi tarafından özel olarak amplifiye edilir ve tanınır. CRISPR/Cas12a, crRNA ile bağlanır ve Cas12a-crRNA kompleksi, hedef DNA ile bağlanır, bu da ssDNA raportör problarında CRISPR/Cas12a’nın kollateral bölünmesini tetikler. Raportör üzerindeki florofor serbest bırakılır ve floresan, ticarileştirilmiş bir plaka okuyucu veya oluşturduğumuz SPM tarafından algılanır. Floresan görüntülerini sınıflandırmak için AlexNet, DenseNet-121 ve transfer öğrenmeli EfficientNet-B7 dahil olmak üzere üç farklı derin öğrenme modeli kullanılır. Bu figür Lei ve ark.35’in izniyle yeniden kullanılmıştır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
Bu yöntemde yapay zeka yardımıyla hızlı, uygulaması kolay, son derece hassas, diziye özgü ve POC DNA virüsü tespit sistemi geliştiriyoruz. Numuneler alındıktan sonra, hedef diziyi amplifiye etmek için RPA uygulanır ve ardından CRISPR/Cas12a, hedef DNA’yı tanıyabilir ve algılama sinyalini büyüten floresan serbest bırakabilir. Taşınabilir akıllı telefon mikroskobu, floresan görüntüleri almak için üretilmiştir ve pozitif ve negatif numunelerin görüntülerinin ikili sınıflandırması için…
Declarações
The authors have nothing to disclose.
Acknowledgements
Bu çalışma, Çin Ulusal Doğa Bilimleri Vakfı 31970752, Shenzhen Belediyesi Bilim, Teknoloji, İnovasyon Komisyonu JCYJ20190809180003689, JSGG20200225150707332, JSGG20191129110812708, WDZC20200820173710001; Shenzhen Körfezi Laboratuvarı Açık Finansmanı, SZBL2020090501004; Çin Doktora Sonrası Bilim Vakfı 2020M680023; ve Çin Halk Cumhuriyeti Gümrük Genel İdaresi 2021HK007.
Materials
| 20x amplification | OLYMPUS | OPLN20X | |
| 532 nm green laser | Thorlabs | PL201 | with 0.9 mW output power |
| 535 nm cutoff wavelength | chrome | AT535 | |
| 6x DNA loading buffer | Thermo scientific | R0611 | |
| 96-well black microplate | Corning Incorporated | 3603 | Black with flat clear bottom |
| Aspherical lens | Lubang | N/A | |
| Bandpass filter | SEMROCK | FF01-542/27-25 | |
| Bsu DNA Polymerase | ATG Biotechnology | M103 | Large Fragment |
| crRNA | Sangon Biotech | N/A | |
| DNA fragments | Sangon Biotech | N/A | |
| Dichroic holders | Ruicage | N/A | |
| Dichroic mirror | SEMROCK | FF555-Di03-25×36 | with a cutoff wavelength of 535 nm |
| E.Z.N.A Gel Extraction Kit | Omega Biotek | D2500-02 | |
| EnGen Lba Cas12a (Cpf1) | New England Biolabs (Beijing) LTD | M0653T | |
| Filter holders | Ruicage | N/A | |
| Fluorophore-ssDNA-Quencher reporter probes | Sangon Biotech | N/A | TAMRA (carboxy tetramethylrhodamine) as the fluorophore at the 5 ends; BHQ2 (Black Hole Quencher-2) as the quencher at the 3 ends |
| GP32 | ATG Biotechnology | M104 | |
| ImageJ | Open-source | Version 1.53t 24 | Downloaded from https://imagej.nih.gov/ij/ |
| Microplate reader | SPARK, TECAN | N/A | |
| Multi-Block thermal Cycler PCR instrument | LongGene | N/A | |
| NanoDrop 2000/2000c Spectrophotometers | Thermo Scientific | ND-2000 | |
| NEBuffer r2.1 | New England Biolabs (Beijing) LTD | B6002S | 10x CRISPR/Cas12a Reaction buffer |
| Oxygen plasma treatment | Electro-Technic Products | N/A | |
| Pathogen Inactivate, Nucleic acid extraction-free, Direct-to-PCR Buffer with Proteinase K (PINDBK) | Ebio | PINDBK -25mL | |
| PCR primer pairs | Sangon Biotech | N/A | |
| PDMS | Dow Corning | Sylgard 184 | |
| RPA primer pairs | Sangon Biotech | N/A | |
| Smartphone | Huawei | Mate10 | |
| Translation stages | Ruicage | N/A | |
| Transmitted neutral density filters | Thorlabs | ND40A | |
| Triplet achromatic lenses | Thorlabs | TRH127-020-A | |
| UvsX | ATG Biotechnology | M105 | |
| UvsY | ATG Biotechnology | M106 |
Referências
- Gire, S. K., et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science. 345 (6202), 1369-1372 (2014).
- The Ebola Outbreak Epidemiology Team. Outbreak of Ebola virus disease in the Democratic Republic of the Congo, April-May 2018: an epidemiological study. Lancet. 392 (10143), 213-221 (2018).
- Zumla, A., Hui, D. S., Perlman, S. Middle East respiratory syndrome. Lancet. 386 (9997), 995-1007 (2015).
- Plourde, A. R., Bloch, E. M. A literature review of Zika virus. Emerg Infect Dis. 22 (7), 1185-1192 (2016).
- Yuan, X., et al. Current and perspective diagnostic techniques for COVID-19. ACS Infect Dis. 6 (8), 1998-2016 (2020).
- Minhaj, F. S., et al. Monkeypox outbreak – nine states, May 2022. MMWR Morb Mortal Wkly Rep. 71 (23), 764-769 (2022).
- Bao, M., et al. Challenges and opportunities for clustered regularly interspaced short palindromic repeats based molecular biosensing. ACS Sens. 6 (7), 2497-2522 (2021).
- Broughton, J. P., et al. CRISPR-Cas12-based detection of SARS-CoV-2. Nat Biotechnol. 38 (7), 870-874 (2020).
- Chen, J. S., et al. CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity. Science. 360 (6387), 436-439 (2018).
- Gootenberg, J. S., et al. Nucleic acid detection with CRISPR-Cas13a/C2c2. Science. 356 (6336), 438-442 (2017).
- Kellner, M. J., Koob, J. G., Gootenberg, J. S., Abudayyeh, O. O., Zhang, F. SHERLOCK: nucleic acid detection with CRISPR nucleases. Nat Protoc. 14 (10), 2986-3012 (2019).
- Mukama, O., et al. An ultrasensitive and specific point-of-care CRISPR/Cas12 based lateral flow biosensor for the rapid detection of nucleic acids. Biosens Bioelectron. 159, 112143 (2020).
- Schwank, G., et al. Functional repair of CFTR by CRISPR/Cas9 in intestinal stem cell organoids of cystic fibrosis patients. Cell Stem Cell. 13 (6), 653-658 (2013).
- Yin, L., Man, S., Ye, S., Liu, G., Ma, L. CRISPR-Cas based virus detection: Recent advances and perspectives. Biosens Bioelectron. 193, 113541 (2021).
- Dronina, J., Bubniene, U. S., Ramanavicius, A. The application of DNA polymerases and Cas9 as representative of DNA-modifying enzymes group in DNA sensor design (review). Biosens Bioelectron. 175, 112867 (2021).
- Fozouni, P., et al. Amplification-free detection of SARS-CoV-2 with CRISPR-Cas13a and mobile phone microscopy. Cell. 184 (2), 323-333.e9 (2021).
- Kumar, M., et al. FnCas9-based CRISPR diagnostic for rapid and accurate detection of major SARS-CoV-2 variants on a paper strip. eLife. 10, e67130 (2021).
- Lee, R. A., et al. Ultrasensitive CRISPR-based diagnostic for field-applicable detection of Plasmodium species in symptomatic and asymptomatic malaria. Proc Natl Acad Sci U S A. 117 (41), 25722-25731 (2020).
- Ganguli, A., et al. Hands-free smartphone-based diagnostics for simultaneous detection of Zika, Chikungunya, and Dengue at point-of-care. Biomed Microdevices. 19 (4), 73 (2017).
- Yeo, S. J., et al. Smartphone-based fluorescent diagnostic system for highly pathogenic H5N1 viruses. Theranostics. 6 (2), 231-242 (2016).
- von Chamier, L., et al. Democratising deep learning for microscopy with ZeroCostDL4Mic. Nat Commun. 12 (1), 2276 (2021).
- Shiaelis, N., et al. Virus detection and identification in minutes using single-particle imaging and deep learning. ACS Nano. 17 (1), 697-710 (2020).
- Liu, Y., et al. Mixed-UNet: Refined class activation mapping for weakly-supervised semantic segmentation with multi-scale inference. Front. Comput. Sci. 4, 1036934 (2022).
- Lawrimore, J., Doshi, A., Walker, B., Bloom, K. AI-assisted forward modeling of biological structures. Front Cell Dev Biol. 7, 279 (2019).
- Yang, Y., Hu, Y., Zhang, X., Wang, S. Two-stage selective ensemble of CNN via deep tree training for medical image classification. IEEE Trans Cybern. 52 (9), 9194-9207 (2022).
- Zhang, R., et al. RCMNet: A deep learning model assists CAR-T therapy for leukemia. Comput Biol Med. 150, 106084 (2022).
- Xie, Y., et al. Stroke prediction from electrocardiograms by deep neural network. Multimed Tools Appl. 80, 17291-17297 (2021).
- Wang, J., Zhu, H., Wang, S., Zhang, Y. -. D. A review of deep learning on medical image analysis. Mobile Netw Appl. 26, 351-380 (2021).
- Artoni, P., et al. Deep learning of spontaneous arousal fluctuations detects early cholinergic defects across neurodevelopmental mouse models and patients. Proc Natl Acad Sci U S A. 117 (38), 23298-23303 (2020).
- Li, J., et al. DeepLearnMOR: a deep-learning framework for fluorescence image-based classification of organelle morphology. Plant Physiol. 186 (4), 1786-1799 (2021).
- Yosinski, J., Clune, J., Bengio, Y., Lipson, H. How transferable are features in deep neural networks. Proceedings of the 27th International Conference on Neural Information Processing Systems. 2, 3320-3328 (2014).
- He, Q., et al. High-throughput and all-solution phase African Swine Fever Virus (ASFV) detection using CRISPR-Cas12a and fluorescence based point-of-care system. Biosens Bioelectron. 154, 112068 (2020).
- Krizhevsky, A., Sutskever, I., Hinton, G. E. ImageNet classification with deep convolutional neural networks. Commun. ACM. 60 (6), 84-90 (2017).
- Lawton, S., Viriri, S. Detection of COVID-19 from CT lung scans using transfer learning. Comput Intell Neurosci. 2021, 5527923 (2021).
- Lei, Z., et al. Detection of frog virus 3 by integrating RPA-CRISPR/Cas12a-SPM with deep learning. ACS Omega. 8 (36), 32555-32564 (2023).
- Chen, Z., Huang, J., Zhang, F., Zhou, Y., Huang, H. Detection of shrimp hemocyte iridescent virus by recombinase polymerase amplification assay. Mol Cell Probes. 49, 101475 (2020).
- Fu, X., Sun, J., Ye, Y., Zhang, Y., Sun, X. A rapid and ultrasensitive dual detection platform based on Cas12a for simultaneous detection of virulence and resistance genes of drug-resistant Salmonella. Biosens Bioelectron. 195, 113682 (2022).
- Habimana, J. D., et al. Mechanistic insights of CRISPR/Cas nucleases for programmable targeting and early-stage diagnosis: A review. Biosens Bioelectron. 203, 114033 (2022).
- Liang, Y., Lin, H., Zou, L., Deng, X., Tang, S. Rapid detection and tracking of Omicron variant of SARS-CoV-2 using CRISPR-Cas12a-based assay. Biosens Bioelectron. 205, 114098 (2022).
- Sivaraman, D., Biswas, P., Cella, L. N., Yates, M. V., Chen, W. Detecting RNA viruses in living mammalian cells by fluorescence microscopy. Trends Biotechnol. 29 (7), 307-313 (2011).
- Wang, I. H., Burckhardt, C. J., Yakimovich, A., Greber, U. F. Imaging, tracking and computational analyses of virus entry and egress with the cytoskeleton. Viruses. 10 (4), 166 (2018).

