Ein Open-Source-Framework für die Massenberechnung von Antikörper-basierten therapeutischen Molekülen
Summary
Dieser Artikel beschreibt die Verwendung einer Softwareanwendung, mAbScale, für die Berechnung von Massen für monoklonale Antikörper-basierte Proteintherapeutika.
Abstract
Biotherapeutische Massen sind ein Mittel zur Überprüfung der Identität und der strukturellen Integrität. Die Massenspektrometrie (MS) von intakten Proteinen oder Proteinuntereinheiten bietet ein einfaches Analysewerkzeug für verschiedene Stadien der biopharmazeutischen Entwicklung. Die Identität des Proteins wird bestätigt, wenn die experimentelle Masse aus MS innerhalb eines vordefinierten Massenfehlerbereichs der theoretischen Masse liegt. Es gibt zwar mehrere Berechnungswerkzeuge für die Berechnung von Protein- und Peptidmolekulargewichten, aber sie wurden entweder nicht für die direkte Anwendung in biotherapeutischen Einrichtungen entwickelt, haben Zugriffsbeschränkungen aufgrund kostenpflichtiger Lizenzen oder erfordern das Hochladen von Proteinsequenzen auf Host-Server.
Wir haben eine modulare Massenberechnungsroutine entwickelt, die die einfache Bestimmung der durchschnittlichen oder monoisotopischen Massen und der elementaren Zusammensetzung von therapeutischen Glykoproteinen ermöglicht, einschließlich monoklonaler Antikörper (mAb), bispezifischer Antikörper (bsAb) und Antikörper-Wirkstoff-Konjugate (ADC). Der modulare Charakter dieses Python-basierten Berechnungsframeworks wird es ermöglichen, diese Plattform in Zukunft auf andere Modalitäten wie Impfstoffe, Fusionsproteine und Oligonukleotide auszuweiten, und dieses Framework könnte auch für die Abfrage von Top-Down-Massenspektrometriedaten nützlich sein. Durch die Erstellung einer eigenständigen Open-Source-Desktop-Anwendung mit einer grafischen Benutzeroberfläche (GUI) hoffen wir, die Einschränkungen bei der Verwendung in Umgebungen zu überwinden, in denen proprietäre Informationen nicht in webbasierte Tools hochgeladen werden können. Dieser Artikel beschreibt die Algorithmen und die Anwendung dieses Werkzeugs, mAbScale, auf verschiedene antikörperbasierte therapeutische Modalitäten.
Introduction
In den letzten zwei Jahrzehnten haben sich Biotherapeutika zu einer tragenden Säule der modernen Pharmaindustrie entwickelt. Die SARS-CoV2-Pandemie und andere lebensbedrohliche Erkrankungen haben den Bedarf an einer schnelleren und breiteren Entwicklung biopharmazeutischer Moleküle weiter erhöht 1,2,3.
Das biotherapeutische Molekulargewicht ist in Kombination mit anderen analytischen Assays entscheidend für die Identifizierung des Moleküls. Die intakten und reduzierten Untereinheitenmassen werden während des gesamten Forschungs- und Entwicklungslebenszyklus als Teil von Kontrollstrategien verwendet, die darauf abzielen, die Qualität aufrechtzuerhalten, wie im QTPP (Quality Target Product Profile)4 beschrieben.
Die analytische Entwicklung in der biopharmazeutischen Industrie stützt sich in hohem Maße auf Massenmessungen für die Analyse intakter Massen und eine tiefgreifende Charakterisierung mittels Peptid-Mapping oder Multi-Attribut-Methode (MAM)-Überwachung. Im Mittelpunkt dieser Techniken, bei denen moderne Massenspektrometrie-Plattformen (MS) zum Einsatz kommen, steht die Fähigkeit, hochauflösende, genaue Massenmessungen (HR/AM) durchzuführen. Die meisten HR/AM-Geräte liefern Massengenauigkeiten im Bereich von 0,5 bis 5 ppm, die mit dem Massenbereich skaliert werden. Die Fähigkeit, Massen für intakte große Moleküle genau zu messen, ermöglicht die schnelle und sichere Identifizierung von Therapeutika mit großen Molekülen. Da die Isotopenauflösung unter typischen experimentellen Bedingungen für große Moleküle (>10 kDa) nicht erreicht werden kann, müssen für den Vergleich und die Identifizierung mittlere Massen berechnet werden 5,6.
Ein typisches Massenspektrum eines intakten oder untergliedrigen Proteins stellt das gesamte Proteoformprofil dar, das zusammengesetzte Informationen über die verschiedenen molekularen Formen enthält, die sich aus posttranslationalen Modifikationen (PTM) und etwaigen primären Strukturunterschieden, wie z. B. Clips oder Sequenzvarianten, ergeben. Die relativ einfache Natur und der hohe Durchsatz dieser Messungen machen sie attraktiv für die Charakterisierung und als In-Prozess-Überwachungskontrollen 7,8. Die Datenanalyse für diese Experimente erfordert in der Regel, dass der Benutzer den Suchraum für molekulare Formen (Bereich von PTMs oder anderen molekularen Formen) definiert. Für glykosylierte Proteine wird dieser Suchraum weitgehend durch die Heterogenität der Glykoformen bestimmt. Kombinationen aus mehreren PTMs, Disulfidbrückenkonfigurationen und anderen Variationen entlang der Primärstruktur machen die Berechnung aller möglichen Molekülformen zu einer mühsamen Aufgabe. Daher ist die manuelle Berechnung der möglichen Molekülformen ein zeit- und ressourcenintensiver Prozess mit einem hohen Potenzial für menschliche Fehler.
Hier stellen wir ein Massenberechnungstool vor, das unter Berücksichtigung der wichtigsten Eigenschaften biotherapeutischer Moleküle, wie z.B. mAbs, bsAbs, ADCs, etc., entwickelt wurde. Das Werkzeug ermöglicht die einfache Einbindung von Suchraumvariablen zur konsistenten Berechnung von Massen und Elementzusammensetzungen. Der modulare Charakter dieses Werkzeugs wird es ermöglichen, es weiterzuentwickeln und auf die Massenberechnung und das Massenanpassungssystem für andere Modalitäten anzuwenden.
Das GUI-Modul ermöglicht es dem Benutzer, die Eingabe für die Massenberechnung anzugeben, wie in Abbildung 1 gezeigt. Konkret gibt der Anwender einbuchstabige Aminosäuresequenzen für leichte und schwere Antikörperketten ein. Gängige Modifikationen für die N-terminale Zyklisierung mit schweren Ketten und das C-terminale Lysin-Clipping sind als Kontrollkästchen enthalten. Ferner kann die chemische Formel/Elementzusammensetzung über das entsprechende Chem Mod-Textfeld zu diesen Proteinketten addiert/subtrahiert werden. Dies gibt dem Benutzer die Flexibilität, eine elementare Zusammensetzung hinzuzufügen, die mehrere posttranslationale Modifikationen oder im Falle eines ADC eine kleinmolekulare Nutzlast enthält. Da die meisten therapeutischen mAbs so konstruiert sind, dass sie die Glykosylierungsstellen in der Leichtkette entfernen, ist die Glykosylierung in der Leichtkette optional und kann über ein Kontrollkästchen in der GUI angegeben werden.
Eine typische Variante der Analyse intakter Massen für Antikörper ist eine Analyse mit reduzierter Untereinheitsmasse, bei der die leichte Kette von der schweren Kette gelöst wird, indem die Disulfidbrücken zwischen den Ketten reduziert werden. Abhängig von der Stärke des verwendeten Reduktionsmittels können die Disulfidbindungen innerhalb der Kette gespalten werden oder nicht. Die Benutzer haben die Flexibilität, die Gesamtzahl der Disulfidbrücken in Abhängigkeit vom IgG-Subtyp oder im Falle eines Cystein-konjugierten ADC9 einzugeben.
Die Anwendung berechnet Massen nach dem Bottom-up-Verfahren, wobei zunächst die elementaren Zusammensetzungen für die einzelnen schweren Ketten und leichten Ketten berechnet werden. Als nächstes wird das Lys-Clipping der Schwerketten-N-terminalen Cyclisierung (HC) durch Anpassung der berechneten Elementzusammensetzungen berücksichtigt. Alle spezifizierten chemischen Modifikationen werden dann auf die schweren und/oder leichten Ketten aufgebracht. Abhängig von der Art der Analyse und den vom Anwender vorgegebenen Disulfidbindungsmustern wird die Anzahl der Wasserstoffe für die beiden Polypeptidketten angepasst. Die glykosylierten HC- und Leichtkettenmassen (LC) (optional) werden auf der Grundlage der Eingaben des Benutzers berechnet. Schließlich werden mehrere HC- und LC-Massen kombiniert, und die Disulfidbrückennummern werden automatisch für die Berechnung der intakten Masse aktualisiert.
Bei größeren Molekülen, wie z.B. intakten Proteinen, können monoisotopische Massen aufgrund des additiven Massendefekts bei der Verwendung von Massenspektrometern mit typischem Auflösungsvermögen nicht gemessen werden. Stattdessen werden Nenn- oder Durchschnittsmassen gemessen oder gemeldet 5,10,11,12,13. Die durchschnittlichen Elementmassen können je nach der Quelle, die für die kuratierten Massen verwendet wird, variieren14,15. Die Unterschiede in den Elementmassen mögen zwar gering sein, können sich aber für die Berechnung des Molekulargewichts großer Moleküle zu signifikanten Werten summieren. Die durchschnittlichen Elementmassen, die standardmäßig in der Softwareanwendung verwendet werden, sind in der ergänzenden Tabelle 1 aufgeführt. Für regulierte Umgebungen wie den Bereich der biopharmazeutischen Forschung und Entwicklung (F&E) ist es wichtig, konsistente Molekülmassen beizubehalten, da Änderungen der Massen Änderungen an der molekularen Einheit während der Zulassungsanträge bedeuten können. Um eine konsistente Verwendung von Elementmassen zu ermöglichen, ist ein Wörterbuch der Elementmassen als CSV-Textdatei (Comma-Separated Value) im Software-Tool enthalten: Element_Mass.csv (Supplementary Coding File 1). In ähnlicher Weise ist eine kuratierte Liste von Glykanzusammensetzungen enthalten, die typischerweise auf mAbs zu sehen sind: Glycan.csv (Supplementary Coding File 2). Beide Dateien werden im selben Ordner wie eine ausführbare Anwendung gespeichert und können vom Benutzer geändert werden, um eine bestimmte Elementmassenliste oder Glykanbibliothek zu verwenden.
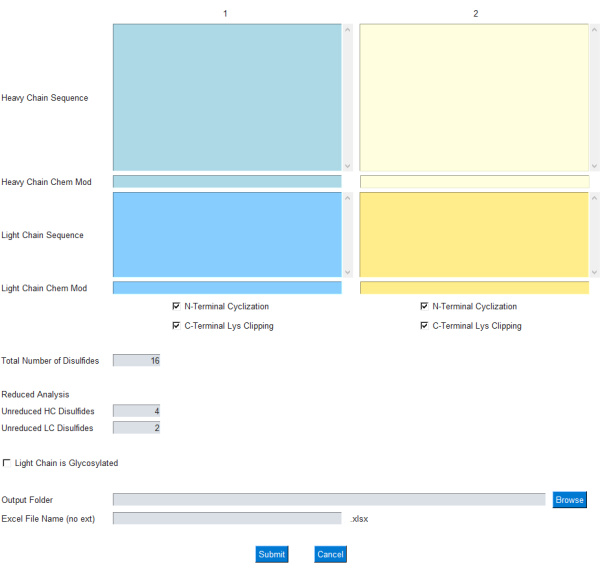
Abbildung 1: GUI-Schnittstelle für die mAbScale-Anwendung. Das GUI-Modul ermöglicht es dem Benutzer, die Eingabe für die Massenberechnung anzugeben. Der Benutzer gibt einbuchstabige Aminosäuresequenzen für die leichten und schweren Antikörperketten ein. Gängige Modifikationen für die N-terminale Zyklisierung mit schweren Ketten und das C-terminale Lysin-Clipping sind als Kontrollkästchen enthalten. Chemische Formeln/Elementzusammensetzungen können über das entsprechende Chem Mod-Textfeld addiert/subtrahiert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protocol
Representative Results
Discussion
mAbScale bietet eine intuitive Benutzeroberfläche mit der Flexibilität, die Bausteine für Massen- und Elementberechnungen zu ändern. Von den Anwendern wird erwartet, dass sie ein grundlegendes Verständnis des Zielmoleküls haben, um die Anwendung zu verwenden, korrekte Massen abzuleiten und die Ergebnisse zu interpretieren. Zum Beispiel kann das intakte oder reduzierte Massenausgabeblatt aufgrund der zahlreichen Reihen intakter oder reduzierter Massen überwältigend sein, da die Standard-Glykandatenbank 88 N-verkn?…
Declarações
The authors have nothing to disclose.
Acknowledgements
Die Autoren danken Robert Schuster für die Unterstützung bei der Datenüberprüfung.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
Referências
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

