Een open-source raamwerk voor massaberekening van op antilichamen gebaseerde therapeutische moleculen
Summary
Dit artikel beschrijft het gebruik van een softwaretoepassing, mAbScale, voor de berekening van massa’s voor op monoklonale antilichamen gebaseerde eiwittherapieën.
Abstract
Biotherapeutische massa’s zijn een middel om identiteit en structurele integriteit te verifiëren. Massaspectrometrie (MS) van intacte eiwitten of eiwitsubeenheden biedt een eenvoudig analytisch hulpmiddel voor verschillende stadia van biofarmaceutische ontwikkeling. De identiteit van het eiwit wordt bevestigd wanneer de experimentele massa van MS binnen een vooraf gedefinieerd massafoutbereik van de theoretische massa ligt. Hoewel er verschillende computationele tools bestaan voor de berekening van eiwit- en peptidemolecuulgewichten, zijn ze ofwel niet ontworpen voor directe toepassing op biotherapeutische entiteiten, hebben ze toegangsbeperkingen als gevolg van betaalde licenties, of vereisen ze het uploaden van eiwitsequenties naar hostservers.
We hebben een modulaire massaberekeningsroutine ontwikkeld die het mogelijk maakt om eenvoudig de gemiddelde of mono-isotopische massa’s en elementaire samenstellingen van therapeutische glycoproteïnen te bepalen, waaronder monoklonale antilichamen (mAb), bispecifieke antilichamen (bsAb) en antilichaam-geneesmiddelconjugaten (ADC). Het modulaire karakter van dit op Python gebaseerde berekeningsraamwerk zal het mogelijk maken om dit platform in de toekomst uit te breiden naar andere modaliteiten zoals vaccins, fusie-eiwitten en oligonucleotiden, en dit raamwerk zou ook nuttig kunnen zijn voor het ondervragen van top-down massaspectrometriegegevens. Door een open-source standalone desktoptoepassing met een grafische gebruikersinterface (GUI) te maken, hopen we de beperkingen rond gebruik in omgevingen te overwinnen waar bedrijfseigen informatie niet kan worden geüpload naar webgebaseerde tools. Dit artikel beschrijft de algoritmen en toepassing van deze tool, mAbScale, op verschillende op antilichamen gebaseerde therapeutische modaliteiten.
Introduction
In de afgelopen twee decennia zijn biotherapeutica geëvolueerd tot een steunpilaar van de moderne farmaceutische industrie. De SARS-CoV2-pandemie en andere levensbedreigende omstandigheden hebben de behoefte aan een snellere en bredere ontwikkeling van biofarmaceutische moleculen verder vergroot 1,2,3.
Het biotherapeutische molecuulgewicht is van cruciaal belang voor de identificatie van het molecuul, in combinatie met andere analytische tests. De intacte en gereduceerde subeenheidmassa’s worden gedurende de hele ontdekkings- en ontwikkelingscyclus gebruikt als onderdeel van controlestrategieën die gericht zijn op het handhaven van de kwaliteit, zoals beschreven in het QTPP (Quality Target Product Profile)4.
Analytische ontwikkeling in de biofarmaceutische industrie is sterk afhankelijk van massametingen voor intacte massa-analyse en diepe karakterisering met behulp van peptidemapping of multi-attribute method (MAM) monitoring. Centraal in deze technieken die gebruik maken van moderne massaspectrometrieplatforms (MS) staat de mogelijkheid om nauwkeurige massametingen (HR/AM) met hoge resolutie te leveren. De meeste HR/AM-instrumenten leveren massanauwkeurigheden in het bereik van 0,5-5 ppm, die schalen met het massabereik. Het vermogen om massa’s nauwkeurig te meten voor intacte grote moleculen maakt de snelle en betrouwbare identificatie van therapieën met grote moleculen mogelijk. Aangezien de isotopenresolutie niet kan worden bereikt onder typische experimentele omstandigheden voor grote moleculen (>10 kDa), moeten de gemiddelde massa’s worden berekend voor vergelijking en identificatie 5,6.
Een typisch intact of subeenheid eiwitmassaspectrum vertegenwoordigt het algemene proteoforme profiel, dat samengestelde informatie bevat over de verschillende moleculaire vormen die het gevolg zijn van posttranslationele modificaties (PTM) en eventuele verschillen in de primaire structuur, zoals clips of sequentievarianten. Het relatief eenvoudige en hoge doorvoerkarakter van deze metingen maakt ze aantrekkelijk voor karakterisering en als controlemechanismen tijdens het proces 7,8. Data-analyse voor deze experimenten vereist meestal dat de gebruiker de zoekruimte voor moleculaire vormen (bereik van PTM’s of andere moleculaire vormen) definieert. Voor geglycosyleerde eiwitten wordt deze zoekruimte grotendeels bepaald door glycoforme heterogeniteit. Combinaties van meerdere PTM’s, disulfidebindingsconfiguraties en andere variaties langs de primaire structuur maken het berekenen van alle mogelijke moleculaire vormen een vervelende taak. Daarom is de handmatige berekening van de mogelijke moleculaire vormen een tijdrovend en tijdrovend proces met een grote kans op menselijke fouten.
Hier presenteren we een massaberekeningstool die is ontwikkeld rekening houdend met de belangrijkste kenmerken van biotherapeutische moleculen, zoals mAbs, bsAbs, ADC’s, enz. De tool maakt het mogelijk om eenvoudig zoekruimtevariabelen op te nemen voor de consistente berekening van massa’s en elementaire samenstellingen. Het modulaire karakter van deze tool zal het mogelijk maken om deze verder te ontwikkelen en toe te passen op massaberekening en massa-matching voor andere modaliteiten.
De GUI-module stelt de gebruiker in staat om de invoer voor de massaberekening te specificeren, zoals weergegeven in figuur 1; In het bijzonder voert de gebruiker aminozuursequenties van één letter in voor lichte en zware antilichaamketens. Veelvoorkomende modificaties voor zware keten N-terminale cyclisatie en C-terminale lysineclipping zijn opgenomen als selectievakjes. Verder kan de chemische formule/elementaire samenstelling worden opgeteld/afgetrokken van deze eiwitketens via het respectievelijke Chem Mod-tekstvak . Dit geeft de gebruiker de flexibiliteit om een elementaire samenstelling toe te voegen die meerdere posttranslationele modificaties of een kleine molecuullading bevat in het geval van een ADC. Aangezien de meeste therapeutische mAbs zijn ontworpen om de glycosyleringsplaatsen in de lichte keten te verwijderen, wordt glycosylering in de lichte keten optioneel gelaten en kan deze worden gespecificeerd met behulp van een selectievakje op de GUI.
Een typische variatie op intacte massa-analyse voor antilichamen is een verminderde subeenheidmassa-analyse, waarbij de lichte keten wordt losgemaakt van de zware keten door de disulfidebindingen tussen de ketens te verminderen. Afhankelijk van de sterkte van het gebruikte reductiemiddel kunnen de disulfidebindingen binnen de keten al dan niet worden gesplitst. De gebruikers hebben de flexibiliteit om het totale aantal disulfidebindingen in te voeren, afhankelijk van het IgG-subtype of in het geval van een cysteïne-geconjugeerde ADC9.
De applicatie berekent massa’s op een bottom-up manier, waarbij eerst de elementaire samenstellingen worden berekend voor de individuele zware ketens en lichte ketens. Vervolgens wordt rekening gehouden met zware keten (HC) N-terminale cyclisatie Lys-clipping door de berekende elementaire samenstellingen aan te passen. Eventuele gespecificeerde chemische modificaties worden vervolgens toegepast op de zware en/of lichte kettingen. Afhankelijk van het type analyse en de door de gebruiker gespecificeerde disulfidebindingspatronen, wordt het aantal waterstofatomen aangepast voor de twee polypeptideketens. De geglycosyleerde HC en lichte keten (LC) (optioneel) massa’s worden berekend op basis van de input van de gebruiker. Ten slotte worden meerdere HC- en LC-massa’s gecombineerd en worden de disulfidebindingsnummers automatisch bijgewerkt voor de berekening van de intacte massa.
Bij grotere moleculen, zoals intacte eiwitten, kunnen mono-isotopische massa’s niet worden gemeten vanwege het additieve massadefect bij gebruik van massaspectrometers met een typisch oplossend vermogen. In plaats daarvan worden nominale of gemiddelde massa’s gemeten of gerapporteerd 5,10,11,12,13. De gemiddelde elementaire massa’s kunnen variëren op basis van de bron die wordt gebruikt voor de samengestelde massa’s14,15. Hoewel de verschillen in elementaire massa’s klein kunnen zijn, kunnen ze oplopen tot significante waarden voor berekeningen van het molecuulgewicht van grote moleculen. De gemiddelde elementaire massa’s die standaard in de softwaretoepassing worden gebruikt, worden weergegeven in aanvullende tabel 1. Voor gereguleerde omgevingen zoals het biofarmaceutische onderzoeks- en ontwikkelingsveld (R&D) is het belangrijk om consistente molecuulmassa’s te behouden, omdat veranderingen in massa’s veranderingen in de moleculaire entiteit kunnen impliceren tijdens regelgevende deponeringen. Om consistentie in het gebruik van elementaire massa’s mogelijk te maken, is een woordenboek van elementaire massa’s bij de softwaretool opgenomen als een door komma’s gescheiden waarde (csv) tekstbestand: Element_Mass.csv (Supplementary Coding File 1). Evenzo is een samengestelde lijst van glycaansamenstellingen opgenomen die typisch zijn voor mAbs: Glycaan.csv (Supplementary Coding File 2). Beide bestanden worden opgeslagen op dezelfde maplocatie als een uitvoerbare toepassing en kunnen door de gebruiker worden gewijzigd om een specifieke elementaire massalijst of glycaanbibliotheek te gebruiken.
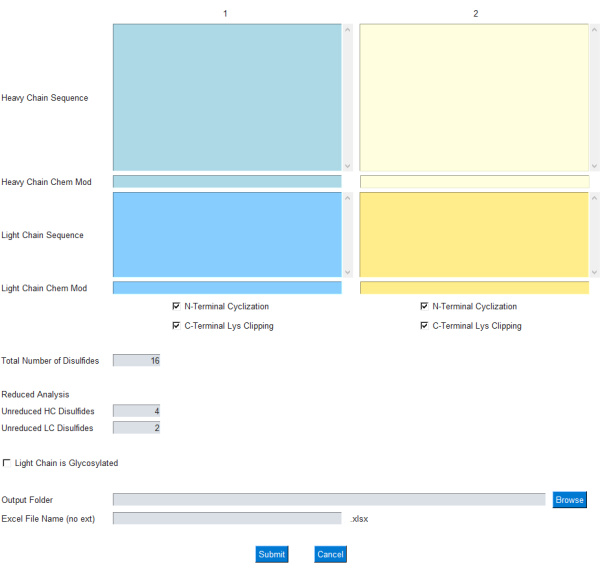
Figuur 1: GUI-interface voor de mAbScale-applicatie. De GUI-module stelt de gebruiker in staat om de invoer voor de massaberekening te specificeren. De gebruiker voert aminozuursequenties van één letter in voor de lichte en zware antilichaamketens. Veelvoorkomende modificaties voor de zware-keten N-terminale cyclisatie en C-terminale lysineclipping zijn opgenomen als selectievakjes. Chemische formules/elementaire samenstellingen kunnen worden opgeteld/afgetrokken via het respectievelijke Chem Mod-tekstvak . Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
mAbScale biedt een intuïtieve gebruikersinterface met de flexibiliteit om de bouwstenen voor massa- en elementaire berekeningen te wijzigen. Van de gebruikers wordt verwacht dat ze een basiskennis hebben van het doelmolecuul om de applicatie te gebruiken, de juiste massa’s af te leiden en de resultaten te interpreteren. Het blad met intacte of gereduceerde massa-output kan bijvoorbeeld overweldigend zijn vanwege de vele rijen intacte of gereduceerde massa’s, aangezien de standaard glycaandatabase 88 N-gebonden glycanen …
Declarações
The authors have nothing to disclose.
Acknowledgements
De auteurs bedanken Robert Schuster voor zijn hulp bij de verificatie van de gegevens.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
Referências
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

