Antikor Bazlı Terapötik Moleküllerin Kütle Hesaplaması için Açık Kaynaklı Bir Çerçeve
Summary
Bu makale, monoklonal antikor bazlı protein terapötikleri için kitlelerin hesaplanması için bir yazılım uygulaması olan mAbScale’in kullanımını açıklamaktadır.
Abstract
Biyoterapötik kitleler, kimliği ve yapısal bütünlüğü doğrulamanın bir yoludur. Bozulmamış proteinlerin veya protein alt birimlerinin kütle spektrometresi (MS), biyofarmasötik gelişimin farklı aşamaları için kolay bir analitik araç sağlar. Proteinin kimliği, MS’den gelen deneysel kütle, teorik kütlenin önceden tanımlanmış bir kütle hatası aralığında olduğunda doğrulanır. Protein ve peptit moleküler ağırlıklarının hesaplanması için çeşitli hesaplama araçları mevcut olsa da, bunlar ya biyoterapötik varlıklara doğrudan uygulama için tasarlanmamıştır, ücretli lisanslar nedeniyle erişim sınırlamalarına sahiptir veya protein dizilerinin ana sunuculara yüklenmesini gerektirir.
Monoklonal antikorlar (mAb), bispesifik antikorlar (bsAb) ve antikor-ilaç konjugatları (ADC) dahil olmak üzere terapötik glikoproteinlerin ortalama veya monoizotopik kütlelerinin ve temel bileşimlerinin kolayca belirlenmesini sağlayan modüler bir kütle hesaplama rutini geliştirdik. Bu Python tabanlı hesaplama çerçevesinin modüler yapısı, bu platformun gelecekte aşılar, füzyon proteinleri ve oligonükleotidler gibi diğer modalitelere genişletilmesine izin verecektir ve bu çerçeve, yukarıdan aşağıya kütle spektrometrisi verilerinin sorgulanması için de yararlı olabilir. Grafik kullanıcı arayüzüne (GUI) sahip açık kaynaklı bağımsız bir masaüstü uygulaması oluşturarak, özel bilgilerin web tabanlı araçlara yüklenemediği ortamlarda kullanımla ilgili kısıtlamaların üstesinden gelmeyi umuyoruz. Bu makale, bu aracın, mAbScale’in algoritmalarını ve farklı antikor bazlı terapötik modalitelere uygulanmasını açıklamaktadır.
Introduction
Son yirmi yılda, biyoterapötikler modern ilaç endüstrisinin temel dayanağı haline geldi. SARS-CoV2 salgını ve yaşamı tehdit eden diğer koşullar, biyofarmasötik moleküllerin daha hızlı ve daha geniş bir şekilde geliştirilmesine olan ihtiyacıdaha da artırmıştır 1,2,3.
Biyoterapötik moleküler ağırlık, diğer analitik tahlillerle kombinasyon halinde molekülün tanımlanması için kritik öneme sahiptir. Sağlam ve azaltılmış alt birim kütleleri, QTPP’de (Kalite Hedef Ürün Profili)4 açıklandığı gibi, kaliteyi korumayı amaçlayan kontrol stratejilerinin bir parçası olarak keşif ve geliştirme yaşam döngüleri boyunca kullanılır.
Biyofarmasötik endüstrisindeki analitik gelişme, büyük ölçüde bozulmamış kütle analizi için kütle ölçümlerine ve peptit haritalama veya çok özellikli yöntem (MAM) izleme kullanılarak derin karakterizasyona dayanır. Modern kütle spektrometresi (MS) platformlarını kullanan bu tekniklerin merkezinde, yüksek çözünürlüklü doğru kütle (HR/) ölçümleri sağlama yeteneği vardır. Çoğu HR/cihazı, kütle aralığına göre ölçeklenen 0,5-5 ppm aralığında kütle doğruluğu sağlar. Bozulmamış büyük moleküller için kütleleri doğru bir şekilde ölçme yeteneği, büyük moleküllü terapötiklerin hızlı ve güvenli bir şekilde tanımlanmasını sağlar. Büyük moleküller için tipik deney koşulları (>10 kDa) kullanılarak izotopik çözünürlük elde edilemediğinden, karşılaştırma ve tanımlamaiçin ortalama kütleler hesaplanmalıdır 5,6.
Tipik bir bozulmamış veya alt birim protein kütle spektrumu, translasyon sonrası modifikasyonlardan (PTM) kaynaklanan çeşitli moleküler formlar ve klipler veya dizi varyantları gibi herhangi bir birincil yapı farklılığı hakkında kompozit bilgiler içeren genel proteoform profilini temsil eder. Bu ölçümlerin nispeten kolay ve yüksek verimli doğası, onları karakterizasyon ve proses içi izleme kontrolleri için çekici kılmaktadır 7,8. Bu deneyler için veri analizi genellikle kullanıcının moleküler formlar (PTM’ler veya diğer moleküler formlar aralığı) için arama alanını tanımlamasını gerektirir. Glikosile edilmiş proteinler için, bu arama alanı büyük ölçüde glikoform heterojenliği tarafından yönlendirilir. Çoklu PTM’lerin, disülfür bağ konfigürasyonlarının ve birincil yapı boyunca diğer varyasyonların kombinasyonları, olası tüm moleküler formların hesaplanmasını sıkıcı bir görev haline getirir. Bu nedenle, olası moleküler formların manuel olarak hesaplanması, insan hatası potansiyeli yüksek, zaman ve kaynak tüketen bir süreçtir.
Burada, mAbs, bsAbs, ADC’ler vb. gibi biyoterapötik moleküllerin en önemli özellikleri göz önünde bulundurularak geliştirilmiş bir kütle hesaplama aracı sunuyoruz. Araç, kütlelerin ve temel bileşimlerin tutarlı bir şekilde hesaplanması için arama alanı değişkenlerinin kolayca birleştirilmesine olanak tanır. Bu aracın modüler yapısı, daha da geliştirilmesini ve diğer modaliteler için kütle hesaplaması ve kütle eşleştirmesine uygulanmasını sağlayacaktır.
GUI modülü, kullanıcının Şekil 1’de gösterildiği gibi kütle hesaplaması için girişi belirlemesine izin verir; Spesifik olarak, kullanıcı hafif ve ağır antikor zincirleri için tek harfli amino asit dizileri girer. Ağır zincirli N-terminal siklizasyonu ve C-terminal lizin kırpma için yaygın değişiklikler onay kutuları olarak dahil edilmiştir. Ayrıca, kimyasal formül/element bileşimi, ilgili Chem Mod metin kutusu aracılığıyla bu protein zincirlerine eklenebilir/çıkarılabilir. Bu, kullanıcıya, bir ADC durumunda birden fazla translasyon sonrası modifikasyon veya küçük moleküllü bir yük içeren bir temel bileşim ekleme esnekliği sağlar. Çoğu terapötik mAb, hafif zincirdeki glikozilasyon bölgelerini uzaklaştırmak için tasarlandığından, hafif zincirdeki glikozilasyon isteğe bağlı bırakılır ve GUI’deki bir onay kutusu kullanılarak belirtilebilir.
Antikorlar için bozulmamış kütle analizinin tipik bir varyasyonu, zincirler arası disülfür bağlarını azaltarak hafif zincirin ağır zincirden ayrıldığı indirgenmiş bir alt birim kütle analizidir. Kullanılan indirgeyici ajanın gücüne bağlı olarak, zincir içi disülfür bağları parçalanabilir veya bölünmeyebilir. Kullanıcılar, IgG alt tipine bağlı olarak veya sistein konjuge ADC9 durumunda toplam disülfür bağı sayısını girme esnekliğine sahiptir.
Uygulama, kütleleri aşağıdan yukarıya doğru hesaplar, burada temel bileşimler ilk olarak bireysel ağır zincirler ve hafif zincirler için hesaplanır. Daha sonra, ağır zincir (HC) N-terminal siklizasyonu Lys-clipping, hesaplanan temel bileşimlerin ayarlanmasıyla hesaba katılır. Belirtilen herhangi bir kimyasal modifikasyon daha sonra ağır ve/veya hafif zincirlere uygulanır. Analizin türüne ve kullanıcı tarafından belirtilen disülfür-bağ modellerine bağlı olarak, iki polipeptit zinciri için hidrojen sayısı ayarlanır. Glikosile HC ve hafif zincir (LC) (isteğe bağlı) kütleleri, kullanıcının girdisine göre hesaplanır. Son olarak, birden fazla HC ve LC kütlesi birleştirilir ve bozulmamış kütle hesaplaması için disülfür bağ numaraları otomatik olarak güncellenir.
Bozulmamış proteinler gibi daha büyük moleküllerde, tipik çözme gücüne sahip kütle spektrometreleri kullanılırken ilave kütle kusuru nedeniyle monoizotopik kütleler ölçülemez. Bunun yerine, nominal veya ortalama kütlelerölçülür veya rapor edilir 5,10,11,12,13. Ortalama temel kütleler, küratörlüğünde kütleler14,15 için kullanılan kaynağa göre değişebilir. Element kütlelerindeki farklılıklar küçük olsa da, büyük moleküllü moleküler ağırlık hesaplamaları için önemli değerler ekleyebilirler. Yazılım uygulamasında varsayılan olarak kullanılan ortalama element kütleleri Ek Tablo 1’de gösterilmektedir. Biyofarmasötik araştırma ve geliştirme (Ar-Ge) alanı gibi düzenlenmiş ortamlar için, tutarlı moleküler kütlelerin korunması önemlidir, çünkü kütlelerdeki değişiklikler, düzenleyici başvurular sırasında moleküler varlıkta değişiklikler anlamına gelebilir. Element kütlelerinin kullanımında tutarlılığı sağlamak için, yazılım aracına virgülle ayrılmış değer (csv) metin dosyası olarak bir element kütleleri sözlüğü eklenmiştir: Element_Mass.csv (Ek Kodlama Dosyası 1). Benzer şekilde, mAb’lerde tipik olarak görülen glikan bileşimlerinin küratörlüğünde bir listesi dahil edilmiştir: Glikan.csv (Ek Kodlama Dosyası 2). Her iki dosya da çalıştırılabilir bir uygulamayla aynı klasör konumuna kaydedilir ve kullanıcı tarafından belirli bir temel kütle listesi veya glikan kitaplığı kullanacak şekilde değiştirilebilir.
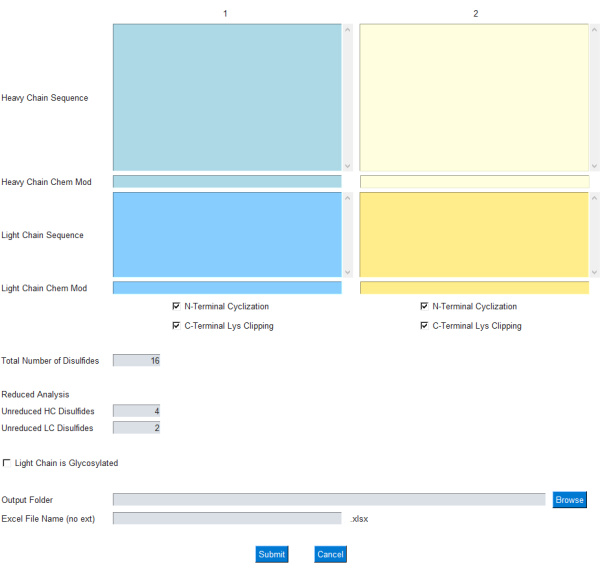
Şekil 1: mAbScale uygulaması için GUI arayüzü. GUI modülü, kullanıcının kütle hesaplaması için girişi belirlemesine izin verir. Kullanıcı, hafif ve ağır antikor zincirleri için tek harfli amino asit dizileri girer. Ağır zincirli N-terminal siklizasyonu ve C-terminal lizin kırpması için yaygın değişiklikler onay kutuları olarak dahil edilmiştir. Kimyasal formüller/temel bileşimler, ilgili Chem Mod metin kutusu aracılığıyla eklenebilir/çıkarılabilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protocol
Representative Results
Discussion
mAbScale, kütle ve element hesaplamaları için yapı taşlarını değiştirme esnekliğine sahip sezgisel bir kullanıcı arayüzü sağlar. Kullanıcıların, uygulamayı kullanmak, doğru kütleleri elde etmek ve sonuçları yorumlamak için hedef molekül hakkında temel bir anlayışa sahip olmaları beklenir. Örneğin, bozulmamış veya azaltılmış kütle çıktı sayfası, çok sayıda bozulmamış veya azaltılmış kütle sırası nedeniyle bunaltıcı olabilir, çünkü varsayılan glikan veritabanı, tera…
Declarações
The authors have nothing to disclose.
Acknowledgements
Yazarlar, veri doğrulama konusundaki yardımı için Robert Schuster’e teşekkür eder.
Materials
| Acquity UPLC system | Waters Corp., Milford, MA | N/A | Modular system |
| Antibody-drug conjugate (ADC) | GlaxoSmithKline | N/A | Proprietory molecule |
| BEH 200 SEC column | Waters Corp., Milford, MA | 176003904 | |
| Bispecific mAb | GlaxoSmithKline | N/A | Proprietory molecule |
| Byos | Protein Metrics, Cupertino, CA | https://proteinmetrics.com/byos/ Version 4.5 |
|
| GPMAW | GPMAW | http://www.gpmaw.com/ | |
| LC-MS grade water | Thermo Fisher Scientific, Waltham, MA | W6-1 | |
| mAb standard | Waters Corp., Milford, MA | 186009125 | Waters Humanized mAb Mass Check Standard |
| mAbScale | GlaxoSmithKline | Apache License, Version 2.0 | |
| Xevo G2 Q-TOF mass spectrometer | Waters Corp., Milford, MA | N/A | Modular system |
Referências
- Reichert, J. M., Valge-Archer, V. E. Development trends for monoclonal antibody cancer therapeutics. Nature Reviews Drug Discovery. 6 (5), 349-356 (2007).
- Kintzing, J. R., Filsinger Interrante, M. V., Cochran, J. R. Emerging strategies for developing next-generation protein therapeutics for cancer treatment. Trends in Pharmacological Sciences. 37 (12), 993-1008 (2016).
- Wang, M. -. Y., et al. SARS-CoV-2: Structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, 587269 (2020).
- ICH Q8 (R2) Pharmaceutical Development – Scientific Guideline. European Medicines Agency Available from: https://www.ema.europa.eu/en/ch-q8-r2-pharmaceutical-development-scientific-guideline (2018)
- Donnelly, D. P., et al. Best practices and benchmarks for intact protein analysis for top-down mass spectrometry. Nature Methods. 16 (7), 587-594 (2019).
- Gadgil, H. S., Pipes, G. D., Dillon, T. M., Treuheit, M. J., Bondarenko, P. V. Improving mass accuracy of high performance liquid chromatography/electrospray ionization time-of-flight mass spectrometry of intact antibodies. Journal of the American Society for Mass Spectrometry. 17 (6), 867-872 (2006).
- Beck, A., Sanglier-Cianférani, S., Van Dorsselaer, A. Biosimilar, biobetter, and next generation antibody characterization by mass spectrometry. Analytical Chemistry. 84 (11), 4637-4646 (2012).
- Camperi, J., Goyon, A., Guillarme, D., Zhang, K., Stella, C. Multi-dimensional LC-MS: the next generation characterization of antibody-based therapeutics by unified online bottom-up, middle-up and intact approaches. Analyst. 146 (3), 747-769 (2021).
- Liu, H., May, K. Disulfide bond structures of IgG molecules. mAbs. 4 (1), 17-23 (2012).
- Jakes, C., Füssl, F., Zaborowska, I., Bones, J. Rapid analysis of biotherapeutics using protein a chromatography coupled to orbitrap mass spectrometry. Analytical Chemistry. 93 (40), 13505-13512 (2021).
- Robotham, A. C., Kelly, J. F., Matte, A. Chapter 1 – LC-MS characterization of antibody-based therapeutics: Recent highlights and future prospects. Approaches to the Purification, Analysis and Characterization of Antibody-Based Therapeutics. , 1-33 (2020).
- Valeja, S. G., et al. Unit mass baseline resolution for an intact 148 kDa therapeutic monoclonal antibody by fourier transform ion cyclotron resonance mass spectrometry. Analytical Chemistry. 83 (22), 8391-8395 (2011).
- Fornelli, L., Ayoub, D., Aizikov, K., Beck, A., Tsybin, Y. O. Middle-down analysis of monoclonal antibodies with electron transfer dissociation orbitrap fourier transform mass spectrometry. Analytical Chemistry. 86 (6), 3005-3012 (2014).
- Berglund, M., Wieser, M. E. Isotopic compositions of the elements 2009 (IUPAC Technical Report). Pure and Applied Chemistry. 83 (2), 397-410 (2011).
- Wang, M., et al. The Ame2012 atomic mass evaluation. Chinese Physics C. 36 (12), 1603-2014 (2012).
- Peri, S., Steen, H., Pandey, A. GPMAW–A software tool for analyzing proteins and peptides. Trends in Biochemical Sciences. 26 (11), 687-689 (2001).
- Tipton, J. D., et al. Analysis of intact protein isoforms by mass spectrometry. The Journal of Biological Chemistry. 286 (29), 25451-25458 (2011).
- De Leoz, M. L. A., et al. interlaboratory study on glycosylation analysis of monoclonal antibodies: Comparison of results from diverse analytical methods. Molecular & Cellular Proteomics. 19 (1), 11-30 (2020).
- Cymer, F., Beck, H., Rohde, A., Reusch, D. Therapeutic monoclonal antibody N-glycosylation – Structure, function and therapeutic potential. Biologicals. 52, 1-11 (2018).
- Baker, P. R., Trinidad, J. C., Chalkley, R. J. Modification site localization scoring integrated into a search engine. Molecular & Cellular Proteomics. 10 (7), (2011).
- Chalkley, R. J., Clauser, K. R. Modification site localization scoring: Strategies and performance. Molecular & Cellular Proteomics. 11 (5), 3-14 (2012).

