RNA Secondaria previsione della struttura utilizzando la forma di alta velocità
Summary
High-throughput selettivo 2 'acilazione idrossile analizzato da primer extension (SHAPE) utilizza un romanzo chemical probing tecnologia, la trascrizione inversa, elettroforesi capillare e software di predizione della struttura secondaria per determinare le strutture di RNA da diverse centinaia a diverse migliaia di nucleotidi a singolo nucleotide risoluzione.
Abstract
Comprendere la funzione di RNA coinvolti in processi biologici richiede una conoscenza approfondita della struttura dell'RNA. A questo fine, la metodologia denominata "2 selettivo high-throughput 'acilazione idrossile analizzato da primer extension", o la forma, permette la previsione di RNA struttura secondaria con risoluzione singolo nucleotide. Questo approccio utilizza agenti chimici che probing acilato preferenzialmente singole regioni o flessibile filamento di RNA in soluzione acquosa. Siti di modificazione chimica vengono rilevati mediante trascrizione inversa del RNA modificati, ed i prodotti di questa reazione sono frazionati mediante elettroforesi capillare automatizzato (CE). Dal momento che la transcriptasi inversa si ferma a quei nucleotidi di RNA modificati dai reagenti SHAPE, la libreria di cDNA risultante indirettamente associa quei ribonucleotidi che sono a singolo filamento nel contesto delle RNA piegati. Utilizzando il software ShapeFinder, i elettroferogrammi prodotti dalla automatizzato CE vengono elaborati e convertiti in nutavoli reattività cleotide che sono a loro volta convertiti in vincoli pseudo-energetiche utilizzate nel RNAStructure (V5.3) algoritmo di predizione. Le strutture di RNA bidimensionali ottenute combinando SHAPE sondando con in silico RNA predizione della struttura secondaria sono stati trovati per essere molto più preciso di strutture ottenute utilizzando il metodo da solo.
Introduction
Per capire le funzioni di RNA catalitici e non codificanti coinvolti nella regolazione dello splicing, la traduzione, la replicazione del virus e il cancro, una conoscenza dettagliata della struttura dell'RNA è richiesto 1,2. Purtroppo, la previsione accurata di RNA folding rappresenta una sfida formidabile. Agenti di sondaggio classici soffrono molti svantaggi come la tossicità, incompleta copertura dei nucleotidi e / o velocità limitata a 100-150 nucleotidi per esperimento. Senza aiuto secondarie algoritmi di predizione di struttura sono allo stesso modo svantaggioso, a causa di imprecisioni derivanti dalla loro incapacità di distinguere efficacemente tra strutture energeticamente simili. Grandi RNA in particolare sono spesso refrattari ai metodi di determinazione della struttura 3D come cristallografia a raggi X e risonanza magnetica nucleare (NMR), grazie alla loro flessibilità conformazionale e grandi quantità di campioni altamente puri richiesti per queste tecniche.
High-capacità SHAPE risolve molti di questi problemi fornendo un efficace, semplice approccio per sondare le strutture di grandi RNA a singolo nucleotide risoluzione. Inoltre, i reagenti utilizzati per la forma sono sicuro, facile da maneggiare e, a differenza della maggior parte dei reagenti chimici probing, reagiscono con tutti e quattro i ribonucleotidi. Questi reagenti possono anche penetrare le membrane cellulari, rendendo possibile sondare RNA nel loro contesto in vivo (s) 3. Originariamente sviluppato nel laboratorio 4 settimane, SHAPE è stato utilizzato per analizzare una grande varietà di RNA, l'esempio più notevole è determinazione della struttura secondaria completa del ~ 9 kb HIV-1 RNA genoma 5. Altri risultati di rilievo utilizzando FORMA includono delucidazione delle strutture di viroidi infettive 6, RNA umani lunghi non codificanti 7, ribosomi lievito 8 e riboswitches 9, nonché per identificare i siti di legame delle proteine nel virione-associata HIV-1 RNA 3. While le varianti originali e high-throughput del protocollo SHAPE sono stati pubblicati altrove 10-12, il presente lavoro fornisce una descrizione dettagliata della determinazione della struttura secondaria dell'RNA da SHAPE high-throughput utilizzando oligonucleotidi fluorescenti, la Beckman Coulter CEQ 8000 Genetic Analyzer, e (v5.3) software SHAPEfinder e RNAStructure. Sono inclusi anche inediti dettagli tecnici e consigli sulla risoluzione dei problemi.
Variazioni di forma
L'essenza di SHAPE e sue variazioni è l'esposizione di RNA in soluzione acquosa a anidridi elettrofili che acilato selettivamente 2'-idrossile (2'-OH) gruppi ribosio, producendo addotti ingombranti nei siti di modificazione. Questa reazione chimica serve come mezzo di interrogare dinamica strutturale RNA locali, come nucleotidi a singolo filamento sono più inclini ad adottare conformazioni favoriscano attacco elettrofilo da questi reagenti, mentre la base accoppiato o architettonico fabbrnucleotidi ained sono meno o non reattivo 10. Siti di formazione di addotti vengono rilevati mediante trascrizione inversa avviando da fluorescente o radioattivo primer ibridati ad un sito specifico sulla RNA modificato (il "(+)" reazione di innesco di estensione). Quando trascrittasi inversa (RT) non riesce ad attraversare i ribonucleotidi acil derivati, un pool di cDNA prodotti viene prodotto il cui lunghezze coincidono con i siti di modificazione. Un controllo, "(-)" primer estensione reazione utilizzando RNA che non è stato esposto a reagente viene eseguita anche in modo che la chiusura prematura della sintesi del DNA (vale a dire "stop") a causa di RNA struttura, non specifico filamento di RNA rotture, ecc, maggio. essere distinta da facendo una pausa prodotto mediante modificazione chimica. Infine, due reazioni di sequenziamento dideossi-avvio dagli stessi primer sono utilizzati come marcatori per correlare nucleotidi reattivi con la sequenza primaria di RNA dopo elettroforesi.
In applicazione originale di SHAPE, lo stesso 32 P-end-marcato primer viene utilizzato per il (+), (-), e due reazioni di sequenziamento. Prodotti di queste reazioni sono caricati in pozzetti adiacenti in un gel di poliacrilammide lastra 5-8%, e frazionato mediante elettroforesi su gel denaturante di poliacrilammide (PAGE; Figura 1). Analisi quantitativa delle immagini gel prodotte da SHAPE convenzionale può essere eseguita utilizzando SAFA, un software semi-automatico di analisi footprinting 13.
In contrasto, la forma ad alta velocità impiega primer fluorescente ed elettroforesi capillare automatizzata. Specificamente, per ogni regione di RNA in esame, una serie di quattro primer DNA avente una sequenza comune ma diverso 5 'etichette fluorescenti devono essere sintetizzati o acquistati. Questi oligonucleotidi diversamente etichettati servono a primi due reazioni forma e le due reazioni di sequenziamento, i cui prodotti sono raggruppati e frazionato / rilevati mediante elettroforesi capillare automatizzato (CE). Whereas il profilo di reattività 100-150 nt di RNA può essere ottenuto da una serie di quattro reazioni usando l'approccio originale, SHAPE alta produttività consente risoluzione del 300-600 nt da un singolo campione riunito 3. Fino a 8 serie di reazioni può essere frazionata simultaneamente, mentre ben 96 campioni possono preparato per frazionamento nel corso di 12 consecutivi piste CE (Figura 2). Inoltre, il software SHAPEfinder, sviluppato per elaborare e analizzare i dati che emergono dal QVC e altri analizzatori genetici, è più automatizzato e richiede meno interventi da parte dell'utente di SAFA 13 o altri pacchetti di gel di analisi.
Metodologie high-throughput più avanzati hanno recentemente emerso come PARS (analisi in parallelo di struttura RNA) 14 e Frag-Seq (frammento-sequencing) 15, che utilizzano enzimi struttura-specifiche, piuttosto che reattivi alchilazione in combinazione con tecniche di sequenziamento di nuova generazione per ottenere information su la struttura dell'RNA. Nonostante l'attrattiva di queste tecniche, le molte limitazioni inerenti alla nucleasi tastatura rimangono ancora 16. Questi problemi possono essere aggirate nel sequenziamento SHAPE (SHAPE-Seq) protocollo 17, dove sequenziamento di nuova generazione è preceduta dalla modificazione chimica e trascrizione inversa di RNA in una maniera simile a quella effettuata per SHAPE convenzionale. Mentre questi metodi possono rappresentare il futuro della determinazione della struttura dell'RNA, è importante ricordare che sequenziamento di nuova generazione è molto costoso, e rimane non disponibile per molti laboratori.
FORMA Data Analysis
Dati prodotti nell'analizzatore genetico è presentato nella forma di un elettroferogramma, in cui l'intensità di fluorescenza del campione (s) che fluisce attraverso il rivelatore capillare è tracciata contro un indice di tempo di migrazione. Questa trama prende la forma di tracce sovrapposte corrispondenti ai quattro canali di fluorescenzas utilizzato per rilevare i diversi fluorofori, e dove ogni traccia è composta da picchi corrispondenti ai singoli prodotti di cDNA o sequenziamento. Dati elettroferogramma vengono esportati dalla analizzatore genetico come file di testo delimitato da tabulazioni e importati in trasformazione ShapeFinder e software di analisi 18.
ShapeFinder è inizialmente utilizzato per eseguire una serie di trasformazioni matematiche sui dati per garantire che i tempi di migrazione e di volumi di picco rispecchiano fedelmente le identità e le quantità dei prodotti di reazione, rispettivamente. Picchi sono poi allineati e integrati, ed i risultati tabulati insieme alla sequenza di RNA primario. Un "profilo reattività" per il segmento pertinente del RNA è ottenuto sottraendo i valori di controllo della (+) valori associati a ogni RNA nucleotide, e normalizzare i dati come descritto di seguito. Questo profilo viene importato in RNAstructure (v5.3) 19,20 software che converte la reattività val normalizzatoUES in vincoli pseudo-energia che sono incorporati nella algoritmo pieghevole struttura secondaria dell'RNA. Combinando chemical probing e piegatura algoritmi in questo modo migliora significativamente l'accuratezza di previsione strutturale rispetto a uno metodo da solo 12,21. L'uscita di RNAstructure (v5.3) comprende le immagini di più bassa energia RNA strutture secondarie con codice colore Con il profilo SHAPE reattività (s), così come le stesse strutture in testuale notazione dot-staffa. Questi ultimi possono essere successivamente esportati in software dedicato alla visualizzazione grafica di RNA struttura secondaria come Varna 22 e PseudoViewer 23.
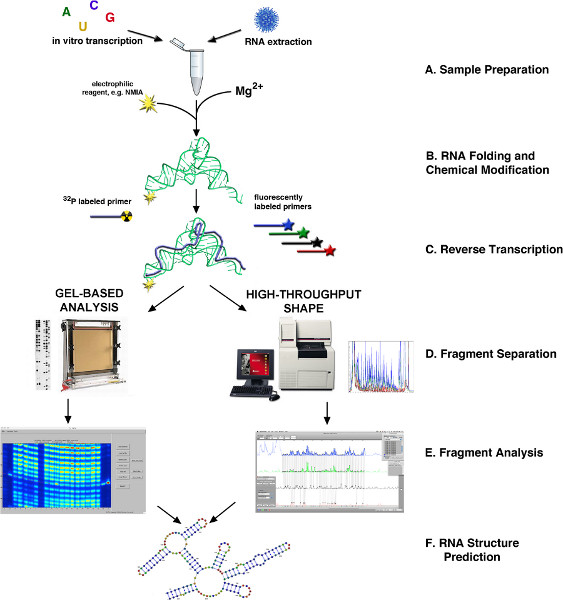
Figura 1. Diagramma di flusso della determinazione della struttura dell'RNA tramite SHAPE 4,10. (A) RNA may essere ottenuto da campioni biologici o di trascrizione in vitro. (B) A seconda della sorgente, l'RNA è piegato o altrimenti elaborati e modificati con il reattivo di SHAPE. (C) trascrizione inversa utilizzando fluorescente o radioattiva primer marcati. (d) i prodotti di cDNA sono frazionato sia tramite elettroforesi su gel a base capillare o lastra. (E) Analisi Fragment. (F) previsione della struttura dell'RNA. Clicca qui per ingrandire la figura.
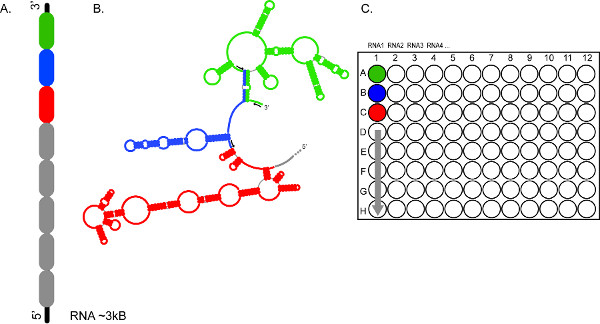
Figura 2. Il carattere high-throughput di SHAPE CE-based permette una rapida analisi di molteplici RNA, e / o più segmenti dello stesso RNA. (A) </strong> Rappresenta come un RNA può essere diviso in sezioni 300-600 nt (codice colore in verde, blu e rosso) (B) Sezioni di RNA vengono sondati in modo indipendente utilizzando diversi set di primer fluorescenti (frecce nere) (C) imposta di Le reazioni sono raggruppati e caricati nei pozzetti A1, B1, C1, ecc, rispettivamente, fornisce una copertura completa per il ~ 3 kb RNA1. Prodotti di reazione di RNA 2, 3, 4, ecc possono essere preparati in modo simile per frazionamento in corse elettroforetiche consecutivi. Clicca qui per ingrandire la figura.
Protocol
Representative Results
Discussion
Presentiamo qui un protocollo dettagliato per la forma ad alta velocità, una tecnica che permette la determinazione della struttura secondaria a risoluzione singolo nucleotide di RNA di qualsiasi dimensione. Inoltre, abbinando i dati di forme sperimentali con secondari algoritmi di predizione di struttura facilita la generazione di modelli di RNA in 2D con un maggior grado di precisione di quanto sia possibile con uno dei due metodi da solo. La combinazione di primer fluorescenza marcata e automatizzato CE offre signif…
Disclosures
The authors have nothing to disclose.
Acknowledgements
S. Lusvarghi, J. Sztuba-Solinska, KJ Purzycka, JW Rausch e SFJ Le Grice sono supportate dal programma di ricerca intramurale del National Cancer Institute, National Institutes of Health, USA.
Materials
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5′ labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied – including any 3’terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies |
References
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 ‘-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 ‘-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2′-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues – A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).

