使用氢交换质谱分析蛋白质动力学
Summary
蛋白质构象和动力学的关键是理解蛋白质结构与功能之间的关系。氢交换耦合用高分辨率质谱法是研究蛋白质的构象动力学以及表征蛋白质 – 配体和蛋白质之间的相互作用,其中包括接触接口和变构效应的灵活的方法。
Abstract
所有的细胞过程依赖于蛋白质的功能。尽管给定的蛋白质的功能是其独特的氨基酸序列的直接后果,它仅由多肽链的折叠成一个单一的定义的三维结构或更常见为构象之间互相的合奏来实现。研究蛋白质构象与功能之间的联系是必不可少的,因此对于蛋白质如何能够履行其种类繁多的任务的完整理解。一种可能性,研究的构象变化,而 通过其官能周期进展的蛋白质经历是氢-1H / 2 H-交换与高分辨率质谱(HX-MS)的组合。 HX-MS是增加了一个新的层面,通过如结晶获得结构信息的灵活和可靠的方法。它是用来研究蛋白质折叠和展开,小摩尔结合ecule配体,蛋白质 – 蛋白质相互作用,挂酶催化的构象变化,和变构效应。此外,HX-MS通常被用于当蛋白质的量是非常有限的或蛋白质的结晶是不可行的。在这里,我们提供了一个通用协议,用于研究蛋白质动力学与HX-MS和描述,例如,如何显示两种蛋白质的相互作用界面在复杂的。
Introduction
蛋白和蛋白复合物的晶体结构的数量近年来迅速增加。他们提出这些蛋白质的结构组织的非常宝贵的快照,并为结构 – 功能分析的基础。然而,蛋白质和构象变化,这对于其功能必需的动力,也很少由X-射线结晶学揭示。冷冻电镜,另一方面,能够捕获蛋白和蛋白复合物在不同的构象,但一般不能解析的构象变化下降到二级结构的第1级。在原子细节的蛋白质在溶液中的构象动力学只能通过NMR来解决,但这种方法仍局限于相对较小的尺寸(通常≤30 kDa)的蛋白,并且需要高浓度的蛋白质(≥100微米),这阻碍了实验低聚或易聚集的蛋白质2。一种方法,该方法能够高分辨率X-射线晶体学和低温电镜和其之间的桥梁不是由蛋白质大小或浓度的限制是酰胺氢-1与质谱(MS)结合的H / 2 H-交换(HX)。近年来,这种方法已经发展到用于蛋白质动力学,蛋白质折叠,蛋白质稳定性和构象变化3-5的分析有价值的分析工具。该方法的分子基础是骨干酰胺氢中的蛋白质,这将与氘原子交换,当蛋白质被放置在D 2 O中的溶液中不稳定的性质。随着时间的推移随后增加蛋白质量的测量与高分辨率质谱。
总之非结构化肽HX只依赖于温度,催化剂浓度(OH – ,H 3 O +,即 pH值, 见图3)和相邻的残基,由于电感,猫氨基酸侧链alytic和空间位阻效应。在固有的化学交换率k CH这些影响已经由白等人 。6被量化的优雅和程序是可用的(礼貌Z.张),其中计算K 通道的多肽取决于pH值和温度范围内每个氨基酸。在中性pH值和温度,Ķch是在10 1 -10 3秒-1的顺序。在折叠的蛋白质HX可以是2-9个数量级慢,主要是由于在二级结构的氢键和一个较小的程度,由于水合OH的受限制的访问–离子以紧密折叠的蛋白质的内部。因此HX的天然蛋白质牵连局部或全局展开,化学交换,并根据公式折叠到天然态(1)和观察到的外汇报价K OBS依赖于开放率k 运算 ,收盘率k cl和内在化学交换RA特ķ 沟道根据等式(2)。
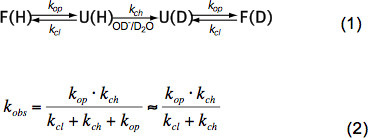
根据天然状态条件Ķ 运算比k 通道小得多,并且可以在分母被忽略。有所谓的EX1和EX2两个极端的汇率制度。如果第k CL比k 通道 (EX1)小得多所观察到的速率几乎等于开口率和HX允许直接观察的结构元件的展开的。这种汇率制度,所有的酰胺质子交换一次开盘时的结构元素,是很容易观察到在MS通过的同位素峰7双峰分布。如果k CL比k 通道大得多(EX2)所观察到的速率正比至K 通道 ,由此比例常数等于该折叠-展开平衡点常数K U = K 运 </sub> / K CL。在这些条件下,许多打开和关闭事件是必要的,在对于氘核所有的酰胺质子交换,导致平均质量逐渐提高,而同位素分布保持大致相同。该EX2制度容许展开ΔGu的自由能的决心和结构元素,因此稳定性。在自然状态条件下的EX2政权是最常见的。增加了pH值和增加离液剂的可以交流机制转移到EX1。因此,HX-MS可以用来探索热力学以及蛋白质折叠和构象变化的动力学参数。
如上面提到的HX是本质的pH和温度依赖的和骨干酰胺基的溶剂完全暴露质子交换半衰期为5-400毫秒,在生理pH值(pH值7.6)和30℃,但10分钟之间,以> 15小时用在pH值2.9,平均> 2小时,0°C(除了一种多肽,它具有交换的半衰期大约为1-2分钟的第一个骨干酰胺键的质子)。在这样慢交换条件,能够使用的蛋白酶( 如胃蛋白酶),这些活性在这些条件下,与出丢失所有包含在掺入氘核的信息来消化样品。自消化道的消化缓慢交换条件下引入,全长蛋白质不仅整体HX动力学进行分析,但HX可以将其定位于特定的区域8,9。空间分辨率目前仅限于所产生的可消化性的片段,这是在一般的10-30残基之间的尺寸。然而,通过胃蛋白酶产生由于裂解的非特异性性质重叠的碎片可能会导致增加的空间分辨率。此外,其它一些蛋白酶被发现淬火条件下有活性,但是,比胃蛋白酶10少得多的效率。进一步increa空间分辨率的本身可以通过肽在气相中的分片到达由保藏的氘化图案如电子捕获解离(ECD),电子转移解离(ETD)和红外多光子解离(IRMPD)11-13的方法。这些技术避免空间分辨率的损失是由于分子内质子迁移(“加扰”),这是由碰撞诱导解离(CID)所观察到的最常用的破碎技术。然而,这些方法需要优化每个个体肽,因而仍然非常具有挑战性。
HX-MS已经被用于分析蛋白质-配体和蛋白质之间的相互作用,包括病毒衣壳组装14-17。蛋白质变性和复性,以及温度诱导的构象变化进行了调查7,18,19。磷酸化和单个氨基酸突变相关的构象变化16,20和nucleotIDE引起的变化进行了分析21,22。因此,这种方法似乎非常适合于分析组件和分子机器的动力学。一个有吸引力的候选人,其机理是非常普遍的兴趣,是Hsp90的分子伴侣复合体。
Protocol
Representative Results
Discussion
一个互动的合作伙伴,一个蛋白质的结合不可避免地导致对结合位点的改变溶剂可及性。此外,许多蛋白进行结合后的动态构象变化,从而影响其他区域比实际的结合界面。 HX-MS是一种可靠的方法来监测这些变化,甚至能够在时间尺度是其他方法不能覆盖揭示蛋白质构象变化的。
为了成功执行HX-MS三点是关键:1)一个最佳的系统设置; 2)指定肽谱和解释结果时,精确的执行?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
我们感谢M.百胜对稿件的意见。该项目是由德意志研究联合会(SFB638和MA 1278/4-1到MPM,与集群卓越:CellNetworks EXC一分之八十一)。 MPM是卓越的集群调查:CellNetworks。
Materials
| Reagent/Equipment | Company | Catalogue number | Comments (optional) |
| maXis QTOF | Bruker | ||
| nanoAcquity UPLC | Waters Corp. | ||
| Shimadzu 10AD-VP | Shimadzu | ||
| 6-port Valve EPC6W with microelectric actuator | Valco | #EPC6W | |
| Injection valve (manual) | Rheodyne | #7725 | |
| Poros AL20 media | Applied Biosystems | #1-6029-06 | |
| Poros R2 | Applied Biosystems | #1-1118-02 | |
| Pepsin | Sigma | #P6887 | use fresh pepsin |
| Microbore (1 mm) | IDEX | #C-128 | |
| Microbore (2 mm) | IDEX | #C-130B | |
| Acquity UPLC BEH C8 Column | Waters Corp. | #186002876 | |
| Thermomixer | Eppendorf | #5355000.011 | |
| Tubing (various diameters) | IDEX | ||
| Fittings | IDEX | #PK-110 with PK-100 |
References
- Saibil, H. R. Conformational changes studied by cryo-electron microscopy. Nat. Struct. Biol. 7 (9), 711-714 (2000).
- Mittermaier, A., Kay, L. E. New tools provide new insights in NMR studies of protein dynamics. Science. 312 (5771), 224-228 (2006).
- Hoofnagle, A. N., Resing, K. A., Ahn, N. G. Protein analysis by hydrogen exchange mass spectrometry. Annu. Rev. Biophys. Biomol. Struct. 32, 1-25 (2003).
- Wales, T. E., Engen, J. R. Hydrogen exchange mass spectrometry for the analysis of protein dynamics. Mass. Spectrom. Rev. 25 (1), 158-170 (2006).
- Konermann, L., Pan, J., Liu, Y. -. H. Hydrogen exchange mass spectrometry for studying protein structure and dynamics. Chem Soc. Rev. 40 (3), 1224-1210 (2011).
- Bai, Y., Milne, J. S., Mayne, L., Englander, S. W. Primary structure effects on peptide group hydrogen exchange. Proteins. 17 (1), 75-86 (1993).
- Rist, W., Jørgensen, T. J. D., Roepstorff, P., Bukau, B., Mayer, M. P. Mapping temperature-induced conformational changes in the Escherichia coli heat shock transcription factor sigma 32 by amide hydrogen exchange. The Journal of biological chemistry. 278 (51), 51415-51421 (1074).
- Englander, J. J., Rogero, J. R., Englander, S. W. Protein hydrogen exchange studied by the fragment separation method. Anal Biochem. 147 (1), 234-244 (1985).
- Zhang, Z., Smith, D. L. Determination of amide hydrogen exchange by mass spectrometry: a new tool for protein structure elucidation. Protein Sci. 2 (4), 522-531 (1993).
- Cravello, L., Lascoux, D., Forest, E. Use of different proteases working in acidic conditions to improve sequence coverage and resolution in hydrogen/deuterium exchange of large proteins. Rapid Commun. Mass Spectrom. : RCM. 17 (21), 2387-2393 (2003).
- Rand, K. D., Zehl, M., Jensen, O. N., Jørgensen, T. J. D. Protein hydrogen exchange measured at single-residue resolution by electron transfer dissociation mass spectrometry. Anal chem. 81 (14), 5577-5584 (2009).
- Pan, J., Han, J., Borchers, C. H., Konermann, L. Electron capture dissociation of electrosprayed protein ions for spatially resolved hydrogen exchange measurements. J. Am. Chem. Soc. 130 (35), 11574-11575 (2008).
- Yamada, N., Suzuki, E. -. I., Hirayama, K. Identification of the interface of a large protein-protein complex using H/D exchange and Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 16 (4), 293-299 (2002).
- Lee, T., Hoofnagle, A. N., et al. Docking motif interactions in MAP kinases revealed by hydrogen exchange mass spectrometry. Mol. Cell. 14 (1), 43-55 (2004).
- Hasan, A., Smith, D. L., Smith, J. B. Alpha-crystallin regions affected by adenosine 5′-triphosphate identified by hydrogen-deuterium exchange. Biochem. 41 (52), 15876-15882 (2002).
- Lanman, J., Lam, T. T., Emmett, M. R., Marshall, A. G., Sakalian, M., Prevelige, P. E. Key interactions in HIV-1 maturation identified by hydrogen-deuterium exchange. Nat. Struct. Mol. Biol. 11 (7), 676-677 (2004).
- Wang, L., Lane, L. C., Smith, D. L. Detecting structural changes in viral capsids by hydrogen exchange and mass spectrometry. Protein Sci. 10 (6), 1234-1243 (2001).
- Pan, H., Raza, A. S., Smith, D. L. Equilibrium and kinetic folding of rabbit muscle triosephosphate isomerase by hydrogen exchange mass spectrometry. J. Mol. Biol. 336 (5), 1251-1263 (2004).
- Mazon, H., Marcillat, O., Forest, E., Smith, D. L., Vial, C. Conformational dynamics of the GdmHCl-induced molten globule state of creatine kinase monitored by hydrogen exchange and mass spectrometry. Biochem. 43 (17), 5045-5054 (2004).
- Lanman, J., Lam, T. T., et al. Identification of novel interactions in HIV-1 capsid protein assembly by high-resolution mass spectrometry. J. Mol. Biol. 325 (4), 759-772 (2003).
- Rist, W., Graf, C., Bukau, B., Mayer, M. P. Amide hydrogen exchange reveals conformational changes in hsp70 chaperones important for allosteric regulation. J. Biol. chem. 281 (24), 16493-16501 (2006).
- Graf, C., Stankiewicz, M., Kramer, G., Mayer, M. P. Spatially and kinetically resolved changes in the conformational dynamics of the Hsp90 chaperone machine. EMBO J. 28 (5), 602-613 (2009).
- Lee, C. -. T., Graf, C., Mayer, F. J., Richter, S. M., Mayer, M. P. Dynamics of the regulation of Hsp90 by the co-chaperone Sti1. EMBO J. 31 (6), 1518-1528 (2012).
- Lou, X., Kirchner, M., et al. Deuteration distribution estimation with improved sequence coverage for HX/MS experiments. Bioinformatics. 26 (12), 1535-1541 (2010).
- Kreshuk, A., Stankiewicz, M., Lou, X., Kirchner, M., Hamprecht, F. A., Mayer, M. P. Automated detection and analysis of bimodal isotope peak distributions in H/D exchange mass spectrometry using HeXicon. Intl. J. mass spectrom. 302 (1-3), 125-131 (2011).
- Walters, B. T., Ricciuti, A., Mayne, L., Englander, S. W. Minimizing back exchange in the hydrogen exchange-mass spectrometry experiment. J. Am. Soc. Mass Spectrom. 23 (12), 2132-2139 (2012).
- Weis, D. D., Wales, T. E., Engen, J. R., Hotchko, M., Ten Eyck, L. F. Identification and characterization of EX1 kinetics in H/D exchange mass spectrometry by peak width analysis. J. Am. Soc. Mass Spectrom. 17 (11), 1498-1509 (2006).
- Fang, J., Rand, K. D., Beuning, P. J., Engen, J. R. False EX1 signatures caused by sample carryover during HX MS analyses. Intl. J. Mass Spectrom. 302 (1-3), 19-25 (2011).
- Deng, Y., Smith, D. L. Identification of unfolding domains in large proteins by their unfolding rates. Biochem. 37 (18), 6256-6262 (1998).
- Wales, T. E., Engen, J. R. Partial unfolding of diverse SH3 domains on a wide timescale. J. Mol. Biol. 357 (5), 1592-1604 (2006).
- Pan, Y., Piyadasa, H., O’Neil, J. D., Konermann, L. Conformational dynamics of a membrane transport protein probed by h/d exchange and covalent labeling: the glycerol facilitator. J. Mol. Biol. 416 (3), 400-413 (2012).
- Hebling, C. M., Morgan, C. R., Stafford, D. W., Jorgenson, J. W., Rand, K. D., Engen, J. R. Conformational analysis of membrane proteins in phospholipid bilayer nanodiscs by hydrogen exchange mass spectrometry. Anal chem. 82 (13), 5415-5419 (2010).
- Abzalimov, R. R., Kaplan, D. A., Easterling, M. L., Kaltashov, I. A. Protein conformations can be probed in top-down HDX MS experiments utilizing electron transfer dissociation of protein ions without hydrogen scrambling. J. Am. Soc. Mass Spectrom. 20 (8), 1514-1517 (2009).

