Karakterisering af komplekse systemer ved hjælp af udformning af eksperimenter Fremgangsmåde: Transient Protein Expression i tobak som et casestudie
Summary
Vi beskriver en udformning af eksperimenter tilgang, der kan anvendes til at bestemme og modellere indflydelse transgene regulatoriske elementer, parametre planters vækst og udvikling og inkuberingsbetingelser på forbigående ekspression af monoklonale antistoffer og reporter-proteiner i planter.
Abstract
Planter giver flere fordele for produktionen af biofarmaceutiske herunder lave omkostninger, skalerbarhed og sikkerhed. Forbigående udtryk giver den yderligere fordel af korte udviklings-og produktionstider, men udtryk niveauer kan variere betydeligt mellem partier og dermed give anledning til samme reguleringsmæssige problemer i forbindelse med god fremstillingspraksis. Vi brugte et design af eksperimenter (DOE) tilgang til at fastslå virkningen af væsentlige faktorer, såsom regulatoriske elementer i ekspressionskonstruktet, plantevækst og udvikling parametre og inkubationsbetingelserne under udtryk, om variabilitet i udtryk mellem partier. Vi testede planter, der udtrykker en model anti-HIV monoklonale antistof (2G12) og en fluorescerende markør protein (DsRed). Vi diskuterer begrundelsen for at vælge bestemte egenskaber af model og identificere dens potentielle begrænsninger. Den generelle indstilling kan nemt overføres til andre problemer, fordi principperne i modellen enre bredt anvendelig: videnbaseret parameter udvælgelse, kompleksitet reduktion ved at opdele det oprindelige problem i mindre moduler, software-guidet opsætning af optimale eksperimentkombinationer og trinvis design augmentation. Derfor er den metode er ikke kun nyttig til at karakterisere protein ekspression i planter, men også til undersøgelse af andre komplekse systemer, der mangler en mekanistisk beskrivelse. De prædiktive ligninger, der beskriver sammenkoblingen mellem parametre kan bruges til at etablere mekanistiske modeller for andre komplekse systemer.
Introduction
Produktionen af biofarmaceutiske proteiner i planter er fordelagtig, fordi planter er billig at vokse, kan platformen blive skaleret op blot ved at dyrke flere planter og humane patogener er i stand til at replikere 1,2. Transiente ekspressionssystemer strategier baseret for eksempel på infiltration af blade med Agrobacterium tumefaciens giver yderligere fordele, fordi tiden mellem det punkt af DNA levering og levering af et renset produkt nedsættes fra år til mindre end 2 måneder 3. Transient ekspression er også anvendes til funktionel analyse, fx at teste gener for deres evne til at komplementere tab af funktion mutanter eller for at undersøge protein-interaktioner 4-6. Men forbigående udtryk niveauer tendens til at vise større batch-til-batch variation end ekspressionsniveauer i transgene planter 7-9. Dette reducerer sandsynligheden for, at biofarmaceutiske fremstillingsprocesser baseret på forbigående ekspression will blive godkendt i forbindelse med god fremstillingspraksis (GMP), fordi reproducerbarhed er en attribut kritisk kvalitet og er underlagt risikovurdering 10. Sådanne variationer kan også maskere enhver interaktion, forskerne har til hensigt at undersøge. Derfor er vi i gang med at identificere de væsentligste faktorer, der påvirker forbigående ekspressionsniveauer i planter og til at opbygge en høj kvalitet kvantitativ prædiktiv model.
Den ene-faktor-på-en-tid (OFAT) metode bruges ofte til at karakterisere effekten (virkning) for visse parametre (faktorer) om resultatet (respons) af et eksperiment 11. Men det er ikke optimalt, fordi de enkelte tests (kører) under en kontrolundersøgelse (eksperiment) vil blive rettet ind som perler på en snor gennem det potentielle areal udspændt af de faktorer, der er testet (design plads). Dækningen af designet rum og dermed graden af information udledt fra eksperimentetlav, som det er vist i figur 1A 12. Desuden kan den indbyrdes afhængighed mellem de forskellige faktorer (faktor interaktioner) forbliver skjult resulterer i dårlige modeller og / eller forudsigelse af falsk optima, som vist i figur 1B 13.
De ovenfor beskrevne ulemper kan undgås ved hjælp af et design af eksperimenter (DOE) tilgang, hvor kørsler af et eksperiment er spredt mere jævnt over hele design plads, hvilket betyder, at mere end én faktor er varieret mellem to kørsler 14. Der er specialiserede designs for blandinger, screening faktorer (Faktorforsøg) og kvantificering af faktor virkninger på reaktioner (respons overflade metoder RSM s) 15. Desuden kan RSMs realiseres som centrale-komposit design, men kan også opnås effektivt ved hjælp af specialiseret software, som kan anvende forskellige kriterier for udvælgelse af kørsler. For eksempel, den såkaldte D-optimality kriterium vælger løber så at minimere fejlen i koefficienterne af den resulterende model, mens IV-optimalitet kriterium vælger kørsler, der opnår den laveste forudsigelse varians i hele design plads 15,16. RSM vi beskriver her tillader præcis kvantificering af forbigående protein ekspression i planter, men det kan nemt overføres til en hvilken som helst system, der involverer flere (~ 5-8) numeriske faktorer (fx. temperatur, tid, koncentration), og et par (~ 2 – 4) kategorisk faktorer (fx promotor, farve), i hvilken en mekanistisk beskrivelse er utilgængelig eller for komplekse til at modellere.
DOE tilgang opstod i jordbrugsvidenskab, men har spredt sig til andre områder, fordi det kan overføres til en hvilken som helst situation, hvor det er nyttigt at reducere antallet af kørsler er nødvendige for at opnå pålidelige data og generere beskrivende modeller for komplekse processer. Dette har igen ført til inddragelse af DoE i "Vejledning forIndustri, Q8 (R2) Farmaceutisk udvikling "udgivet af Den Internationale Konference om Harmonisering af tekniske krav til registrering af lægemidler til mennesker (ICH) 17. DoE er nu anvendes bredt i videnskabelig forskning og industri 18 år. Dog skal man være omhyggelig under planlægning og udførelse af forsøget, fordi du vælger en forkert polynomium for flere lineær regressionsmodel (basismodel) kan introducere et behov for yderligere kørsler til at modellere alle faktor virkninger korrekt. Desuden beskadiget eller manglende data genererer forkerte modeller og fejlbehæftet forudsigelser, og kan endda forhindre enhver modelbygning forsøg som beskrevet i protokollen og diskussion § 18. i protokollen afsnit vil vi oprindeligt fastsat de vigtigste planlægning skridt til et RSM-baserede eksperiment og derefter forklare design baseret på DoE software DesignExpert v8.1. Men lignende design kan bygges med anden software including JMP, MODDE og STATISTISK. De eksperimentelle procedurer følges af instruktioner til dataanalyse og evaluering.
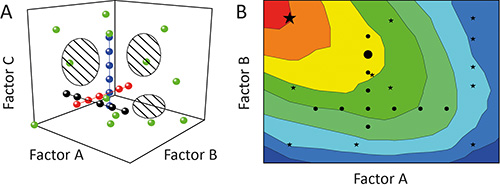
Figur 1. Sammenligning af OFAT og DoE. A. Sekventiel variation af én faktor ad gangen (OFAT) i et eksperiment (sort, rød og blå cirkler) opnår en lav dækning af designet rum (skraverede områder). I modsætning hertil variation af mere end én faktor ad gangen ved hjælp af udformning af eksperimenter (DOE) strategi (grønne cirkler) forbedrer dækning og dermed præcisionen af de resulterende modeller. B. Den forudindtaget design plads dækning betyder, at OFAT eksperimenter (sorte cirkler) kan også undlade at identificere optimale drifts-regioner (rød) og forudsige sub-optimale løsninger (stor sort cirkel), mens DoE strategyes (sorte stjerner) er mere tilbøjelige til at identificere foretrukne forhold (store sorte stjerne).
Protocol
Representative Results
Discussion
Hvert eksperiment kræver omhyggelig planlægning, fordi ressourcerne er ofte knappe og dyre. Dette gælder især for ministeriets strategier, fordi fejl i planlægningsfasen (fx vælge en base model, der ikke dækker alle væsentlige faktor interaktioner) væsentligt kan formindske den prædiktive effekt af de resulterende modeller, og dermed devaluere hele eksperimentet. Dog kan disse fejl let undgås ved at følge grundlæggende procedurer.
Overvejelser i løbet DoE planlægning </p…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Forfatterne er taknemmelige til Dr. Thomas Rademacher for at levere ppam planteekspressionsvektor og Ibrahim Al Amedi til dyrkning af tobaksplanter, der anvendes i denne undersøgelse. Vi vil gerne takke Dr. Richard M. Twyman for hans hjælp med at redigere manuskriptet. Dette arbejde blev delvist finansieret af Det Europæiske Forskningsråd Advanced Grant "Future-Pharma", forslag nummer 269.110 og Fraunhofer Zukunftsstiftung (Fraunhofer Future Foundation).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

