Karakterisering av komplexa system Använda Försöks Tillvägagångssätt: Transient Protein Expression i tobak som en fallstudie
Summary
Vi beskriver en design av experiment tillvägagångssätt som kan användas för att bestämma och modellera inverkan av transgen regulatoriska element, tillväxt och utveckling parametrar växt och inkubationsbetingelser för transient expression av monoklonala antikroppar och reporter proteiner i växter.
Abstract
Växter ger flera fördelar för produktion av biologiska läkemedel, inklusive låga kostnader, skalbarhet och säkerhet. Övergående uttryck ger den ytterligare fördelen med korta utvecklings-och produktionstider, men uttrycksnivåer kan variera kraftigt mellan partier vilket ger upphov till regleringsproblem i samband med god tillverkningssed. Vi använde en försöks (DoE) metod för att bedöma effekten av viktiga faktorer såsom reglerande element i expressionskonstruktet, växternas tillväxt och utveckling parametrar och inkubationsbetingelserna under uttryck, om variationen i uttryck mellan partier. Vi testade växter som uttrycker en modell anti-HIV-monoklonal antikropp (2G12), och ett fluorescerande markörprotein (DsRed). Vi diskuterar den logiska grunden för att välja vissa egenskaper hos den modell och identifiera dess potentiella begränsningar. Kan enkelt överföras till andra problem Den allmänna inriktningen, eftersom principerna i modellen enre bred tillämpning: kunskaps parameterval, komplexitet minskning genom att dela upp det ursprungliga problemet i mindre moduler, program-guidad installation av optimala experimentkombinationer och stegvis konstruktion augmentation. Därför är inte bara användbar för att karakterisera proteinuttryck i växter utan också för undersökning av andra komplexa system som saknar en mekanistisk beskrivning av metodiken. De prediktiva ekvationer som beskriver sammankoppling parametrar kan användas för att etablera mekanistiska modeller för andra komplexa system.
Introduction
Produktionen av biofarmaceutiska proteiner i växter är fördelaktig eftersom växter är billig att växa, kan plattformen skalas upp bara genom att växa mer växter, och humana patogener är oförmögna att replikera 1,2. Transienta expressionsstrategier baserade till exempel på infiltration av blad med Agrobacterium tumefaciens ger ytterligare fördelar eftersom tiden mellan punkten för DNA-leverans och leverans av en renad produkt reduceras från år till mindre än två månader 3. Transient uttryck används också för funktionell analys, t.ex. för att testa gener för sin förmåga att komplettera förlust av funktion mutanter eller för att undersöka protein-interaktioner 4-6. Emellertid transienta expressionsnivåer tenderar att visa större batch-to-batch variation än expressionsnivåer i transgena växter 7-9. Detta minskar sannolikheten för att biofarmaceutiska tillverkningsprocesser baserade på transient expression will godkännas inom ramen för god tillverkningssed (GMP) eftersom reproducerbarhet är en kritisk attribut kvalitet och är föremål för riskbedömning 10. Denna variation kan också maskera eventuella interaktioner som forskare har för avsikt att undersöka. Därför satte vi ut för att identifiera de viktigaste faktorerna som påverkar övergående uttrycksnivåer hos växter och att bygga en högkvalitativ kvantitativ prediktiv modell.
Den en-faktor-på-en-gång (OFAT) metod används ofta för att karakterisera effekten (effekt) av vissa parametrar (faktorer) om resultatet (respons) i ett experiment 11. Men detta är suboptimal eftersom de enskilda tester (kör) under en undersökning (experiment) kommer att anpassas som pärlor på ett snöre genom det potentiella området sträckte sig från de faktorer som testas (design utrymme). Täckningen av design utrymme och därmed graden av information från försöket ärlåg, såsom visas i figur 1 A 12. Dessutom kan ömsesidiga sambandet mellan olika faktorer (faktor interaktioner) förblir dold resulterar i dåliga modeller och / eller förutsägelse av falsk optima, såsom visas i Figur 1B 13.
De ovan beskrivna nackdelarna kan undvikas genom användning av en design av experiment (DOE) strategi, där körningar av ett experiment är utspridda jämnare över hela designutrymmet, vilket innebär att mer än en faktor varieras mellan två körningar 14. Det finns specialiserade designer för blandningar, screening faktorer (faktorförsök) och kvantifiering av faktor påverkan på svaren (metoder svars yta, RSM s) 15. Dessutom kan RSMS realiseras som central-komposit konstruktioner men kan även ske med hjälp av specialiserad programvara som kan använda olika kriterier för urval av körningar. Till exempel, det så kallade D-optimality kriterium väljer körningar så att minimera felet i koefficienterna den resulterande modellen, medan IV-optima kriteriet väljer körningar som uppnår den lägsta förutsägelse variansen i hela designen utrymmet 15,16. RSM vi beskriver här låter exakt kvantifiering av övergående proteinuttryck i växter, men det kan lätt överföras till något system med flera (~ 5-8) numeriska faktorer (t.ex. temperatur, tid, koncentration) och några (~ 2 – 4) kategoriska faktorer (t.ex. promotor, färg), i vilken en mekanistisk beskrivning är tillgänglig eller är alltför komplicerade för att modellera.
DOE strategi har sitt ursprung i de areella näringarna, men har spridit sig till andra områden, eftersom det är överförbart till alla situationer där det är lämpligt att minska antalet körningar som krävs för att få tillförlitliga data och generera beskrivande modeller för komplexa processer. Detta i sin tur har lett till införandet av DoE i "Vägledning förIndustri, Q8 (R2) Pharmaceutical Development "som publiceras av den internationella konferensen om harmonisering av tekniska krav för registrering av humanläkemedel (ICH) 17. DoE används nu i stor utsträckning i vetenskaplig forskning och industri 18. Dock måste man vara försiktig vid planering och genomförande av experimentet, eftersom du väljer en felaktig polynom grad för multipel-linjär regressionsmodell (basmodellen) kan införa ett behov av ytterligare körningar för att modellera alla faktoreffekter på rätt sätt. Dessutom skadade eller saknade uppgifter genererar felaktiga modeller och bristfällig förutsägelser, och kan till och med förhindra modellbygge försök som beskrivs i protokollet och diskussionssektioner 18. I protokollet avsnitt kommer vi inledningsvis anges de viktigaste planeringssteg för en RSM-baserade experiment och sedan förklara design baserad på DoE programvara DesignExpert v8.1. Men liknande konstruktioner kan byggas med annan programvara including JMP, Modde och STATISTICA. De experimentella procedurer följs av anvisningar för dataanalys och utvärdering.
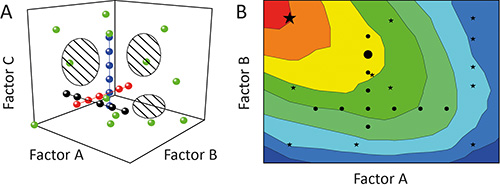
Figur 1. Jämförelse av OFAT och DoE. A. Sekventiell variant av en faktor i taget (OFAT) i ett experiment (svarta, röda och blå cirklar) uppnår en låg täckning av design utrymme (skuggade områden). Däremot variationen av mer än en faktor i taget med hjälp av försöksplanering (DoE) strategi (gröna cirklar) förbättrar täckningen och därmed precisionen i de resulterande modellerna. B. Den partisk design utrymme täckning innebär att OFAT experiment (svarta cirklar) också kan misslyckas med att identifiera optimala driftsområden (röd) och förutsäga suboptimala lösningar (stor svart cirkel), medan DoE strategisktes (svarta stjärnor) är mer benägna att identifiera föredra förhållanden (stor svart stjärna).
Protocol
Representative Results
Discussion
Varje experiment kräver noggrann planering eftersom resurserna är ofta knappa och dyra. Detta gäller särskilt för DoE strategier eftersom fel under planeringsfasen (t.ex. välja en basmodell som inte täcker alla viktiga faktor interaktioner) avsevärt kan minska den prediktiva kraften i de resulterande modellerna och därmed devalvera hela experimentet. Dock kan dessa fel lätt undvikas genom att följa grundläggande förfaranden.
Överväganden under DoE planering
<p class=…Disclosures
The authors have nothing to disclose.
Acknowledgements
Författarna är tacksamma till Dr Thomas Rademacher för att tillhandahålla ppam växtexpressionsvektor och Ibrahim Al Amedi för odling av tobaksplantor som används i denna studie. Vi vill tacka Dr Richard M. Twyman för hans hjälp med att redigera manuskriptet. Detta arbete har delvis finansierats av Europeiska forskningsrådet Advanced Grant "Future-Pharma", förslagsnummer 269.110 och Fraunhofer Zukunftsstiftung (Fraunhofer Future Foundation).
Materials
| Design-Expert(R) 8 | Stat-Ease, Inc. | n.a. | DoE software |
| Tryptone | Carl Roth GmbH | 8952.2 | Media component |
| Yeast extract | Carl Roth GmbH | 2363.2 | Media component |
| Sodium chloride | Carl Roth GmbH | P029.2 | Media component |
| Ampicillin | Carl Roth GmbH | K029.2 | Antibiotic |
| Agar-Agar | Carl Roth GmbH | 5210.2 | Media component |
| Escherichia coli K12 DH5a | Life technologies | 18263-012 | Microorganism |
| pPAM | GenBank | AY027531 | Cloning/expression vector; |
| NucleoSpin Plasmid | MACHEREY-NAGEL GmbH | 740588.250 | Plasmid DNA isolation kit |
| NucleoSpin Gel and PCR Clean-up | MACHEREY-NAGEL GmbH | 740609.250 | Plasmid DNA purification kit |
| NanoDrop 2000 | Thermo Scientific | n.a. | Spectrophotometer |
| NcoI | New England Biolabs Inc. | R3193L | Restrictionendonuclease |
| EcoRI | New England Biolabs Inc. | R3101L | Restrictionendonuclease |
| AscI | New England Biolabs Inc. | R0558L | Restrictionendonuclease |
| NEB 4 | New England Biolabs Inc. | B7004S | Restrictionendonuclease buffer |
| TRIS | Carl Roth GmbH | 4855.3 | Media component |
| Disodium tetraborate | Carl Roth GmbH | 4403.3 | Media component |
| EDTA | Carl Roth GmbH | 8040.2 | Media component |
| Agarose | Carl Roth GmbH | 6352.4 | Media component |
| Bromophenol blue | Carl Roth GmbH | A512.1 | Color indicator |
| Xylene cyanol | Carl Roth GmbH | A513.1 | Color indicator |
| Glycerol | Carl Roth GmbH | 7530.2 | Media component |
| Mini-Sub Cell GT Cell | BioRad | 170-4406 | Gel electrophoresis chamber |
| Agrobacterium tumefaciens strain GV3101:pMP90RK | DSMZ | 12365 | Microorganism |
| Electroporator 2510 | Eppendorf | 4307000.658 | Electroporator |
| Beef extract | Carl Roth GmbH | X975.2 | Media component |
| Peptone | Carl Roth GmbH | 2365.2 | Media component |
| Sucrose | Carl Roth GmbH | 4621.2 | Media component |
| Magnesium sulfate | Carl Roth GmbH | 0261.3 | Media component |
| Carbenicillin | Carl Roth GmbH | 6344.2 | Antibiotic |
| Kanamycin | Carl Roth GmbH | T832.3 | Antibiotic |
| Rifampicin | Carl Roth GmbH | 4163.2 | Antibiotic |
| FWD primer | Eurofins MWG Operon | n.a. | CCT CAG GAA GAG CAA TAC |
| REV primer | Eurofins MWG Operon | n.a. | CCA AAG CGA GTA CAC AAC |
| 2720 Thermal cycler | Applied Biosystems | 4359659 | Thermocycler |
| RNAfold webserver | University of Vienna | n.a. | Software |
| Ferty 2 Mega | Kammlott | 5.220072 | Fertilizer |
| Grodan Rockwool Cubes 10x10cm | Grodan | n.a. | Rockwool block |
| Greenhouse | n.a. | n.a. | For plant cultivation |
| Phytotron | Ilka Zell | n.a. | For plant cultivation |
| Omnifix-F Solo | B. Braun | 6064204 | Syringe |
| Murashige and Skoog salts | Duchefa | M 0222.0010 | Media component |
| Glucose | Carl Roth GmbH | 6780.2 | Media component |
| Acetosyringone | Sigma-Aldrich | D134406-5G | Phytohormon analogon |
| BioPhotometer plus | Eppendorf | 6132 000.008 | Photometer |
| Osram cool white 36 W | Osram | 4930440 | Light source |
| Disodium phosphate | Carl Roth GmbH | 4984.3 | Media component |
| Centrifuge 5415D | Eppendorf | 5424 000.410 | Centrifuge |
| Forma -86C ULT freezer | ThermoFisher | 88400 | Freezer |
| Synergy HT | BioTek | SIAFRT | Fluorescence plate reader |
| Biacore T200 | GE Healthcare | n.a. | SPR device |
| Protein A | Life technologies | 10-1006 | Antibody binding protein |
| HEPES | Carl Roth GmbH | 9105.3 | Media component |
| Tween-20 | Carl Roth GmbH | 9127.3 | Media component |
| 2G12 antibody | Polymun | AB002 | Reference antibody |
References
- Fischer, R., Emans, N. Molecular farming of pharmaceutical proteins. Transgenic research. 9, 277-299 (2000).
- Commandeur, U., Twyman, R. M., Fischer, R. The biosafety of molecular farming in plants. AgBiotechNet. 5, 9 (2003).
- Shoji, Y., et al. A plant-based system for rapid production of influenza vaccine antigens. Influenza Other Resp. 6, 204-210 (2012).
- Goodin, M. M., Zaitlin, D., Naidu, R. A., Lommel, S. A. Nicotiana benthamiana: Its history and future as a model for plant-pathogen interactions. Mol Plant Microbe In. 21, 1015-1026 (2008).
- Berg, R. H., Beachy, R. N. Fluorescent protein applications in plants. Method Cell Biol. 85, 153 (2008).
- Chung, S. M., Vaidya, M., Tzfira, T. Agrobacterium is not alone: gene transfer to plants by viruses and other bacteria. Trends in plant science. 11, 1-4 (2006).
- Sheludko, Y. V., Sindarovska, Y. R., Gerasymenko, I. M., Bannikova, M. A., Kuchuk, N. V. Comparison of several Nicotiana species as hosts for high-scale Agrobacterium-mediated transient expression. Biotechnology and Bioengineering. 96, 608-614 (2007).
- Wydro, M., Kozubek, E., Lehmann, P. Optimization of transient Agrobacterium-mediated gene expression system in leaves of Nicotiana benthamiana. Acta Biochimica Polonica. 53, 289-298 (2006).
- Buyel, J. F., Fischer, R. Processing heterogeneous biomass: Overcoming the hurdles in model building. Bioengineered. 4, (2013).
- Fischer, R., Schillberg, S., Hellwig, S., Twyman, R. M., Drossard, J. GMP issues for recombinant plant-derived pharmaceutical proteins. Biotechnol Adv. 30, 434-439 (2012).
- Daniel, C. One-at-a-time plans. Journal of the American Statistical Association. 68, 353-360 (1973).
- Czitrom, V. One-Factor-at-a-Time versus Designed Experiments The American Statistician. 53, 6 (1999).
- Anderson, M. J., Kraber, S. L. Keys to successful designed experiments. ASQ – The global voice of quality. 6, 6 (1999).
- Montgomery, D. C. . Design and Analysis of Experiments. , (2007).
- Myers, R. H., Montgomery, D. C., Anderson-Cook, C. M. . Response Surface Methodology: Process and Product Optimization Using Designed Experiments. , (2009).
- Piepel, G. F. Programs for generating extreme vertices and centroids of linearly constrained experimental regions. J Qual Technol. 20, 15 (1988).
- . . FDA. , (2009).
- Shivhare, M., McCreath, G. Practical Considerations for DoE Implementation in Quality By Design. BioProcess International. 8, 9 (2010).
- Buyel, J. F., Fischer, R. Predictive models for transient protein expression in tobacco (Nicotiana tabacum L.) can optimize process time, yield, and downstream costs. Biotechnology and bioengineering. 109, 2575-2588 (2012).
- Buyel, J. F., Kaever, T., Buyel, J. J., Fischer, R. Predictive models for the accumulation of a fluorescent marker protein in tobacco leaves according to the promoter/5’UTR combination. Biotechnology and bioengineering. 110, 471-482 (2013).
- Anderson, M. J., Whitcomb, P. J. . DOE Simplified: Practical Tools for Effective Experimentation. , (2000).
- Anderson, M. J., Whitcomb, P. J. . Response Surface Methods Simplified. , (2005).
- De Gryze, S., Langhans, I., Vandebroek, M. Using the correct intervals for prediction: A tutorial on tolerance intervals for ordinary least-squares regression. Chemometr Intell Lab. 87, 147-154 (2007).
- . . Plasmid DNA purification User manual. , (2012).
- . . PCR clean-up Gel extraction User manual. , (2012).
- . . Quick Ligation Protocol. 4, (2009).
- Inoue, H., Nojima, H., Okayama, H. High-Efficiency Transformation of Escherichia-Coli with Plasmids. Gene. 96, 23-28 (1990).
- Main, G. D., Reynolds, S., Gartland, J. S. Electroporation protocols for Agrobacterium. Methods in Molecular Biology. 44, 405-412 (1995).
- Gruber, A. R., Lorenz, R., Bernhart, S. H., Neubock, R., Hofacker, I. L. The Vienna RNA websuite. Nucleic acids research. 36, 70-74 (2008).
- Howell, S., Kenmore, M., Kirkland, M., Badley, R. A. High-density immobilization of an antibody fragment to a carboxymethylated dextran-linked biosensor surface. J Mol Recognit. 11, 200-203 (1998).
- Newcombe, A. R., et al. Evaluation of a biosensor assay to quantify polyclonal IgG in ovine serum used for the production of biotherapeutic antibody fragments. Process Biochem. 41, 842-847 (2006).
- Peixoto, J. L. Hierarchical Variable Selection in Polynomial Regression-Models. Am Stat. 41, 311-313 (1987).
- Peixoto, J. L. A Property of Well-Formulated Polynomial Regression-Models. Am Stat. 44, 26-30 (1990).
- Sanders, P. R., Winter, J. A., Barnason, A. R., Rogers, S. G., Fraley, R. T. Comparison of cauliflower mosaic virus 35S and nopaline synthase promoters in transgenic plants. Nucleic acids research. 15, 1543-1558 (1987).
- Ma, J. K. C., et al. Generation and Assembly of Secretory Antibodies in Plants. Science. 268, 716-719 (1995).
- Wycoff, K. L. Secretory IgA antibodies from plants. Curr Pharm Design. 11, 2429-2437 (2005).
- Pace, C. N., Vajdos, F., Fee, L., Grimsley, G., Gray, T. How to measure and predict the molar absorption coefficient of a protein. Protein Sci. 4, 2411-2423 (1995).

