Från Snabb fluorescens avbildning till molekylär diffusion lagen om Live cellmembran i en kommersiell mikroskop
Summary
Spatial distribution and temporal dynamics of plasma membrane proteins and lipids is a hot topic in biology. Here this issue is addressed by a spatio-temporal image fluctuation analysis that provides conceptually the same physical quantities of single particle tracking, but it uses small molecular labels and standard microscopy setups.
Abstract
Det har blivit alltmer uppenbart att den geografiska fördelningen och rörelse membrankomponenter såsom lipider och proteiner är nyckelfaktorer i regleringen av många cellulära funktioner. Men på grund av de snabba dynamiken och de små strukturer är involverade, är en mycket hög spatio-temporal upplösning som krävs för att fånga den verkliga beteendet hos molekyler. Här presenterar vi det experimentella protokollet för att studera dynamiken i fluorescensmärkta plasmamembran proteiner och lipider i levande celler med hög Spatiotemporal upplösning. Noterbart är, denna strategi behöver inte spåra varje molekyl, men det beräknar befolknings beteende med hjälp av alla molekyler i en viss region av membranet. Utgångspunkten är en snabb avbildning av en viss region på membranet. Efteråt är en komplett tid och rum autokorrelationsfunktion beräknas korrelerar förvärvade bilder öka tidsfördröjningar, exempelvis vardera 2, 3, n upprepningar. Det är möjligt att visa att breddeni toppen av de spatiala autokorrelationsfunktionen ökar vid ökande tidsfördröjning som funktion av partikelrörelse på grund av diffusion. Därför passar den serie av autokorrelationsfunktioner gör det möjligt att extrahera den faktiska proteinmedelkvadrat förskjutning från imaging (iMSD), här i form av sken diffusivitet vs genomsnittlig förskjutning. Detta ger en kvantitativ bild av de genomsnittliga dynamiken i enstaka molekyler med nanometer precision. Genom användning av en GFP-märkt variant av transferrinreceptorn (TfR) och en ATTO488 märkt 1-palmitoyl-2-hydroxi-sn-glycero-3-fosfoetanolamin (PPE) är det möjligt att observera spatiotemporal regleringen av protein och lipid diffusion på im stora membran regioner i mikro-till-milli-sekund tidsintervall.
Introduction
Med utgångspunkt från den ursprungliga "vätske mosaik" modell av Singer och Nicolson, bilden av cellplasmamembranet har kontinuerligt uppdaterats under de senaste decennierna för att inkludera den framväxande roll cytoskeleton och lipid domänerna 1,2.
De första observationerna erhölls genom fluorescerande återhämtning efter fotoblekning (FRAP) avtäckningen att en betydande fraktion av membranproteiner är orörlig 3-5. Dessa banbrytande studier, även om mycket informativt, led av den relativt dåliga upplösningen i rymden (mikron) och tid (sekunder) i FRAP inställningar. Dessutom är en ensemble medelvärdesmätning, FRAP saknar i att ge information om enda molekyl beteende.
I detta sammanhang är det möjligt att specifikt märka en enda molekyl med mycket ljusa taggar (möjliggör studiet av diffusionsprocessen en molekyl i taget) har varit mycket framgångsrikt. Speciellt, genom att trycka påtidsupplösning av den inre Particle Tracking (SPT) inställning till mikrosekunder tidsskalan, Kusumi, et al. fått tillgång till okända funktioner i lipid och proteindynamik som kraftigt bidragit till erkännandet av den roll aktin baserade membranskelett i membranfysiologi 6 , 7. Dessa fynd genererade den så kallade "strejkvakt och staket" modell, där lipid och protein diffusion regleras av aktin baserade skelettet. Men för att få tillgång till den enorma mängd information som SPT många experimentella frågor måste lösas. Speciellt är märkningsförfarandet vanligen består av många steg som produktion, rening och införande av de märkta arter i systemet. Dessutom stora etiketter, som kvantprickar eller metallnanopartiklar, ofta krävs för att nå under millisekund tid och tvärbindningen av målmolekyler med etiketten kunde inte undvikas i många fall. Slutligen, många banormåste registreras för att passa statistiska kriterier och samtidigt en låg densitet av etiketten krävs för att möjliggöra spårning.
Jämfört med SPT, fluorescens-korrelationsspektroskopi (FCS), övervinna många av dessa nackdelar, är ett mycket lovande tillvägagångssätt för att studera molekylär dynamik. I själva verket fungerar FCS bra även med dim och täta etiketter, som gör det möjligt att studera dynamiken i fluorescerande protein-märkta molekyler i gående transfekterade celler. Dessutom möjliggör det att nå höga statistik i en begränsad tid. Slutligen, trots den "höga" densitet av etiketter FCS erbjuder enstaka molekyler information. Tack vare alla dessa egenskaper, FCS är en mycket enkel metod och har tillämpats i stor utsträckning för att studera lipid och protein dynamik både i modellmembran och i levande-celler 8-10. Många olika metoder har föreslagits för att öka förmågan hos FCS att avslöja detaljerna i molekylär diffusion. Till exempel var det shegna som genom att utföra FCS på olika stora observationsområdena kan man definiera en "FCS diffusion lag" upplysande dolda funktioner i molekylär rörelse 11,12. Förutom att vara varierade i storlek, var det prioriterade området också dupliceras 13, flyttade i rymden längs linjerna 14-20 eller konjugerade med snabba kameror 21,22. Med hjälp av dessa "tid och rum" korrelation tillvägagångssätt, var relevanta biologiska parametrar för flera membrankomponenter kvantitativt beskrivas både modellmembran och faktiska biologiska sådana, vilket således ger inblick i membran rumslig organisation.
Men i alla FRAP och FCS-program hittills beskrivna storleken på det prioriterade området utgör en gräns i rumslig upplösning som inte kan övervinnas. Flera super upplösning avbildningsmetoder har nyligen utvecklats för att kringgå denna gräns. En del bygger på lokalisering precision, till exempel stokastiska optisk rekonstruktion mikroskopi (STORM) <sup> 23,24, foto lokalisering mikroskopi (PALM) 25, fluorescens PALM (FPALM) 26, och single-partikel tracking PALM (sptPALM) 27: den relativt stora mängd fotoner som krävs på varje stillbild, men begränsar den tid upplösning dessa metoder till åtminstone flera millisekunder, vilket hämmar deras tillämplighet in vivo.
Däremot har ett lovande alternativ för superupplösning avbildning öppnats av rumsligt modulera fluorescens emission med stimulerade utarmning utsläppsmetoder (STED eller reversibla mätt optiska fluorescens övergångar (RESOLFT)) 28,29. Dessa tillvägagångssätt kombinerar utformningen av observationsvolymen långt under diffraktionsgränsen med möjlighet att använda snabb scanning mikroskop och detekteringssystem. I kombination med fluorescensfluktuationer analys, STED mikroskopi tillåtet att direkt söka av nanoskala Spatiotemporal dynamik lipider och proteins i levande cellmembran 30,31.
Samma fysiska kvantiteter av STED baserad mikroskopi kan erhållas genom en modifierad tid och rum bildkorrelationsspektroskopi (STICS 32,33) metod som är lämplig för att studera dynamiken i fluorescerande-märkta membranproteiner och / eller lipider i levande celler och genom en kommersiell mikroskop. Den experimentella protokollet presenteras här består av några få steg. Den första kräver en snabb avbildning av regionen av intresse på membranet. Därefter resulte trave bilder används för att beräkna den genomsnittliga rumsliga-temporala korrelationsfunktioner. Genom att montera den serie av korrelationsfunktioner, kan den molekylära "diffusion lag" erhållas direkt från avbildning i form av en skenbar diffusivitet (D app) – vs -Genomsnittlig förskjutning tomt. Denna tomt är kritiskt beroende miljön utforskas av molekylerna och tillåter att erkänna direkt själva spridningssättav lipid / protein av intresse.
I mer detaljer, som tidigare visats 34, den tid och rum autokorrelationsfunktion för den förvärvade bildserie beror kritiskt på dynamiken i molekylerna rör sig i den insamlade bildserien (observera att samma resonemang kan tillämpas på en rad förvärv där bara en dimension i rymden anses). Särskilt definierar vi korrelationsfunktionen som:
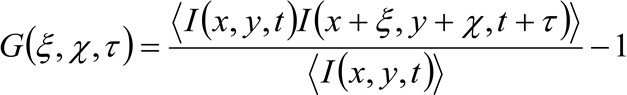 (1)
(1)
där 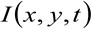 representerar den uppmätta fluorescensintensiteten i den position x, y och vid tiden t,
representerar den uppmätta fluorescensintensiteten i den position x, y och vid tiden t, ![]() och
och ![]() representerar avståndet i x-ochy-riktningarna respektive,
representerar avståndet i x-ochy-riktningarna respektive, ![]() representerar viss tid och
representerar viss tid och ![]() representerar genomsnittet. Denna funktion kan uttryckas som:
representerar genomsnittet. Denna funktion kan uttryckas som:
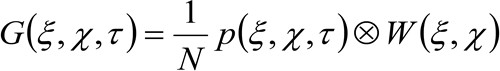 (2)
(2)
där "N" representerar det genomsnittliga antalet molekyler i området observation, ![]() representerar konvolution drift i rymden, och
representerar konvolution drift i rymden, och 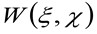 representerar autokorrelationen för den instrumen midjan. Detta senare kan tolkas som ett mått på hur fotoner från en enda sändare är utspridda i rymden på grund av att den optiska / inspelning setup (den så kallade punktspridningsfunktionen, PSF, genrally väl approximeras med en Gauss-funktion). Slutligen
representerar autokorrelationen för den instrumen midjan. Detta senare kan tolkas som ett mått på hur fotoner från en enda sändare är utspridda i rymden på grund av att den optiska / inspelning setup (den så kallade punktspridningsfunktionen, PSF, genrally väl approximeras med en Gauss-funktion). Slutligen 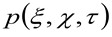 representerar sannolikheten för att finna en partikel på ett avstånd
representerar sannolikheten för att finna en partikel på ett avstånd ![]() och
och ![]() efter en tidsfördröjning
efter en tidsfördröjning ![]() . Om vi betraktar en diffusiva dynamik, där partiklar rör sig slumpmässigt i alla riktningar och nettoflöden inte är närvarande, är denna funktion också väl approximeras med en Gauss-funktion där variansen kan identifieras som Mean Square Displacement (MSD) i rörliga partikeln . Sålunda midjan på korrelationsfunktionen (även kallat
. Om vi betraktar en diffusiva dynamik, där partiklar rör sig slumpmässigt i alla riktningar och nettoflöden inte är närvarande, är denna funktion också väl approximeras med en Gauss-funktion där variansen kan identifieras som Mean Square Displacement (MSD) i rörliga partikeln . Sålunda midjan på korrelationsfunktionen (även kallat ![]() ), Kan definieras som summan av de partikel muskuloskeletala sjukdomar och den instrument midja och kan mätas genom en gaussisk passformting av korrelationsfunktionen för varje tidsfördröjning. Den uppmätta i MSD kan användas för att beräkna en skenbar diffusiviteten hos de rörliga molekyler
), Kan definieras som summan av de partikel muskuloskeletala sjukdomar och den instrument midja och kan mätas genom en gaussisk passformting av korrelationsfunktionen för varje tidsfördröjning. Den uppmätta i MSD kan användas för att beräkna en skenbar diffusiviteten hos de rörliga molekyler ![]() och en genomsnittlig förskjutning
och en genomsnittlig förskjutning ![]() som:
som:
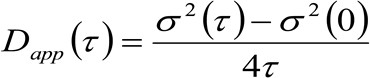 (3)
(3)
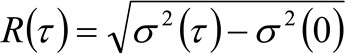 (4)
(4)
Några överväganden på experimentuppställning som används kan vägleda läsaren genom de följande avsnitten. För att selektivt excitera fluoroforema på basalmembranet av levande celler som vi kommer att använda en total inre reflektion (TIR) belysning, med hjälp av ett kommersiellt TIR fluorescens (TIRF) mikroskop (detaljer finns i materialet avsnitt). Dessutom, för att samla in the fluorescens vi kommer att använda en hög förstoring mål (100X NA 1,47, är hög numerisk apertur som krävs för TIRF belysning) och en EMCCD kamera (fysiska storleken på pixel på chipet 16 um). För att nå en pixelstorlek på 100 nm tillämpar vi en extra förstoringslins 1,6X. Såsom diskuteras nedan, skulle en tidsupplösning under en ms krävas för att korrekt beskriva dynamiken i snabba membranlipider nedanför 100 nm. För att uppnå denna tidsupplösning måste vi välja ett område av intresse (ROI) som är mindre än hela chipet av kameran (512 x 512). På detta sätt kommer kameran läsa ett reducerat antal linjer ökande tidsupplösningen. Men i denna avläsning regim ramtiden skulle begränsas av den tid som krävs för att flytta de avgifter från exponering för utläsningschipset på kameran och är oftast i storleksordningen millisekunder för 512 x 512 pixel EMCCD. För att slå denna gräns, kan en ny teknik flytta ROI-linjerna i stället för hela ramen, wed en praktisk effektiv minskning av den exponerade chip storlek (kallas Cropped Sensor läge i vårt EMCCD). För denna konfiguration för att vara effektiv, måste chipet utsidan av ROI täckas av ett par slitsar som är monterade i den optiska banan. Tack vare denna inställning en tidsupplösning ned till 10 -4 sekunder kan uppnås. Observera dock att detta tillvägagångssätt kan kopplas till många olika experimentella uppställningar, som förklaras i "diskussionen" avsnittet.
Demonstration av metoden kommer att tillhandahållas i levande celler, genom att använda både en ATTO488 märkt 1-palmitoyl-2-hydroxi-sn-glycero-3-fosfoetanolamin (ATTO488-PPE) och en GFP-märkt variant av transferrinreceptorn (GFP- TfR). I fallet med ATTO488-PPE kan detta tillvägagångssätt lyckades återställa en nästan konstant D appen som en funktion av genomsnittlig förskjutning indikerande en mestadels fri diffusion, såsom tidigare rapporterats 30,35. Däremot visar TfR-GFP en minskande D <sub> app som funktion av genomsnittlig förskjutning, vilket tyder delvis begränsad diffusion 6. Dessutom är det i det senare fallet är möjligt att kvantifiera den lokala diffusionskonstanten och genomsnitts inneslutning område under många mikrometer på membranplanet.
Protocol
Representative Results
Discussion
Spårning enda partikel (SPT) är en av de vanligaste strategierna för att studera molekyldynamik och har den stora fördelen att mäta partikelbanor. Detta i sin tur gör det möjligt att sondera beteende ens några märkta partiklar i ett komplext system. Men för att nå denna fördel SPT behöver normalt en låg densitet av sonden och mycket ljusa etiketter. Särskilt för att få hög tidsupplösning (ps intervall) oorganiska sond krävs ofta (t.ex. kvantprickar eller metallnanopartiklar): i detta fall en…
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work is supported in part by NIH-P41 P41-RRO3155 and NIH P50-GM076516 (grant to EG), and Fondazione Monte dei Paschi di Siena (grant to FB).
Materials
| Name of Material/ Equipment | Company | Catalog Number | Comments/Description |
| iXon Ultra 897 | Andor | DU-897U-CS0 | |
| Solis | Andor | ||
| CHO-K1 | ATCC | CCL-61 | |
| ATTO 488 labeled PPE | ATTO-TEC GmbH | AD 488-151 | |
| DOPE | Avanti Polar Lipids, Inc. | 850725 | |
| DOTAP | Avanti Polar Lipids, Inc. | 890890 | |
| 100X Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | |
| DMEM/F-12 | Gibco | 21331 | |
| FBS | Gibco | 10082147 | |
| HEPES | Gibco | 15630-106 | |
| PBS | Gibco | 10010-023 | |
| SimFCS 3.0 | Globals Software | the software can be downloaded here: http://www.lfd.uci.edu/globals/ | |
| DMI6000 with TIRF modulus | Leica | ||
| LAS AF | Leica | ||
| Lipofectamine 2000 | Lipofectamine | 11668019 | |
| Matlab | MathWork | ||
| Imagej | NIH | ||
| C-terminal GFP tagged Tranferrin Receptor | OriGene | RG200980 | |
| Agar | Sigma Aldrich | A5306 | |
| Chloroform | Sigma Aldrich | 528730 | |
| Latex beads, fluorescent yellow-green, 30 nm | Sigma Aldrich | L5155 | |
| SONICA Ultrasonic Cleaners | SOLTEC | ETH S3 | |
| Petri Dishes | Willco | GWSt-3522 | |
| Bio-Format importer for Matlab | http://www.openmicroscopy.org/site/support/bio-formats5/users/matlab/ | ||
| ICS-MatLab Tools | https://www.cellmigration.org/resource/imaging/software/ICSMATLAB_28-02-06.zip | ||
| Simulation by Matlab Tutorial | https://www.cellmigration.org/resource/imaging/icsmatlab/ICSTutorial.html | ||
| Simulation by SimFCS Tutorial | https://www.cellmigration.org/resource/imaging/ppt-pdf/RICS%20Simulations.ppt |
References
- Engelman, D. M. Membranes are more mosaic than fluid. Nature. 438 (7068), 578-580 (2005).
- Vereb, G., et al. yet structured: The cell membrane three decades after the Singer-Nicolson model. Proc. Natl. Acad. Sci. U. S. A. 100 (14), 8053-8058 (1073).
- Ishihara, A., Hou, Y., Jacobson, K. The Thy-1 antigen exhibits rapid lateral diffusion in the plasma membrane of rodent lymphoid cells and fibroblasts. 84 (5), 1290-1293 (1987).
- Axelrod, D., et al. Lateral motion of fluorescently labeled acetylcholine receptors in membranes of developing muscle fibers. Proc. Natl. Acad. Sci. U. S. A. 73 (12), 4594-4598 (1976).
- Jacobson, K., Derzko, Z., Wu, E. S., Hou, Y., Poste, G. Measurement of the lateral mobility of cell surface components in single, living cells by fluorescence recovery after photobleaching. J. Supramol. Struct. 5 (4), 10-1002 (1976).
- Kusumi, A., et al. Paradigm shift of the plasma membrane concept from the two-dimensional continuum fluid to the partitioned fluid: high-speed single-molecule tracking of membrane molecules. Annu. Rev. Biophys. Biomol. Struct. 34, 351-378 (2005).
- Kusumi, A., Ike, H., Nakada, C., Murase, K., Fujiwara, T. Single-molecule tracking of membrane molecules: plasma membrane compartmentalization and dynamic assembly of raft-philic signaling molecules. Semin. Immunol. 17 (1), 3-21 (2005).
- Schwille, P., Korlach, J., Webb, W. W. Fluorescence correlation spectroscopy with single-molecule sensitivity on cell and model membranes. Cytometry. 36, 176-182 (1999).
- Gielen, E., et al. Diffusion of sphingomyelin and myelin oligodendrocyte glycoprotein in the membrane of OLN-93 oligodendroglial cells studied by fluorescence correlation spectroscopy. C. R. Biol. 328 (12), 1057-1064 (2005).
- Weiss, M., Hashimoto, H., Nilsson, T. Anomalous protein diffusion in living cells as seen by fluorescence correlation spectroscopy. Biophys. J. 84, 4043-4052 (2003).
- Wawrezinieck, L., Rigneault, H., Marguet, D., Lenne, P. F. Fluorescence correlation spectroscopy diffusion laws to probe the submicron cell membrane organization. Biophys. J. 89 (6), 4029-4042 (2005).
- Lenne, P. F., et al. Dynamic molecular confinement in the plasma membrane by microdomains and the cytoskeleton meshwork. EMBO J. 25 (14), 3245-3256 (2006).
- Ries, J., Schwille, P. Studying slow membrane dynamics with continuous wave scanning fluorescence correlation spectroscopy. Biophys. J. 91 (5), 1915-1924 (2006).
- Ruan, Q., Cheng, M. A., Levi, M., Gratton, E., Mantulin, W. W. Spatial-temporal studies of membrane dynamics: scanning fluorescence correlation spectroscopy (SFCS). Biophys. J. 87 (2), 1260-1267 (2004).
- Berland, K. M., So, P. T., Chen, Y., Mantulin, W. W., Gratton, E. Scanning two-photon fluctuation correlation spectroscopy: particle counting measurements for detection of molecular aggregation. Biophys. J. 71, 410-420 (1996).
- Heinemann, F., Betaneli, V., Thomas, F. A., Schwille, P. Quantifying lipid diffusion by fluorescence correlation spectroscopy: a critical treatise. Langmuir. 28 (37), 13395-13404 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Capturing directed molecular motion in the nuclear pore complex of live cells. Proc. Natl. Acad. Sci. U. S. A. 109 (25), 9863-9868 (2012).
- Sanchez, S. A., Tricerri, M. A., Gratton, E. Laurdan generalized polarization fluctuations measures membrane packing micro-heterogeneity in vivo. Proc. Natl. Acad. Sci. U. S. A. 109 (19), 7314-7319 (2012).
- Cardarelli, F., Lanzano, L., Gratton, E. Fluorescence correlation spectroscopy of intact nuclear pore complexes. Biophys. J. 101 (4), 27-29 (2012).
- Di Rienzo, C., et al. Unveiling LOX-1 receptor interplay with nanotopography: mechanotransduction and atherosclerosis onset. Sci. Rep. 3, 10-1038 (2013).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophys. J. 95 (11), 5385-5398 (2008).
- Kannan, B., et al. Electron multiplying charge-coupled device camera based fluorescence correlation spectroscopy. Anal. Chem. 78 (10), 3444-3451 (2006).
- Jones, S. A., Shim, S. H., He, J., Fast Zhuang, X. three-dimensional super-resolution imaging of live cells. Nat. Methods. 8 (6), 499-508 (2011).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy. 3 (10), 793-795 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Hess, S. T., Girirajan, T. P., Mason, M. D. Ultra-high resolution imaging by fluorescence photoactivation localization microscopy. Biophys. J. 91 (11), 4258-4272 (2006).
- Manley, S., et al. High-density mapping of single-molecule trajectories with photoactivated localization microscopy. Nat. Methods. 5 (2), 155-157 (2008).
- Hell, S. W. Far-field optical nanoscopy. Science. 316 (5828), 1153-1158 (2007).
- Klar, T. A., Hell, S. W. Subdiffraction resolution in far-field fluorescence microscopy. Opt. Lett. 24 (14), 954-956 (1999).
- Eggeling, C., et al. Direct observation of the nanoscale dynamics of membrane lipids in a living cell. Nature. 457 (7233), 1159-1162 (2009).
- Hedde, P. N., et al. Stimulated emission depletion-based raster image correlation spectroscopy reveals biomolecular dynamics in live cells. Nat. Commun. 4, .
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophys. J. 88 (5), 3601-3614 (2005).
- Brown, C. M., et al. Probing the integrin-actin linkage using high-resolution protein velocity mapping. J. Cell Sci. 119, 5204-5214 (2006).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proc. Natl. Acad. Sci. U. S. A. 110 (30), 12307-12312 (2013).
- Mueller, V., et al. STED nanoscopy reveals molecular details of cholesterol- and cytoskeleton-modulated lipid interactions in living cells. Biophys. J. 101 (7), 1651-1660 (2011).
- Kleusch, C., Hersch, N., Hoffmann, B., Merkel, R., Csiszar, A. Fluorescent lipids: functional parts of fusogenic liposomes and tools for cell membrane labeling and visualization. Molecules. 17 (1), 1055-1073 (2012).
- Ries, J., Chiantia, S., Schwille, P. Accurate determination of membrane dynamics with line-scan FCS. Biophys. J. 96 (5), 1999-2008 (2009).
- Kolin, D. L., Wiseman, P. W. Advances in image correlation spectroscopy: measuring number densities, aggregation states, and dynamics of fluorescently labeled macromolecules in cells. Cell Biochem. Biophys. 49 (3), 141-164 (2007).
- Digman, M. A., et al. Measuring fast dynamics in solutions and cells with a laser scanning microscope. Biophys. J. 89 (2), 1317-1327 (2005).
- Ritchie, K., et al. Detection of non-Brownian diffusion in the cell membrane in single molecule tracking. Biophys. J. 88 (3), 2266-2277 (2005).
- Voie, A. H., Burns, D. H., Spelman, F. A. Orthogonal-plane fluorescence optical sectioning: three-dimensional imaging of macroscopic biological specimens. J. Microsc. 170, 229-236 (1993).
- Huisken, J., Swoger, J., Del Bene, ., Wittbrodt, F., J, E. H., Stelzer, Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Wohland, T., Shi, X., Sankaran, J., Stelzer, E. H. Single plane illumination fluorescence correlation spectroscopy (SPIM-FCS) probes inhomogeneous three-dimensional environments. Opt. Express. 18 (10), 10627-10641 (2010).

