Yaratma CRISPR / Cas9 aracılığı monoallelik Delesyonlar Fare Embriyonik Kök Hücreleri Artırıcı Fonksiyonu Eğitim için
Summary
Experimental validation of enhancer activity is best approached by loss-of-function analysis. Presented here is an efficient protocol that uses CRISPR/Cas9 mediated deletion to study allele-specific regulation of gene transcription in F1 ES cells which contain a hybrid genome (Mus musculus129 x Mus castaneus).
Abstract
Enhancers control cell identity by regulating tissue-specific gene expression in a position and orientation independent manner. These enhancers are often located distally from the regulated gene in intergenic regions or even within the body of another gene. The position independent nature of enhancer activity makes it difficult to match enhancers with the genes they regulate. Deletion of an enhancer region provides direct evidence for enhancer activity and is the gold standard to reveal an enhancer’s role in endogenous gene transcription. Conventional homologous recombination based deletion methods have been surpassed by recent advances in genome editing technology which enable rapid and precisely located changes to the genomes of numerous model organisms. CRISPR/Cas9 mediated genome editing can be used to manipulate the genome in many cell types and organisms rapidly and cost effectively, due to the ease with which Cas9 can be targeted to the genome by a guide RNA from a bespoke expression plasmid. Homozygous deletion of essential gene regulatory elements might lead to lethality or alter cellular phenotype whereas monoallelic deletion of transcriptional enhancers allows for the study of cis-regulation of gene expression without this confounding issue. Presented here is a protocol for CRISPR/Cas9 mediated deletion in F1 mouse embryonic stem (ES) cells (Mus musculus129 x Mus castaneus). Monoallelic deletion, screening and expression analysis is facilitated by single nucleotide polymorphisms (SNP) between the two alleles which occur on average every 125 bp in these cells.
Introduction
Kopyalama düzenleyici elemanlar nedeniyle anormal gen ifadesinin 2 hastalığın neden olabilir, bu elemanların geliştirilmesi 1 ve modifikasyonu esnasında gen sentezlenmesinin yerleşim-zaman ince ayar için kritik öneme sahiptir. Genom ilişki çalışmaları ile tanımlanan birçok hastalıkla ilişkili bölgeler kodlayıcı olmayan bölgelerinde ve transkripsiyon arttırıcı 3-4 özelliklerine sahiptir. Güçlendiricileri belirlenmesi ve genellikle birkaç kilobazlık uzaklıkta da düzenleyen genler arasında yer almaktadır ve dokuya özel bir tarzda 5-6 aktive edilebilir karmaşık düzenleyen genler ile bulundu. Artırıcı tahminleri yaygın histon modifikasyonu işaretleri, arabulucu-kohezinin kompleksleri dayalı ve hücre tipine spesifik transkripsiyon bağlayıcı 7-10 faktörler vardır. Tahmin edilen arttırıcı validasyonu çoğunlukla arttırıcı bir raportör gen 11-12 ekspresyonunu aktive olan bir vektör bazlı deney yapılır. Bu veriler, v sağlarvarsayılan arttırıcı dizilerinin düzenleyici potansiyeli hakkında aluable bilgi ama onların endojen genomik bağlamda kendi işlevini açığa ya da düzenleyen genleri tanımlamak yok. Genom düzenleme kaybı fonksiyon-analizi ile kendi endojen bağlamda transkripsiyonel düzenleyici elemanlarının fonksiyonunu incelemek için güçlü bir araç olarak hizmet vermektedir.
genom düzenleme, yani CRISPR / Cas9 genom düzenleme sisteminde son gelişmeler, genom fonksiyonunun soruşturma kolaylaştırmak. CRISPR / Cas9 sistemi birçok biyolojik sistemler için kullanımı kolay ve uyarlanabilir. Cas9 proteini bir kılavuz RNA (gRNA) 13 tarafından genomunda belirli bir site hedeflenmektedir. SpCas9 / gRNA kompleksi protospacer bitişik motifi (PAM) dizisi, NGG 14-15 5 'olmalıdır hedef genomik dizisi için genom tarar. hedefine gRNA, gRNA tamamlayıcı olan bir 20 nükleotid (nt) dizisi baz çiftleşmesi, doubl elde SpCas9 nükleaz aktivitesi aktivee iplikli sonu (DSB) PAM dizisinin yukarı 3 bp. Özgünlük gRNA tohum bölgede tam baz eşleşmesi ile elde edilir, 6-12 PAM bitişik nt; Bunun aksine, tohum, genellikle 16-17 tolere edilir '5 uymuyordur. katılmadan homolog olmayan sonunda (NHEJ) DNA onarım veya homoloji yönettiği onarım (HDR) mechanisms.NHEJ DNA onarım sık sık bozabilir hedef bölgeye bir kaç bp ekleme / silme (indeller) oluşturur ya tanıttı DSB tamir edilebilir bir genin açık okuma çerçevesinin (ORF). Ilgili bölgeyi kuşatan genom iki gRNAs, daha büyük silme oluşturmak için 18-19 kullanılabilir. Bu yaklaşım, lokus kontrol bölgeleri ya da geleneksel arttırıcılar 9,18,20-22 daha büyük olan süper arttırıcı olarak kümelenmiş transkripsiyonel arttırıcılar çalışma için özellikle yararlıdır.
Monoallelik silmeler transkripsiyon sis -Yönetmeliği çalışmak için değerli bir modeldir. gözlenen changBir artırıcı madde monoallelik silindikten sonra transkript düzeyinde e hem alel transkripsiyon hücresel uygunluğu etkileyen potansiyel etkilenir oluşabilir karıştırıcı etkileri olmadan gen regülasyonu bu güçlendirici rolü ilişkilidir. azaltılmış ifade değerlendirilmeden ancak vahşi tip allel silinmiş ayırt yeteneği olmadan zordur. Ayrıca, iki allel ayırt yeteneği olmadan her alel de silmeleri genotipleme özellikle PCR ile tüm yabani tip bölgeyi yükseltmek için zor olduğu 1 Mb 23> 10 kb büyük delesyonlar için, zordur. Mus castaneus ile aradaki Mus musculus 129 tarafından üretilen F1 ES hücrelerinin kullanımı iki aleli alel-spesifik PCR 18,24 ayırt edilmesini sağlar. Bu hücrelerde melez genom alel spesifik silme tarama ve ifade analizi kolaylaştırır. Ortalama olarak bu iki genomları arasındaki her 125 bp bir SNP varİfade ve genotip için astar tasarım esnekliği sağlayan analiz eder. Bir SNP varlığı astar erime sıcaklığı (Tm) etkilemek ve iki allel 25 ayrımcılık sağlayan gerçek zamanlı kantitatif PCR (qPCR) amplifikasyon özgüllük hedefleyebilir. Ayrıca primerin 3 'ucu olan bir uyumsuzluk büyük ölçüde istenmeyen bir alel hedef 26 amplifikasyonunu önleyici astar uzanacak şekilde DNA polimerazın yeteneğini etkiler. CRISPR / Cas9 genom düzenleme sistemi (Şekil 1) kullanılarak daha büyük 1 kb alel spesifik güçlendirici kromozom anormallikleri ve daha sonra ekspresyon analizi için F1 ES hücrelerinin kullanımı, aşağıdaki protokolde tarif edilmektedir.
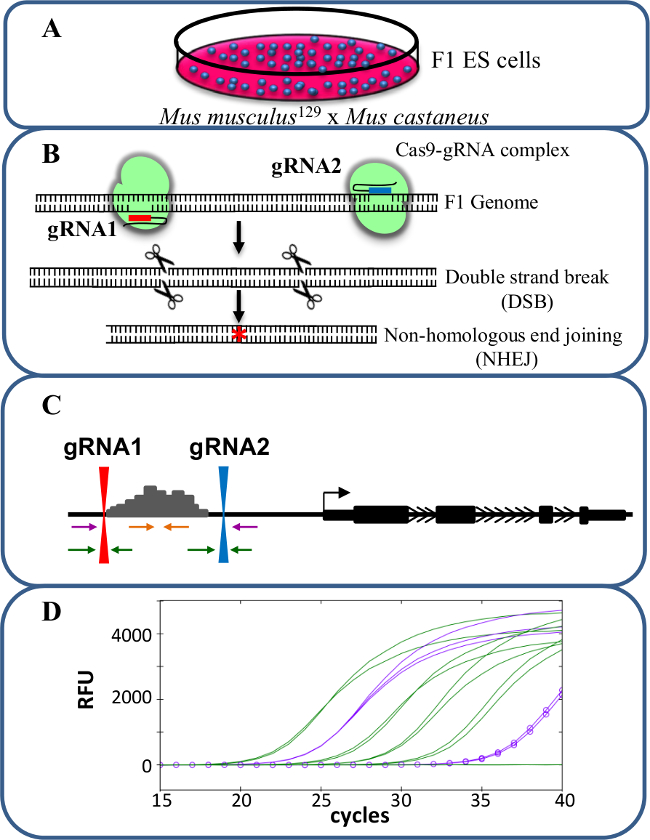
Sis -reg incelemek için CRISPR / Cas9 kullanarak Şekil 1. Artırıcı silmeGen ifadesinin ulation. Mus musculus 129 ve Mus castaneus arasında çapraz tarafından üretilen (A) F1 ES hücreleri alel spesifik silinmesi için izin vermek için kullanılır. (B) iki kılavuz RNA'lar (gRNA) geliştirici bölgesinin büyük Cas9 aracılı silme indüklemek için kullanılır. (C) Primer setleri büyük mono- ve bi-alelik silme tanımlamak için kullanılır. Turuncu primerler içinde primerler olarak mor primerler dış primerleri ve yeşil primerler gRNA kuşatan primerlerdir bulunmaktadır. Gen ekspresyonunda (D) değişiklikleri alel spesifik qPCR kullanılarak izlenmektedir. RFU nispi floresan üniteleri gösterir. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
Protocol
Representative Results
Discussion
CRISPR / Cas9 aracılı genom düzenleme teknolojisi genom değişiklik için, basit, hızlı ve ucuz bir yöntem sağlar. Fonksiyonel güçlendirici karakterizasyonu için monoallelik güçlendirici iptali gerçekleştirilebilir ve analiz etmek için burada açıklanan yöntem F1 fare hücrelerinde SNP yararlanır. Bu tip bir yaklaşımın avantajları şunlardır: 1) monoallelik güçlendirici silme kritik güçlendirici örneğin, her iki allellerinin ölümcül hücre gelen düzenlenmiş genin protein düze…
Disclosures
The authors have nothing to disclose.
Acknowledgements
We would like to thank all the members of the Mitchell lab for helpful discussions. This work was supported by the Canadian Institutes of Health Research, the Canada Foundation for Innovation and the Ontario Ministry of Research and Innovation (operating and infrastructure grants held by JAM).
Materials
| Phusion High-Fidelity DNA Polymerase | NEB | M0530S | high fidelity DNA polymerase used in gRNA assembly |
| Gibson Assembly Master Mix | NEB | E2611L | |
| gRNA_Cloning Vector | Addgene | 41824 | A target sequence is cloned into this vector to create the gRNA plasmid |
| pCas9_GFP | Addgene | 44719 | Codon-optimized SpCas9 and EGFP co-expression plasmid |
| AflII | NEB | R0520S | |
| EcoRI | NEB | R3101S | |
| Neon Transfection System 100 µL Kit | Life Technologies | MPK10096 | Microporator transfection technology |
| prepGEM | ZyGEM | PT10500 | genomic DNA extraction reagent |
| Nucleo Spin Gel & PCR Clean-up | Macherey-Nagel | 740609.5 | |
| High-Speed Plasmid Mini Kit | Geneaid | PD300 | |
| Maxi Plasmid Kit Endotoxin Free | Geneaid | PME25 | |
| SYBR select mix for CFX | Life Technologies | 4472942 | qPCR reagent |
| iScript cDNA synthesis kit | Bio-rad | 170-8891 | Reverse transcription reagent |
| 0.25% Trypsin with EDTA | Life Technologies | 25200072 | |
| PBS without Ca/Mg2+ | Sigma | D8537 | |
| 0.5M EDTA | Bioshop | EDT111.500 | |
| HBSS | Life Technologies | 14175095 | |
| 1M HEPES | Life Technologies | 13630080 | |
| BSA fraction V (7.5%) | Life Technologies | 15260037 | |
| Max Efficiency DH5α competent cells | Invitrogen | 18258012 | |
| FBS | ES cell qualified | FBS is subjected to a prior testing in mouse ES cells for pluripotency | |
| DMSO | Sigma | D2650 | |
| Glutamax | Invitrogen | 35050 | |
| DMEM | Life Technologies | 11960069 | |
| Pencillin/Streptomycin | Invitrogen | 15140 | |
| Sodium pyruvate | Invitrogen | 11360 | |
| Non-essential aminoacid | Invitrogen | 11140 | |
| β-mercaptoethanol | Sigma | M7522 | |
| 96-well plate | Sarstedt | 83.3924 | |
| Sealing tape | Sarstedt | 95.1994 | |
| CoolCell LX | Biocision | BCS-405 | alcohol-free cell freezing container |
| CHIR99021 | Biovision | 1748-5 | Inhibitor for F1 ES cell culture |
| PD0325901 | Invivogen | inh-pd32 | Inhibitor for F1 ES cell culture |
| LIF | Chemicon | ESG1107 | Inhibitor for F1 ES cell culture |
References
- Sagai, T., Hosoya, M., Mizushina, Y., Tamura, M., Shiroishi, T. Elimination of a long-range cis-regulatory module causes complete loss of limb-specific Shh expression and truncation of the mouse limb. Development. 132 (4), 797-803 (2005).
- Kleinjan, D. A., Lettice, L. A. Long-range gene control and genetic disease. Adv Genet. 61, 339-388 (2008).
- Visel, A., Rubin, E. M., Pennacchio, L. A. Genomic views of distant-acting enhancers. Nature. 461 (7261), 199-205 (2009).
- Maurano, M. T., et al. Systematic localization of common disease-associated variation in regulatory DNA. Science. 337 (6099), 1190-1195 (2012).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Shen, Y., et al. A map of the cis-regulatory sequences in the mouse genome. Nature. 488 (7409), 116-120 (2012).
- Johnson, D. S., Mortazavi, A., Myers, R. M., Wold, B. Genome-wide mapping of in vivo protein-DNA interactions. Science. 316 (5830), 1497-1502 (2007).
- Rhee, H. S., Pugh, B. F. Comprehensive genome-wide protein-DNA interactions detected at single-nucleotide resolution. Cell. 147 (6), 1408-1419 (2011).
- Whyte, W. A., et al. Master transcription factors and mediator establish super-enhancers at key cell identity genes. Cell. 153 (2), 307-319 (2013).
- Chen, C. Y., Morris, Q., Mitchell, J. A. Enhancer identification in mouse embryonic stem cells using integrative modeling of chromatin and genomic features. BMC Genomics. 13 (1), 152 (2012).
- Patwardhan, R. P., et al. Massively parallel functional dissection of mammalian enhancers in vivo. Nat Biotechnol. 30 (3), 265-270 (2012).
- Melnikov, A., et al. Systematic dissection and optimization of inducible enhancers in human cells using a massively parallel reporter assay. Nat Biotechnol. 30 (3), 271-277 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Cho, S. W., et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases. Genome Res. 24 (1), 132-141 (2014).
- Zhou, H. Y., et al. A Sox2 distal enhancer cluster regulates embryonic stem cell differentiation potential. Genes Dev. 28 (24), 2699-2711 (2014).
- Fujii, W., Kawasaki, K., Sugiura, K., Naito, K. Efficient generation of large-scale genome-modified mice using gRNA and CAS9 endonuclease. Nucleic Acids Res. 41 (20), e187 (2013).
- Tuan, D. Y., Solomon, W. B., London, I. M., Lee, D. P. An erythroid-specific, developmental-stage-independent enhancer far upstream of the human ‘beta-like globin’ genes. Proc Natl Acad Sci U S A. 86 (8), 2554-2558 (1989).
- Amano, T., et al. Chromosomal dynamics at the Shh locus: limb bud-specific differential regulation of competence and active transcription. Dev Cell. 16 (1), 47-57 (2009).
- Li, Y., et al. CRISPR reveals a distal super-enhancer required for Sox2 expression in mouse embryonic stem cells. PLoS One. 9 (12), e114485 (2014).
- Canver, M. C., et al. Characterization of genomic deletion efficiency mediated by clustered regularly interspaced palindromic repeats (CRISPR)/Cas9 nuclease system in mammalian cells. J Biol Chem. 289 (31), 21312-21324 (2014).
- Mlynarczyk-Evans, S., et al. X chromosomes alternate between two states prior to random X-inactivation. PLoS Biol. 4 (6), e159 (2006).
- Lefever, S., Pattyn, F., Hellemans, J., Vandesompele, J. Single-nucleotide polymorphisms and other mismatches reduce performance of quantitative PCR assays. Clin Chem. 59 (10), 1470-1480 (2013).
- Huang, M. M., Arnheim, N., Goodman, M. F. Extension of base mispairs by Taq DNA polymerase: implications for single nucleotide discrimination in PCR. Nucleic Acids Res. 20 (17), 4567-4573 (1992).
- Keane, T. M., et al. Mouse genomic variation and its effect on phenotypes and gene regulation. Nature. 477 (7364), 289-294 (2011).
- Yalcin, B., et al. Sequence-based characterization of structural variation in the mouse genome. Nature. 477 (7364), 326-329 (2011).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Gibson, D. G., Smith, H. O., Hutchison, C. A., Venter, J. C., Merryman, C. Chemical synthesis of the mouse mitochondrial genome. Nat Methods. 7 (11), 901-903 (2010).
- Ding, Q., et al. Enhanced efficiency of human pluripotent stem cell genome editing through replacing TALENs with CRISPRs. Cell Stem Cell. 12 (4), 393-394 (2013).
- Basu, S., Campbell, H. M., Dittel, B. N., Ray, A. Purification of specific cell population by fluorescence activated cell sorting (FACS). J Vis Exp. (41), (2010).
- Forlenza, M., Kaiser, T., Savelkoul, H. F., Wiegertjes, G. F. The use of real-time quantitative PCR for the analysis of cytokine mRNA levels. Methods Mol Biol. 820, 7-23 (2012).
- Wu, J. H., Hong, P. Y., Liu, W. T. Quantitative effects of position and type of single mismatch on single base primer extension. J Microbiol Methods. 77 (3), 267-275 (2009).
- Sanyal, A., Lajoie, B. R., Jain, G., Dekker, J. The long-range interaction landscape of gene promoters. Nature. 489 (7414), 109-113 (2012).

