软件辅助跟踪动态细胞突变中蛋白质浓度的图形用户界面
Summary
我们提供了一个软件解决方案,用于半自动跟踪动态细胞突起长度的相对蛋白质浓度。
Abstract
Filopodia是与迁移和细胞 – 细胞通信相关联的动态的,指状的细胞突起。为了更好地了解潜伏期起始,延长和随后的稳定或收缩的复杂信号传导机制,确定这些动态结构中的时空蛋白质活性至关重要。为了分析丝状伪足中的蛋白质功能,我们最近开发了一种半自动跟踪算法,适应线形形状变化,从而允许平行分析突起动力学和沿整个长度的整个相对蛋白质浓度。在这里,我们提供了一个详细的逐步协议,用于优化单元处理,图像采集和软件分析。我们进一步提供在图像分析和数据表示期间使用可选功能的说明,以及所有关键步骤的故障排除指南。最后,我们还包括d的比较带有图像分析软件的其他程序可用于丝状伪迹定量。在一起,所提出的方案提供了使用图像分析软件准确分析丝状体突起中的蛋白质动力学的框架。
Introduction
肌动蛋白调节蛋白的时空控制与纤维素酶动力学1,2相关。随着时间的推移,跟踪沿着整个网络长度的空间分辨蛋白质浓度对于促进我们对这些动态结构的起始,伸长,稳定或崩溃的机理的理解至关重要3,4 。不同于细胞质中的蛋白质分析,其中许多细胞形状变化发生在更大的尺度,丝状伪足是持续弯曲5并弯曲的动态微结构,因此使用简单的方法(例如线扫描)排除分析。
可用于跟踪丝状体形状的不同软件解决方案6,7,8,9。 Likewise,细胞体内蛋白质动力学的比例跟踪软件已经开发10,11 。为了结合自动跟踪网格形状和时空蛋白质分析,我们最近开发了基于凸包算法的图像分析软件12 。这种通过图形用户界面(GUI)操作的新型分析方法首次将沿着丝状体长度和生长速度的相对蛋白质浓度相结合,从而允许精确测量时空蛋白质分布,而与这些蛋白质的运动无关动态结构12 。
软件背后的想法(源代码可以免费获得,见下文),凸包的顶点之一将与圆顶的尖端一致( 图1A )。通过查看后续框架for最近的凸包顶点,可以在整个影片中追踪移动的尖端。一旦在每个框架中检测到尖端,它的位置用于通过将尖端与光栅底部的参考点连接起来绘制轴( 图1B )。最后,使用等距节点,其位置由具有与轴正交的线上的最大强度的中值像素确定,用于确定跟随斑点形状的骨架。利用这种自适应骨架,产生一个色谱图 ,以沿着丝状体长度追踪多达三个通道的丝状体生长和蛋白质浓度( 图1C )。
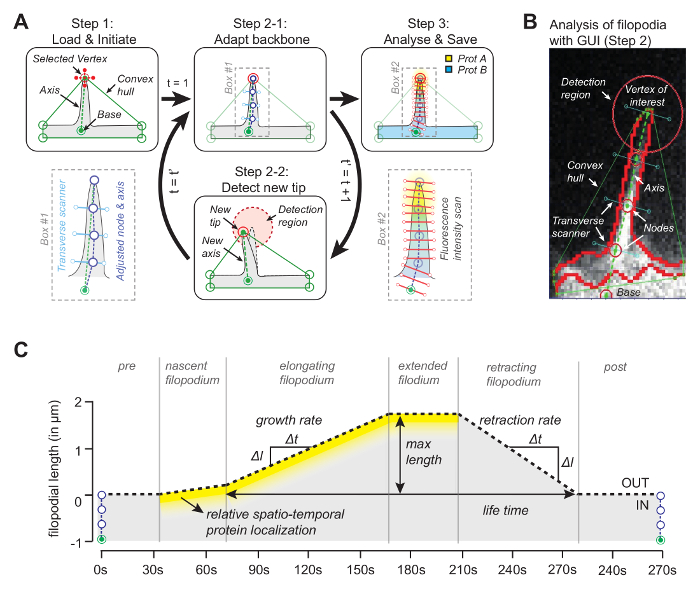
图1:图像分析软件的工作原理。 ( A )后面的算法该软件。在步骤1中,用户指定参考(基数)和顶点的顶点(tip)。在步骤2-1中,使用具有最大强度值的中值像素获得光栅的骨架。在步骤3中,骨架用于空间蛋白质强度分布。在步骤2-2中,软件会自动跟踪后续帧中的提示。整个过程迭代。 ( B )算法的快照,其中包含真正的光纤引入重要元素,例如用于跟踪的凸包。 ( C )使用算法可以测量的参数概述。这个数字已经从参考文献12中修改过。 请点击此处查看此图的较大版本。
图像分析软件通过图形使用在Matlab(简称编程软件)中运行r接口。为了使特定实验设置的灵活性和鲁棒性达到最大化,用户可以调整一系列跟踪参数( 例如允许的弯曲角度和帧间移动),并对电影进行一些修正( 例如裁剪,旋转,删除不需要的对象) ( 图2A和表1) 。
| GUI | 没有。 | 强制性 | 描述 | 名称(在GUI中) | ||||
| #1 | 1A | ÿ | 加载堆叠的.tiff文件,表示单元格主体(框已检入)或从通道创建叠加的单元格体 | CellBody | ||||
| #1 | 1B | ÿ | <td colspan=“5”>加载与蛋白质1相对应的堆叠图像文件蛋白质1 | |||||
| #1 | 1C | ÿ | 加载对应于蛋白质2的堆叠图像文件 | 蛋白质2 | ||||
| #1 | 1D | ÿ | 加载对应于蛋白质3的堆叠图像文件 | 蛋白质3 | ||||
| #1 | 1E | ñ | 将所有内容重置为预先加载的堆叠图像文件 | 重启 | ||||
| #1 | 2A | ÿ | 滚动条确定GUI窗口#2中分析的初始帧 | NA | ||||
| #1 | 2B | ÿ | 滚动条,以确定在GUI窗口#2中进行分析的最终框架 | NA | ||||
| #1 | 2C | ÿ | 代表卷曲的滚动条nt框架 | NA | ||||
| #1 | 2D | ñ | 所有像素将被设置为零以下的像素的灰度值 | NA | ||||
| #1 | 2E | ñ | 所有像素将被设置为最大值的像素的灰度值 | NA | ||||
| #1 | 2F | ñ | 设置由<2e>&<2f>指定的像素的强度值 | 组 | ||||
| #1 | 2克 | ñ | 播放强度调整的电影 | 玩 | ||||
| #1 | 2H | ñ | 作物图像 | 作物 | ||||
| #1 | 2I | ñ | 旋转图像 | 旋转 | ||||
| #1 | 2J | ñ | 删除整个堆栈中的区域 | 删除区域 | ||||
| #1 | 3A | ÿ | 点击打开“分析窗口”(GUI窗口#2) | 跟踪窗口 | ||||
| #1 | 3B | ÿ | 输入像素的大小(以微米为单位) | 输入像素大小 | ||||
| #2 | 4 | ÿ | 单击以生成叠加单元体的边界/边缘图像 | 边界 | ||||
| #2 | 五 | ÿ | 点击以选择网点的基数和提示 | Referernce | ||||
| #2 | 6A | ÿ | 输入段或节点的数量 | 段数 | ||||
| #2 | 6B | ÿ | 输入扫描长度(垂直于轴) | 扫描宽度 | ||||
| #2 | 6C | ÿ | 输入超过该长度的丝网眼开始弯曲的长度 | 准确测量 | ||||
| #2 | 6D | ÿ | 输入尖端检测圆的半径(即顶点可以在下一帧中进行本地化的区域) | 尖端检测半径 | ||||
| #2 | 6E | ÿ | 输入光栅可以从垂直轴弯曲的最大角度 | 角度阈值 | ||||
| #2 | 1207米 | ñ | 为该特定框架的基准和提示添加参考点 | 选择参考 | ||||
| #2 | 6克 | ñ | 输入长度超过该长度的纤维纹身开始弯曲该特定框架 | 准确测量 | ||||
| #2 | <TD> 6Hñ | 输入该特定帧的检测圆的半径 | 尖端检测半径 | |||||
| #2 | 6I | ñ | 输入特定框架的所有参数后,单击以将值存储到内存和文件以供进一步参考 | 加 | ||||
| #2 | 6J | ñ | 单击以删除该框架的设置手动参数 | 删除 | ||||
| #2 | 6K | ñ | 单击以删除手动存储的所有参数,使用所有帧的“可选功能面板” | 重启 | ||||
| #2 | 6升 | ñ | 在跟踪之前检入以将所有跟踪结果存储在内存中,以备将来参考 | 历史追踪 | ||||
| #2 | 7 | ÿ | 点击开始跟踪 | 跟踪及分析 | ||||
| #2 | 8A | ñ | 点击开始跟踪蛋白质通道强度 | 分析蛋白质浓度 | ||||
| #2 | 8B | ñ | 检查沿轨道长度跟踪蛋白质通道强度 | 全丝酵母 | ||||
| #2 | 8C | ñ | 检查参考蛋白或蛋白A的跟踪 | A蛋白 | ||||
| #2 | 8D | ñ | 检查跟踪蛋白B | ProteinB | ||||
| #2 | 8E | ñ | 检查跟踪蛋白质C | P53复合体蛋白 | ||||
| #2 | 8F | ñ | 检查以跟踪平均蛋白质强度提示 | 领先提示 | ||||
| #2 | 8克 | ñ | 输入提示的长度 | 提示长度 | ||||
| #2 | 8小时 | ñ | 输入从底部开始形成尖端的最小距离 | 阈 | ||||
| #2 | 8I | ñ | 点击将顶尖分析结果保存到文件 | 按钮 | ||||
| #2 | 9A | ñ | 点击启动比率蛋白质分析 | 比较 | ||||
| #2 | 9B | ñ | 入住比较蛋白B相对于A | 日志10(B / A) | ||||
| #2 | 9C | ñ | 入住以比较蛋白C相对于A | 日志10(C / A) | ||||
| #2 | <TD> 9Dñ | 入住以比较蛋白B相对于A的尖端 | 日志10(B / A) | |||||
| #2 | 9E | ñ | 入住以比较蛋白C相对于A的尖端 | 日志10(C / A) | ||||
| #2 | 10A | ñ | 选择其他颜色地图(默认值:Jetplot) | 彩色地图 | ||||
| #2 | 10B | ñ | 编辑色彩图 | 编辑色彩图 | ||||
表1:GUI Windows#1和#2中存在的所有功能概述。
一旦完成,该程序将创建一个凸包,并自动跟踪整个电影中的提示。从电影中提取的参数,比如比例计算器,生长速度和光线长度a重新显示并存储在工作文件夹中作为图像和数据文件。然后可以从存储的数据文件中提取和进一步分析其他参数,如光合生存期,生长速率和收缩率( 图2B )。
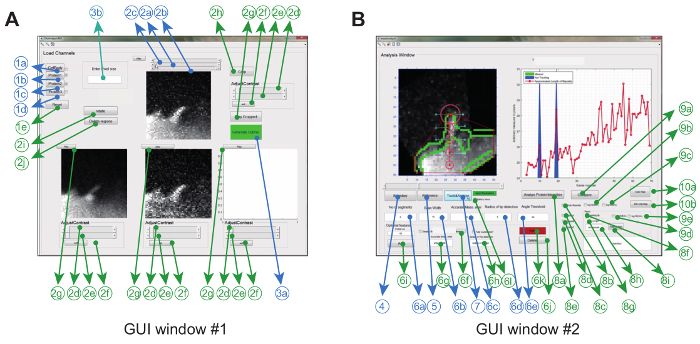
图2:使用图像分析软件的图形用户界面。 ( A )GUI窗口#1用于加载和处理图像。该程序可以加载多达3个蛋白质通道,从而成对比较2个通道。窗口具有强制性(蓝色)和可选功能(绿色),用于在跟踪之前预处理图像( B )GUI窗口2用于跟踪光点以及时空和比率 – 度量蛋白质分析。再次,可选功能标记为绿色。这个数字已经被修改了参考文献12 。 请点击此处查看此图的较大版本。
在这里,我们提供了一个详细的样品制备和软件处理协议。我们从培养细胞和获取优化图像分析的电影的详细说明开始。本部分数据采集之后是操作图像分析软件的详细说明。在整个协议中,我们介绍了在收集和处理数据时应该考虑的关键步骤和可选功能。最后,我们利用图像分析软件分析来自不同模型系统的虚拟障碍,然后将所描述的图像分析软件与可用于虚拟量化的其他程序进行比较,并讨论限制和未来方向。
Protocol
Representative Results
Discussion
在这里,我们提出了一个详细的协议,用于通过凸壳算法跟踪丝状体生长动力学和这些动态结构中相对蛋白质浓度的分析。使用该软件,可以在单次运行中将多达3个通道成对比较,从而在整个延伸/缩回循环中确定两个通道( 即蛋白质)的相对浓度,并将其作为图像和数据文件存储在不同的文件夹中。除了常规操作之外,软件还提供了许多可以进行修改的参数,以优化相应实验的分析。这?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
作者承认了DFG(EXC-1003至MG)的资助。
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
References
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

