واجهة المستخدم الرسومية لتتبع بمساعدة برنامج تركيز البروتين في النتوءات الخلوية الديناميكية
Summary
نقدم حل البرمجيات لتتبع شبه الآلي من تركيز البروتين النسبي على طول نتوءات الخلوية الديناميكية.
Abstract
فيلوبوديا هي ديناميكية، تشبه الإصبع النتوءات الخلوية المرتبطة الهجرة والاتصالات الخلوية الخلية. من أجل فهم أفضل لآليات الإشارات المعقدة الكامنة وراء بدء فيلودوديال، استطالة وتثبيت لاحق أو تراجع، فمن الأهمية بمكان لتحديد النشاط البروتيني الزماني المكاني في هذه الهياكل الحيوية. لتحليل وظيفة البروتين في فيلوبوديا، قمنا مؤخرا بتطوير خوارزمية التتبع شبه الآلي التي تتكيف مع فيلوبوديال شكل التغييرات، مما يسمح تحليل مواز للديناميات نتوء وتركيز البروتين النسبي على طول طول فيلوبوديال كله. هنا، نقدم بروتوكول مفصل خطوة بخطوة لتحسين التعامل مع الخلايا، والحصول على الصور وتحليل البرمجيات. كما نقدم إرشادات لاستخدام الميزات الاختيارية أثناء تحليل الصور وتمثيل البيانات، بالإضافة إلى إرشادات تحري الخلل وإصلاحه لجميع الخطوات المهمة على طول الطريق. وأخيرا، فإننا تشمل أيضا مقارنة دبرنامج تحليل الصور المقتبسة مع البرامج الأخرى المتاحة لتقدير فيلوبوديا. معا، يوفر البروتوكول المقدم إطارا لتحليل دقيق للديناميات البروتين في نتوءات فيلوبوديال باستخدام برنامج تحليل الصور.
Introduction
وترتبط السيطرة المكانية والزمانية للبروتينات التنظيمية أكتين مع ديناميات فيلوبوديوم 1 ، 2 . تتبع مكانيا حل تركيز البروتين على طول طول فيلوبوديال كله من خلال الوقت هو بالتالي حاسمة لتعزيز فهمنا للآليات الكامنة بدء، استطالة، استقرار أو انهيار هذه الهياكل الديناميكية 3 ، 4 . على عكس تحليل البروتين في السيتوسول، حيث تحدث العديد من التغييرات شكل الخلية على نطاق أوسع، فيلوبوديا هي الهياكل الدقيقة الحيوية التي مشبك باستمرار 5 والانحناء، وبالتالي يحول دون تحليل باستخدام نهج بسيط مثل خط المسح الضوئي.
حلول البرمجيات المختلفة لتتبع شكل فيلوبوديال متاحة 6 ، 7 ، 8 ، 9 . Likewأيس، وقد تم تطوير البرمجيات لتتبع راتيوميتريك من ديناميات البروتين داخل الجسم الخلية 10 ، 11 . للجمع بين التتبع الآلي لشكل فيلوبوديال وتحليل البروتين المكاني والزماني، قمنا مؤخرا بتطوير برنامج تحليل الصور على أساس خوارزمية محدبة-بدن 12 . طريقة التحليل الرواية هذه، التي يتم تشغيلها من خلال واجهة المستخدم الرسومية (غوي)، تجمع لأول مرة، تركيز البروتين النسبي على طول طول الفيلوبوديال وسرعة النمو، مما يسمح بالقياس الدقيق لتوزيع البروتين المكاني الزماني المستقل عن حركة هذه الهياكل الديناميكية 12 .
الفكرة وراء البرنامج (شفرة المصدر متاحة بحرية، انظر أدناه) هو أن واحدة من القمم من بدن محدب سوف تتزامن مع غيض من فيلوبوديوم ( الشكل 1A ). من خلال النظر في الإطار اللاحق فوr أقرب قمة الرأس من محدب بدن، ويمكن تتبع طرف متحرك في جميع أنحاء الفيلم كله. مرة واحدة يتم الكشف عن طرف في كل إطار، يتم استخدام موقفها لرسم محور عن طريق الانضمام إلى طرف مع نقطة مرجعية في قاعدة فيلوبوديوم ( الشكل 1B ). وأخيرا، تستخدم النقاط العشرية متساوية البعد، التي يتم تحديد مواقفها من قبل بكسل المتوسط مع أقصى كثافة على طول خط متعامد للمحور، وتستخدم لتحديد العمود الفقري الذي يتبع شكل فيلوبوديال. الاستفادة من هذا العمود الفقري للتكيف، يتم إنشاء كيموجراف لتتبع نمو فيلوبوديال وتركيزات البروتين لمدة تصل إلى ثلاث قنوات على طول طول فيلوبوديال ( الشكل 1C ).
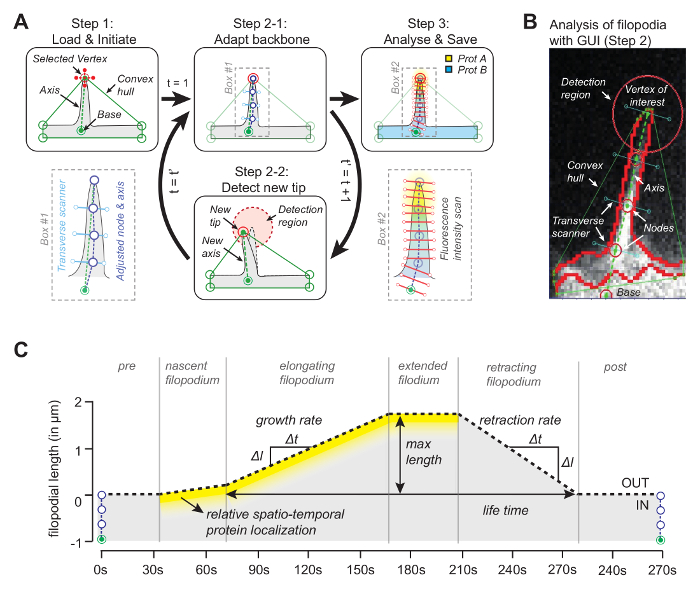
الشكل 1: مبدأ العمل من برامج تحليل الصور. ( A ) الخوارزمية وراءالبرنامج. في STEP1 يحدد المستخدم المرجع (قاعدة) و قمة الرأس (غيض) من فيلوبوديوم. في الخطوة 2-1 يتم الحصول على العمود الفقري للفيلوبوديوم باستخدام بكسل المتوسطة مع أقصى قيمة شدة. في الخطوة 3 يتم استخدام العمود الفقري لملامح كثافة البروتين المكاني. في الخطوة 2-2 يقوم البرنامج بتتبع الطرف تلقائيا في الإطار اللاحق. الإجراء بأكمله يتكرر. ( ب ) لقطة من الخوارزمية مع فيلوبوديوم الحقيقي إدخال عناصر هامة مثل الهيكل المحدب الذي يتم استخدامه للتتبع. ( C ) نظرة عامة على المعلمات التي يمكن قياسها مع الخوارزمية. وقد عدل هذا الرقم من المرجع 12 . الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
يتم تشغيل برنامج تحليل الصور في ماتلاب (المشار إليها باسم برمجة البرمجيات) عن طريق استخدام رسوميةr واجهة. لتحقيق أقصى قدر من المرونة والمتانة لإعداد تجريبي معين، يمكن للمستخدم ضبط سلسلة من المعلمات تتبع (على سبيل المثال يسمح زاوية الانحناء والحركة بين الإطار) وأيضا إجراء بعض التصحيحات على الأفلام ( مثل المحاصيل، والتناوب، وإزالة الأجسام غير المرغوب فيها) ( الشكل 2A والجدول 1) .
| واجهة المستخدم الرسومية | لا. | إلزامي | وصف | الاسم (في واجهة المستخدم الرسومية) | ||||
| # 1 | 1A | Y | تحميل ملف .tiff مكدسة تمثل الجسم الخلية (مع مربع فحص) أو إنشاء جسم خلية فرضه من القنوات | CellBody | ||||
| # 1 | 1B | Y | <td colspan="5"> تحميل ملف صورة مكدسة المقابلة للبروتين 1البروتين 1 | |||||
| # 1 | 1C | Y | تحميل ملف صورة مكدسة المقابلة للبروتين 2 | بروتين 2 | ||||
| # 1 | 1D | Y | تحميل ملف صورة مكدسة المقابلة للبروتين 3 | بروتين 3 | ||||
| # 1 | 1E | N | إعادة تعيين كل شيء لتحميل مسبق ملفات الصور مكدسة | إعادة تعيين | ||||
| # 1 | 2A | Y | شريط التمرير لتحديد الإطار الأولي للتحليل في إطار واجهة المستخدم الرسومية # 2 | NA | ||||
| # 1 | 2B | Y | شريط التمرير لتحديد الإطار النهائي للتحليل في نافذة واجهة المستخدم الرسومية # 2 | NA | ||||
| # 1 | 2C | Y | شريط التمرير يمثل كيرنت الإطار | NA | ||||
| # 1 | 2D | N | القيمة الرمادية للبكسل التي سيتم تعيين جميع وحدات البكسل فيها على الصفر | NA | ||||
| # 1 | 2E | N | القيمة الرمادية للبكسل التي سيتم تعيين جميع وحدات البكسل فيها على القيم القصوى | NA | ||||
| # 1 | 2F | N | عين قيم شدة البكسل المحددة بواسطة <2e> & <2f> | جلس | ||||
| # 1 | 2G | N | تلعب الفيلم تعديل كثافة | لعب | ||||
| # 1 | 2H | N | قص الصوره | ا & قتصاص | ||||
| # 1 | 2I | N | تدوير الصورة | استدارة | ||||
| # 1 | 2J | N | احذف المناطق في المجموعة بأكملها | حذف المناطق | ||||
| # 1 | 3A | Y | انقر لفتح "نافذة التحليل" (نافذة واجهة المستخدم الرسومية رقم 2) | نافذة التتبع | ||||
| # 1 | 3B | Y | أدخل حجم بكسل في ميكرون | أدخل حجم بكسل | ||||
| # 2 | 4 | Y | انقر لتوليد صورة الحدود / حافة الجسم الخلية فرضه | حدود | ||||
| # 2 | 5 | Y | انقر لتحديد قاعدة و غيض من فيلوبوديا | Referernce | ||||
| # 2 | 6A | Y | أدخل عدد الشرائح أو العقد | لا من القطاعات | ||||
| # 2 | 6B | Y | أدخل طول المسح الضوئي (عمودي على المحور) | عرض المسح الضوئي | ||||
| # 2 | 6C | Y | أدخل طول أعلاه الذي فيلوبوديا يبدأ الانحناء | دقيقة دقيقة بعد | ||||
| # 2 | 6D | Y | أدخل نصف قطر الدائرة الكشف عن طرف (أي المنطقة حيث يمكن أن تكون موضعيا قمة الرأس في الإطار التالي) | نصف القطر للكشف عن طرف | ||||
| # 2 | 6E | Y | أدخل أقصى زاوية فيلوبوديوم يمكن أن ينحني من المحور الرأسي | زاوية العتبة | ||||
| # 2 | 6F | N | إضافة نقاط مرجعية للقاعدة والطرف لهذا الإطار المحدد | حدد المرجع | ||||
| # 2 | 6G | N | أدخل طول أعلاه الذي فيلوبوديا يبدأ الانحناء لهذا الإطار محددة | دقيقة دقيقة بعد | ||||
| # 2 | <td> 6HN | أدخل نصف قطر دائرة الكشف عن هذا الإطار المحدد | نصف القطر للكشف عن طرف | |||||
| # 2 | 6I | N | بعد إدخال كافة المعلمات لإطار محدد انقر لتخزين القيم إلى الذاكرة والملف لمزيد من المرجع | إضافة | ||||
| # 2 | 6J | N | انقر لحذف مجموعة المعلمات يدويا لهذا الإطار | حذف | ||||
| # 2 | 6K | N | انقر لحذف جميع المعلمات المخزنة يدويا باستخدام "لوحة الميزات الاختيارية" لجميع الإطارات | إعادة تعيين | ||||
| # 2 | 6L | N | تحقق قبل المتابعة لتخزين جميع نتائج التتبع في الذاكرة للرجوع إليها في المستقبل | تتبع التاريخ | ||||
| # 2 | 7 | Y | انقر لبدء التتبع | تتبع وتحليل | ||||
| # 2 | 8A | N | انقر لبدء تتبع كثافة قناة البروتين | تحليل كثافة البروتين | ||||
| # 2 | 8B | N | تحقق في لتتبع كثافة قناة البروتين على طول طول فيلوبوديال | فيلوبوديا كله | ||||
| # 2 | 8C | N | تحقق في لتتبع البروتين المرجعي أو البروتين A | ProteinA | ||||
| # 2 | 8D | N | تحقق في لتتبع البروتين B | ProteinB | ||||
| # 2 | 8E | N | تحقق في لتتبع البروتين C | ProteinC | ||||
| # 2 | 8F | N | تحقق في لتتبع متوسط كثافة البروتين في ثe تلميح | نصيحة رئيسية | ||||
| # 2 | 8G | N | أدخل طول الطرف | طول النصيحة | ||||
| # 2 | 8H | N | أدخل الحد الأدنى للمسافة من القاعدة أعلاه التي تبدأ غيض تشكيلها | عتبة | ||||
| # 2 | 8I | N | انقر لحفظ نتائج تحليل تلميح الرائدة للملف | اضغط الزر | ||||
| # 2 | 9A | N | انقر لبدء تحليل البروتين راتيوميتريك | قارن | ||||
| # 2 | 9B | N | تحقق في مقارنة البروتين B فيما يتعلق A | LOG10 (B / A) | ||||
| # 2 | 9C | N | تحقق في مقارنة البروتين C فيما يتعلق A | LOG10 (C / A) | ||||
| # 2 | <td> 9DN | تحقق في مقارنة البروتين B فيما يتعلق A في الطرف | LOG10 (B / A) | |||||
| # 2 | 9E | N | تحقق في مقارنة البروتين C فيما يتعلق A في الطرف | LOG10 (C / A) | ||||
| # 2 | 10A | N | اختر خريطة ألوان أخرى (افتراضي: جيتبلوت) | لون خريطة | ||||
| # 2 | 10B | N | عدل كولورماب | تحرير كولورماب | ||||
الجدول 1: نظرة عامة على جميع الوظائف الموجودة في واجهة المستخدم الرسومية ويندوز # 1 و # 2.
مرة واحدة يتم إنجاز هذا، البرنامج بإنشاء بدن محدب وتتبع تلقائيا طرف في جميع أنحاء الفيلم. المعلمات المستخرجة من الفيلم، مثل كيموغراف راتيوميتريك، سرعة النمو، وطول فيلوبوديالإعادة عرض وتخزينها أيضا في مجلد العمل كصور وملفات البيانات. ويمكن بعد ذلك استخراج غيرها من المعالم مثل فيلوبوديال العمر، ومعدل النمو ومعدل التراجع وتحليلها من ملفات البيانات المخزنة ( الشكل 2B ).
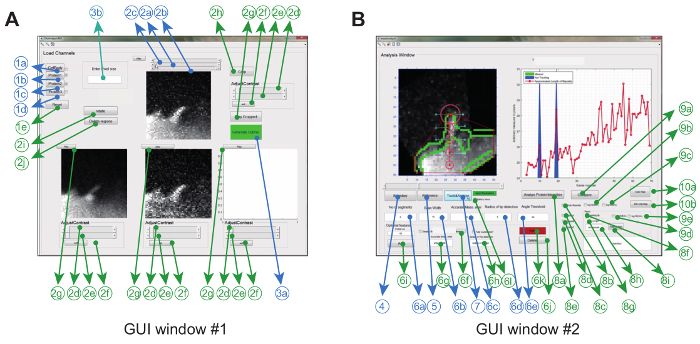
الشكل 2: واجهة المستخدم الرسومية لاستخدام برامج تحليل الصور. ( A ) يستخدم واجهة المستخدم الرسومية نافذة رقم 1 لتحميل الصور ومعالجتها. يمكن للبرنامج تحميل ما يصل إلى 3 قنوات البروتين، حيث يتم مقارنة 2 قنوات زوج من الحكمة. نافذة يأتي مع إلزامية (الأزرق) وميزات اختيارية (الأخضر) لمرحلة ما قبل تجهيز الصور قبل تتبع ( B ) يستخدم واجهة المستخدم الرسومية نافذة # 2 لتتبع فيلوبوديوم وكذلك المكاني الزماني وتحليل نسبة البروتين متري. مرة أخرى، يتم تمييز الميزات الاختيارية باللون الأخضر. وقد كان هذا الرقم معدلاإد من المرجع 12 . الرجاء انقر هنا لعرض نسخة أكبر من هذا الرقم.
هنا، نقدم بروتوكول مفصل لإعداد العينات والتعامل مع البرمجيات. نبدأ مع تعليمات مفصلة حول زراعة الخلايا والحصول على الأفلام الأمثل لتحليل الصور. ويتبع هذا القسم المتعلق باقتناء البيانات وصفا تفصيليا لتشغيل برمجيات تحليل الصور. في جميع أنحاء البروتوكول، ونحن نقدم خطوات حاسمة والميزات الاختيارية التي ينبغي النظر فيها عند جمع ومعالجة البيانات. وأخيرا، فإننا نحلل فيلوبوديا من أنظمة نموذجية مختلفة مع برنامج تحليل الصور، قبل أن يغلق مع مقارنة بين برامج تحليل الصور الموصوفة مع البرامج الأخرى المتاحة للقياس الكمي فيلوبوديال ومناقشة حول القيود والتوجه المستقبلي.
Protocol
Representative Results
Discussion
هنا نقدم بروتوكول مفصل لتتبع ديناميات النمو فيلوبوديال وتحليل تركيزات البروتين النسبية في هذه الهياكل الديناميكية عبر خوارزمية محدبة-بدن. باستخدام البرنامج، يمكن مقارنة ما يصل إلى 3 قنوات زوج الحكمة في تشغيل واحد، حيث يتم تحديد التركيزات النسبية لقناتين ( أي ا?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
يقر المؤلفون بتمويل من دفغ (إكسك-1003 إلى مغ).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
References
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

