Een grafische gebruikersinterface voor software-assisted tracking van eiwitconcentratie in dynamische cellulaire protrusies
Summary
Wij presenteren een software oplossing voor semi-geautomatiseerde tracking van relatieve eiwitconcentratie langs de lengte van dynamische cellulaire uitsteeksels.
Abstract
Filopodia zijn dynamische, vingerachtige cellulaire uitsteeksels die verband houden met migratie en celcelcommunicatie. Om de complexe signaleringsmechanismen die onderliggende filopodiale initiatie, verlenging en daaropvolgende stabilisatie of terugtrekking beter te begrijpen, is het cruciaal om de spatio-temporale eiwitactiviteit in deze dynamische structuren te bepalen. Om de eiwitfunctie in filopodie te analyseren, hebben we onlangs een semi-geautomatiseerd tracking algoritme ontwikkeld dat zich aanpasst aan filopodiale vormveranderingen, waardoor parallelle analyse van protrusiedynamiek en relatieve eiwitconcentratie langs de gehele filopodiale lengte mogelijk is. Hier presenteren we een gedetailleerd stap-voor-stap protocol voor geoptimaliseerde celverwerking, beeldverzameling en software analyse. We geven verder instructies voor het gebruik van optionele functies tijdens beeldanalyse en data weergave, evenals richtlijnen voor het oplossen van problemen voor alle kritische stappen onderweg. Tot slot omvatten we ook een vergelijking van de dBeschadigde beeldanalysesoftware met andere programma's die beschikbaar zijn voor filopodia kwantificering. Samen biedt het gepresenteerde protocol een kader voor nauwkeurige analyse van eiwitdynamiek in filopodiale uitsteeksels met behulp van beeldanalysesoftware.
Introduction
Spatio-temporale controle van actin regulerende eiwitten is geassocieerd met filopodium dynamics 1 , 2 . Het volgen van ruimtelijk opgeloste eiwitconcentratie langs de hele filopodiale lengte door de tijd is dus van cruciaal belang om ons te begrijpen van de mechanismen die onderliggende initiatie, verlenging, stabilisatie of ineenstorting van deze dynamische structuren 3 , 4 liggen . In tegenstelling tot eiwitanalyse in de cytosol, waar veel veranderingen in de celvorming op grotere schaal optreden, zijn filopodia dynamische microstructuren die voortdurend 5 buigen en buigen, waardoor analyse wordt vermeden met behulp van een eenvoudige aanpak, zoals een line-scan.
Verschillende software oplossingen voor het volgen van filopodiale vorm zijn beschikbaar 6 , 7 , 8 , 9 . LikewIse, software voor ratiometrische tracking van eiwitdynamiek in het cellichaam is ontwikkeld 10 , 11 . Om geautomatiseerde tracking van filopodiale vorm en spatio-temporale eiwit analyse te combineren, hebben we onlangs een beeldanalysesoftware ontwikkeld op basis van het convex-rompalgoritme 12 . Deze nieuwe analysemethode, die wordt bediend via een grafische gebruikersinterface (GUI), combineert voor het eerst de relatieve eiwitconcentratie langs de filopodiale lengte en groeitempo, waardoor de nauwkeurige meting van de spatio-temporale eiwitverdeling onafhankelijk van de beweging van deze Dynamische structuren 12 .
Het idee achter de software (broncode is vrij beschikbaar, zie hieronder) is dat een van de hoekpunten van de convexe romp samenvalt met de punt van het filopodium ( figuur 1A ). Door in het volgende frame voor te kijkenR de dichtstbijzijnde hoek van de convex-romp, kan de bewegende punt door de hele film worden gevolgd. Zodra het punt in elk frame is gedetecteerd, wordt de positie ervan gebruikt om een as te trekken door de punt met een referentiepunt aan de basis van het filopodium te koppelen ( Figuur 1B ). Ten slotte wordt gebruik gemaakt van evenwichtige knooppunten, waarvan de posities worden bepaald door de median pixel met maximale intensiteit langs de orthogonale lijn naar de as, om een ruggengraat te bepalen die de filopodiale vorm volgt. Profiteer van deze adaptieve backbone, wordt een kymografie gegenereerd om de filopodiale groei en eiwitconcentraties te detecteren voor maximaal drie kanalen langs de filopodiale lengte ( Figuur 1C ).
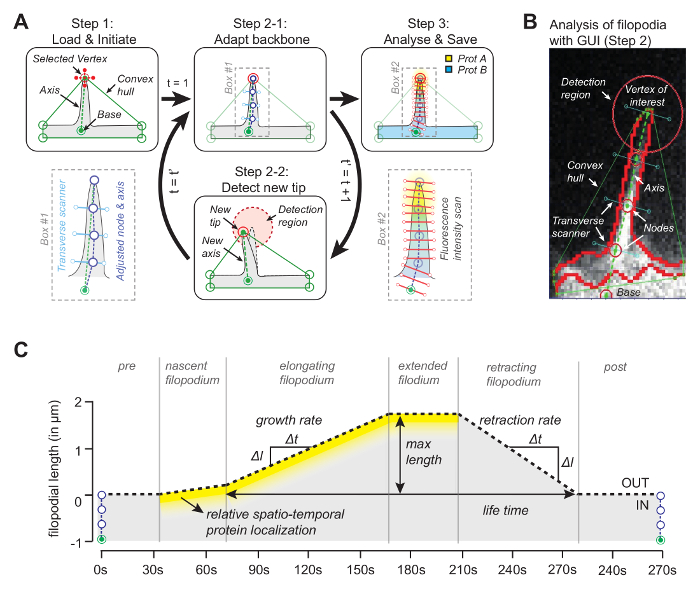
Figuur 1: Werkprincipe van de beeldanalysesoftware. ( A ) Het algoritme achterde software. In stap 1 specificeert de gebruiker de referentie (basis) en de vertex (de punt) van het filopodium. In stap 2-1 wordt de ruggengraat van het filopodium verkregen met behulp van de median pixel met maximale intensiteitswaarde. In stap 3 wordt de ruggengraat gebruikt voor ruimtelijk eiwitintensiteitsprofiel. In stap 2-2 volgt de software automatisch de tip in het volgende frame. De gehele procedure herhaalt. ( B ) Snapshot van het algoritme met echte filopodium waarin belangrijke elementen worden geïntroduceerd, zoals de convexe romp die wordt gebruikt om te volgen. ( C ) Overzicht van parameters die met het algoritme kunnen worden gemeten. Dit cijfer is gewijzigd van referentie 12 . Klik hier om een grotere versie van deze figuur te bekijken.
De beeldanalysesoftware wordt via een grafisch gebruik in Matlab (aangeduid als programmatuursoftware) gebruiktR interface. Om de flexibiliteit en de robuustheid van de betreffende experimentele instelling te maximaliseren, kan de gebruiker een reeks tracking parameters aanpassen ( bijv. Toegestane buighoek en interframe beweging) en ook enkele correcties aan de films maken ( bijv. Bijsnijden, rotatie, verwijdering van ongewenste objecten) ( Figuur 2A en tabel 1) .
| GUI | Nee. | Verplicht | Beschrijving | Naam (in GUI) | ||||
| # 1 | 1a | Y | Het laden van gestapeld .tiff-bestand dat het cellichaam vertegenwoordigt (met een ingevulde doos) of het opgestelde cellichaam van kanalen maken | Cellichaam | ||||
| # 1 | 1b | Y | <td colspan="5"> Gestapeld beeldbestand laden dat overeenkomt met eiwit 1Proteïne 1 | |||||
| # 1 | 1c | Y | Het laden van gestapeld beeldbestand dat overeenkomt met eiwit 2 | Proteïne 2 | ||||
| # 1 | 1d | Y | Het laden van gestapeld beeldbestand dat overeenkomt met eiwit 3 | Proteïne 3 | ||||
| # 1 | 1e | N | Herstelt alles op voorgeladen gestapelde afbeeldingsbestanden | Reset | ||||
| # 1 | 2a | Y | Scrollbalk om het eerste frame voor analyse in GUI-venster # 2 te bepalen | NA | ||||
| # 1 | 2b | Y | Scrollbalk om het laatste frame voor analyse in GUI-venster # 2 te bepalen | NA | ||||
| # 1 | 2c | Y | Scrollbalk voor curreNt frame | NA | ||||
| # 1 | 2d | N | De grijze waarde van de pixels hieronder die alle pixels op nul worden ingesteld | NA | ||||
| # 1 | 2e | N | De grijze waarde van de pixels boven welke alle pixels worden ingesteld op maximale waarden | NA | ||||
| # 1 | 2F | N | Stel de intensiteitswaarden van de pixels die zijn opgegeven door <2e> & <2f> | set | ||||
| # 1 | 2g | N | Speel de intensiteit aangepaste film | Spelen | ||||
| # 1 | 2h | N | Crop Image | Gewas | ||||
| # 1 | 2i | N | Afbeelding roteren | Draaien | ||||
| # 1 | 2j | N | Verwijder regio's in de hele stapel | Verwijder regio's | ||||
| # 1 | 3a | Y | Klik om het 'Analysevenster' te openen (GUI-venster # 2) | Tracking Window | ||||
| # 1 | 3b | Y | Voer de grootte van een pixel in micron in | Voer Pixelformaat in | ||||
| # 2 | 4 | Y | Klik om de grens- / randafbeelding van het bovenliggende cellichaam te genereren | Grens | ||||
| # 2 | 5 | Y | Klik om de basis en de punt van de filopodia te selecteren | Referernce | ||||
| # 2 | 6a | Y | Voer het aantal segmenten of knooppunten in | Aantal segmenten | ||||
| # 2 | 6b | Y | Voer de scanlengte in (loodrecht op de as) | Scanbreedte | ||||
| # 2 | 6c | Y | Voer de lengte in over welke filopodia begint te buigen | Nauwkeurig Meas na | ||||
| # 2 | 6d | Y | Voer de straal in van de tipdetectiecirkel (dwz gebied waar het hoekpunt in het volgende frame kan worden gelokaliseerd) | Radius van puntdetectie | ||||
| # 2 | 6e | Y | Voer de maximale hoek in die het filopodium kan buigen van de verticale as | Hoek drempel | ||||
| # 2 | 6f | N | Voeg referentiepunten toe voor basis en tip voor dat specifieke frame | Selecteer referentie | ||||
| # 2 | 6g | N | Voer de lengte in over welke filopodia voor dat specifieke frame gaat buigen | Nauwkeurig Meas na | ||||
| # 2 | <td> 6hN | Voer de straal van de detectiecirkel voor dat specifieke frame in | Radius van puntdetectie | |||||
| # 2 | 6i | N | Nadat u alle parameters voor het specifieke frame hebt ingevoerd, klikt u om de waarden op te slaan in geheugen en bestand voor verdere verwijzing | Toevoegen | ||||
| # 2 | 6j | N | Klik om de set handmatig parameters voor dat frame te verwijderen | Verwijder | ||||
| # 2 | 6k | N | Klik om alle parameters die handmatig zijn opgeslagen te verwijderen met behulp van het 'optionele functiespaneel' voor alle frames | Reset | ||||
| # 2 | 6L | N | Controleer in voor het volgen om alle trackingresultaten in het geheugen op te slaan voor toekomstige verwijzing | Geschiedenis spoor | ||||
| # 2 | 7 | Y | Klik om te beginnen met volgen | Track & Analyse | ||||
| # 2 | 8a | N | Klik om te beginnen met het opsporen van eiwitkanaalintensiteit | Analyse Protein Intensities | ||||
| # 2 | 8b | N | Controleer de eiwitkanaalintensiteit langs de filopodiale lengte | Hele filopodie | ||||
| # 2 | 8c | N | Inchecken voor het bijhouden van het referentie eiwit of eiwit A | Proteïne | ||||
| # 2 | 8d | N | Inchecken voor het volgen van het eiwit B | ProteinB | ||||
| # 2 | 8e | N | Inchecken voor het volgen van het eiwit C | ProteinC | ||||
| # 2 | 8f | N | Inchecken om de gemiddelde eiwitintensiteit in th te volgenE tip | Leidende tip | ||||
| # 2 | 8g | N | Voer de lengte van de tip in | Tip Lengte | ||||
| # 2 | 8h | N | Voer de minimale afstand van de basis waarboven het punt begint te vormen | drempel | ||||
| # 2 | 8i | N | Klik om de resultaten van de leidende tipanalyses op te slaan in het bestand | Druk op de knop | ||||
| # 2 | 9a | N | Klik om ratiometrische eiwitanalyse te starten | Vergelijken | ||||
| # 2 | 9b | N | Controleer om eiwit B te vergelijken met betrekking tot A | log10 (B / A) | ||||
| # 2 | 9c | N | Controleer om eiwit C te vergelijken met betrekking tot A | log10 (C / A) | ||||
| # 2 | <td> 9dN | Controleer om eiwit B te vergelijken met betrekking tot A bij de tip | log10 (B / A) | |||||
| # 2 | 9e | N | Controleer om eiwit C te vergelijken met betrekking tot A bij de tip | log10 (C / A) | ||||
| # 2 | 10a | N | Kies een andere kleurkaart (standaard: Jetplot) | Kleurkaart | ||||
| # 2 | 10b | N | Bewerk de colormap | Bewerk colormap | ||||
Tabel 1: Overzicht van alle functies aanwezig in de GUI Windows # 1 en # 2.
Zodra dit is bereikt, creëert het programma een convexe romp en volgt de tip automatisch door de film. Parameters die uit de film worden geëxtraheerd, zoals een ratiometrische kymografie, groeisnelheid en filopodiale lengte aOpnieuw weergegeven en ook opgeslagen in de werkmap als afbeeldingen en als gegevensbestanden. Andere parameters zoals levensduur van levensduur, groeipercentage en terugtrekkingsgraad kunnen vervolgens worden geëxtraheerd en verder geanalyseerd uit de opgeslagen gegevensbestanden ( Figuur 2B ).
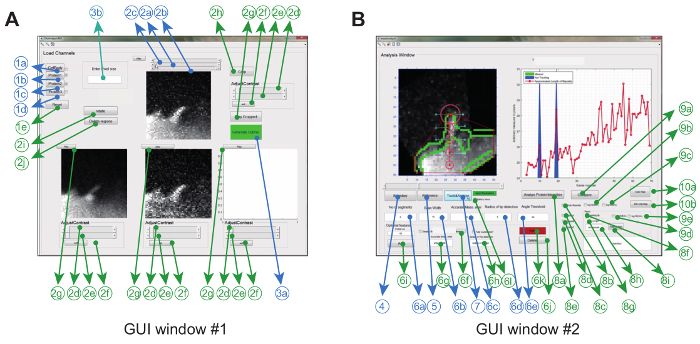
Figuur 2: Grafische gebruikersinterface voor het gebruik van de Image Analysis Software. ( A ) GUI venster # 1 wordt gebruikt voor het laden en verwerken van afbeeldingen. Het programma kan maximaal 3 eiwitkanalen laden, waarbij 2 kanalen in twee verhoudingen worden vergeleken. Het venster verschijnt met verplichte (blauwe) en optionele functies (groen) voor het vooraf verwerken van de afbeeldingen voorafgaand aan het volgen ( B ) GUI Window # 2 wordt gebruikt voor het bijhouden van het filopodium, evenals spatio-temporale en ratio-metrische eiwit analyse. Opnieuw worden optionele functies in groen gemarkeerd. Dit cijfer is gewijzigdUit referentie 12 . Klik hier om een grotere versie van deze figuur te bekijken.
Hier presenteren we een gedetailleerd protocol voor de voorbereiding van het monster en de verwerking van software. We beginnen met gedetailleerde instructies voor het cultiveren van cellen en het verkrijgen van films die zijn geoptimaliseerd voor beeldanalyse. In dit gedeelte over gegevensverzameling wordt een gedetailleerde beschrijving gevolgd voor het bedienen van de beeldanalysesoftware. Tijdens het protocol introduceren we kritieke stappen en optionele functies die in aanmerking moeten worden genomen bij het verzamelen en verwerken van gegevens. Tenslotte analyseren we filopodia uit verschillende modelsystemen met de beeldanalysesoftware, voordat u sluit met een vergelijking van de beschreven beeldanalysesoftware met andere programma's die beschikbaar zijn voor filopodiale kwantificering en een discussie over beperkingen en toekomstige richting.
Protocol
Representative Results
Discussion
Hier presenteren we een gedetailleerd protocol voor het bijhouden van filopodiale groeidynamiek en analyse van relatieve eiwitconcentraties in deze dynamische structuren via het convex-rompalgoritme. Met behulp van de software kunnen maximaal 3 kanalen in een enkele run worden vergeleken, waarbij de relatieve concentraties van twee kanalen (dat wil zeggen eiwitten) door de extensie / terugtrekkingscyclus worden vastgelegd en opgeslagen worden als beeld- en gegevensbestanden in afzonderlijke mappen. Naast de rou…
Disclosures
The authors have nothing to disclose.
Acknowledgements
De auteurs erkennen financiering van de DFG (EXC-1003 naar MG).
Materials
| DMEM | Life Technologies | 31966-021 | |
| 10% Fetal bovine serum | Biochrom AG | L11-044 | |
| Lipofectamine 2000 | Life Technologies | 11668-027 | |
| 1% penicillin/streptomycin | Biochrom AG | 12212 | |
| Neurobasal Medium | Life Technologies | 21103-049 | |
| B27 | Life Technologies | 17504-044 | |
| HEPES (1M stock solution) | Life Technologies | 15630 | |
| Citrine-N1 | Addgene | 54593 | |
| Labtech | Thermo | 155411 | |
| Glutamax-I | Thermo | 35050-061 | |
| Hela | Leibniz Institute DSMZ | ACC-57 | |
| COS 7 | Leibniz Institute DSMZ | ACC-60 | |
| 3T3 cells | Leibniz Institute DSMZ | ACC-59 | |
| Microscope | Nicon Eclipse | ||
| Camera | Andor | DU888 Ultra | |
| Confocal Unit | Yokagawa | CSU-X1 | |
| Pyruvate | Gibco | 31966-021 |
References
- Dunaevsky, A., Tashiro, A., Majewska, A., Mason, C., Yuste, R. Developmental regulation of spine motility in the mammalian central nervous system. Proc Natl Acad Sci U S A. 96 (23), 13438-13443 (1999).
- Matus, A., Brinkhaus, H., Wagner, U. Actin dynamics in dendritic spines: a form of regulated plasticity at excitatory synapses. Hippocampus. 10 (5), 555-560 (2000).
- Galic, M., et al. Dynamic recruitment of the curvature-sensitive protein ArhGAP44 to nanoscale membrane deformations limits exploratory filopodia initiation in neurons. Elife. 3, e03116 (2014).
- Hotulainen, P., et al. Defining mechanisms of actin polymerization and depolymerization during dendritic spine morphogenesis. J Cell Biol. 185 (2), 323-339 (2009).
- Leijnse, N., Oddershede, L. B., Bendix, P. M. Helical buckling of actin inside filopodia generates traction. Proc Natl Acad Sci U S A. 112 (1), 136-141 (2015).
- Tsygankov, D., et al. CellGeo: a computational platform for the analysis of shape changes in cells with complex geometries. J Cell Biol. 204 (3), 443-460 (2014).
- Xiong, Y., et al. Automated characterization of cell shape changes during amoeboid motility by skeletonization. BMC Syst Biol. 4, 33 (2010).
- Styner, M., Gerig, G., Lieberman, J., Jones, D., Weinberger, D. Statistical shape analysis of neuroanatomical structures based on medial models. Med Image Anal. 7 (3), 207-220 (2003).
- Blum, H., Wathen-Dunn, W. A transformation for extracting new descriptors of shape. Models for the Perception of Speech and Visual Form: Proceedings of a Symposium. , 362-380 (1967).
- Barry, D. J., Durkin, C. H., Abella, J. V., Way, M. Open source software for quantification of cell migration, protrusions, and fluorescence intensities. J Cell Biol. 209 (1), 163-180 (2015).
- Machacek, M., et al. Coordination of Rho GTPase activities during cell protrusion. Nature. 461 (7260), 99-103 (2009).
- Saha, T., et al. Automated analysis of filopodial length and spatially resolved protein concentration via adaptive shape tracking. Mol Biol Cell. 27 (22), 3616-3626 (2016).
- Argiro, V., Bunge, M. B., Johnson, M. I. A quantitative study of growth cone filopodial extension. J Neurosci Res. 13 (1-2), 149-162 (1985).
- Mogilner, A., Rubinstein, B. The physics of filopodial protrusion. Biophys J. 89 (2), 782-795 (2005).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat Methods. 2 (12), 905-909 (2005).
- Abramoff, M. D., Magalhaes, P. J., Ram, S. J. Image Processing with ImageJ. Biophotonics International. 11, 36-42 (2004).
- Courtney, J., Woods, E., Scholz, D., Hall, W. W., Gautier, V. W. MATtrack: A MATLAB-Based Quantitative Image Analysis Platform for Investigating Real-Time Photo-Converted Fluorescent Signals in Live Cells. PLoS One. 10 (10), e0140209 (2015).
- Schell, M. J., Erneux, C., Irvine, R. F. Inositol 1,4,5-trisphosphate 3-kinase A associates with F-actin and dendritic spines via its N terminus. J Biol Chem. 276 (40), 37537-37546 (2001).
- Korobova, F., Svitkina, T. Molecular architecture of synaptic actin cytoskeleton in hippocampal neurons reveals a mechanism of dendritic spine morphogenesis. Mol Biol Cell. 21 (1), 165-176 (2010).
- Cheadle, L., Biederer, T. The novel synaptogenic protein Farp1 links postsynaptic cytoskeletal dynamics and transsynaptic organization. J Cell Biol. 199 (6), 985-1001 (2012).
- Tarnok, K., et al. A new tool for the quantitative analysis of dendritic filopodial motility. Cytometry A. 87 (1), 89-96 (2015).
- Hendricusdottir, R., Bergmann, J. H. F-dynamics: automated quantification of dendrite filopodia dynamics in living neurons. J Neurosci Methods. 236, 148-156 (2014).
- Fanti, Z., Martinez-Perez, M. E., De-Miguel, F. F. NeuronGrowth, a software for automatic quantification of neurite and filopodial dynamics from time-lapse sequences of digital images. Dev Neurobiol. 71 (10), 870-881 (2011).
- Costantino, S., et al. Semi-automated quantification of filopodial dynamics. J Neurosci Methods. 171 (1), 165-173 (2008).
- Nilufar, S., Morrow, A. A., Lee, J. M., Perkins, T. J. FiloDetect: automatic detection of filopodia from fluorescence microscopy images. BMC Syst Biol. 7, 66 (2013).

