Ett protokoll för produktionen av integras-brist Lentiviral vektorer för CRISPR/Cas9-medierad gen Knockout i delande celler
Summary
Vi beskriver strategin produktion av integras-brist lentiviral vektorer (IDLVs) som fordon för att leverera CRISPR/Cas9 till celler. Med en förmåga att förmedla snabb och robust gen redigering i celler, IDLVs presentera en säkrare och lika effektiv vektor plattform för gen leverans jämfört med integras-behöriga vektorer.
Abstract
Lentiviral vektorer är ett idealiskt val för att leverera genredigering komponenter till celler på grund av sin kapacitet för stabilt transducing ett brett spektrum av celler och medla höga nivåer av genuttryck. Men deras förmåga att integrera i den mottagande cell arvsmassan ökar risken för insertional mutagenicitet och därmed väcker oro för säkerheten och begränsar deras användning i kliniska inställningar. Ytterligare, långlivade uttryck för genredigering komponenter levereras av dessa integration-behöriga lentiviral vektorer (ICLVs) ökar sannolikheten för promiskuösa gen inriktning. Som ett alternativ, har det utvecklats en ny generation av integras-brist lentiviral vektorer (IDLVs) som tar upp många av dessa farhågor. Här protokollet produktion av en ny och förbättrad IDLV plattform för CRISPR-medierad gen redigering och lista stegen involverade i rening och koncentration av sådana vektorer beskrivs och deras transduktion genredigering effektivitet använder HEK-293T celler visades. Detta protokoll är enkelt skalbar och kan användas för att generera hög titer IDLVs som klarar av transducing celler in vitro och in vivo. Dessutom kan detta protokoll lätt anpassas för produktion av ICLVs.
Introduction
Exakt gen redigering utgör hörnstenen i stora biomedicinsk framsteg som innebär utvecklingen av nya strategier för att hantera genetiska sjukdomar. I spetsen för genredigering teknik är metoden att förlita sig på användningen av det clustered regularly –jagnterspaced short palindromic repeats (CRISPR) / Cas9-system som identifierades inledningsvis som en del av bakteriell immunitet mot invasionen av viral arvsmassa (ses i referenser1,2). En stor fördel av CRISPR/Cas9 systemet över andra genredigering verktyg, såsom zink finger nukleotider (motvilligt) och transkription aktivator-liknande effektor nukleaser (TALENs) (ses i referens3), är den relativa enkelheten i plasmiden design och byggandet av CRISPR komponenter — en funktion som har driv utbyggnaden av genredigering från några specialiserade laboratorier till en mycket bredare forskarsamhället. Enkelheten av CRISPR/Cas9 programmering och dess kapacitet för multiplexering prismalås har dessutom ytterligare underblåst dess popularitet som en kostnadseffektiv och lätt att använda teknik. Bland de olika metoderna tillgängliga för forskare att leverera sådana genredigering komponenter till celler, fortfarande virala vektorer överlägset mest populära och effektiva systemet.
Lentiviral vektorer (LVs) har dykt upp som fordonet val leverera komponenterna i CRISPR/Cas9-system i vivo för applikationsmöjligheter4,5,6,7. Flera viktiga funktioner gör LVs ett populärt val för denna process inklusive deras förmåga att infektera både dela och icke-dividera celler, låg immunogenicitet och minimal cellulär toxicitet (ses i referens8). Som ett resultat, har LV-medierad genterapi varit anställd i behandlingar av smittsamma sjukdomar, som HIV-1, HBV och HSV-1, samt korrigering av defekter underliggande mänskliga ärftliga sjukdomar som cystisk fibros och neo-vaskulär makuladegeneration 4 , 5 , 7 , 9 , 10 , 11. Dessutom LVs effektivt har ändrats för att utföra multiplex gen redigering på distinkt genomisk lokus använder en enda vector systemet12.
Men den inneboende egenskapen hos LVs att integrera i värd genomet kan vara mutagena och ofta svårigheter deras nytta som transgenens leveransfordon, särskilt i kliniska inställningar. Dessutom eftersom stabilt-integrerade LVs express deras transgener på hållbart höga nivåer, är detta system dåligt lämpade för leverans av genredigering komponenter såsom CRISPR/Cas9; överuttryck av Cas9-guide RNA (gärna), och liknande proteiner som motvilligt, är associerade med förhöjda nivåer av off-effekter, som inkluderar oönskade mutationer13,14,15,16 , 17 och kan potentiellt öka cytotoxiciteten18. För att uppnå exakt genredigering med minimal off-target effekter, det är därför absolut nödvändigt att konstruera system som möjliggör övergående uttrycket av genen redigering komponenter.
Under de senaste åren har en mängd olika leveransplattformar utvecklats för att övergående express CRISPR/Cas9 i celler16,19,20,21 (ses i referens22). Dessa inkluderar metoder som förlitar sig på direkt införa renat Cas9 tillsammans med lämplig guide RNASEN i celler, vilket visade sig vara mer effektiva på riktade genredigering jämfört med plasmidmedierad transfection16. Studier har visat att ribonukleoprotein (RNP) komplex bestående av guide RNA/Cas9 partiklar vänds snabbt efter medla DNA klyvning på sina mål, vilket tyder på att kortsiktiga uttryck av dessa komponenter är tillräckligt för att uppnå robust genen redigering16. Möjligen, icke-integrering virala vektorn plattformar såsom adeno-associerade virala vektorer (AAVs) kan ge ett lönsamt alternativ för att leverera genredigering maskiner till celler. Tyvärr AAV förhoppningsvis ha betydligt lägre förpackning kapacitet än LVs (< 5kb), som allvarligt hämmar deras förmåga att paketera flera komponenter CRISPR toolkit inom en enda vektor (ses i referens8). Det är värt att notera att tillägg av föreningar som hämmar Histon deacetylases (e.g., natrium butyrate23) eller hindra cellcykeln (t.ex., koffein24) har visat sig öka lentiviral titrar. Trots de senaste framsteg hindras de övergående uttryck system utvecklat hittills fortfarande av flera brister, såsom lägre produktionseffektivitet, vilket leder till minskad viral titrar och låg transduktion effektiviteten av de virus som genereras genom sådana strategier25.
Integras-brist lentiviral vektorer (IDLVs) utgör ett stort framsteg i utvecklingen av gen-leverans fordon, eftersom de kombinerar LVs förpackning förmåga med fördelen av AAV-liknande episomal underhåll i celler. Dessa funktioner hjälpa IDLVs till stor del kringgå de viktiga frågor i samband med att integrera vektorer, vis-à-vis kontinuerlig överuttryck av potentiellt genotoxiska element och integration-medierad mutagenicitet. Det visades tidigare att IDLVs framgångsrikt kan ändras för att förbättra episomal gen uttryck26,27. När det gäller IDLV-medierad CRISPR/Cas9 leverans begränsar låg produktion titrar och lägre uttryck för episome-burna genomen i förhållande till integras-kunnig lentiviral system deras användbarhet som bona fide verktyg för att leverera editering transgena konstruktioner. Vi har nyligen visat att både transgenens uttryck och viral titrarna är associerade med IDLV produktionen har förbättrats avsevärt genom införandet av bindande platser för Transkriptionfaktorn Sp1 i viral uttryck kassett28. De modifierade IDLVs stöds kraftfullt CRISPR-medierad gen redigering både in vitro- (i HEK-293T celler) och i vivo (i efter mitotiska hjärnans nervceller), medan inducerande minimal off-target mutationer jämfört med motsvarande ICLV-medierad system28. Sammantaget utvecklade vi en roman, kompakt, allt-i-ett CRISPR toolkit transporteras på en IDLV plattform och beskrivs de olika fördelarna med att använda sådant leveransfordon för förbättrade genen redigering.
Här är protokollet produktion av IDLV-CRISPR/Cas9 systemet beskrivs, inklusive de olika steg som ingår i församlingen, rening, koncentration, och titrering av IDLVs, samt strategier för att validera genredigering effekten av dessa vektorer. Detta protokoll är enkelt skalbart för att möta behoven hos olika undersökare och är utformad att framgångsrikt generera LV vektorer med titrar i spänna av 1 x 1010 transducing enheter (TU) / mL. De vektorer som genereras genom detta protokoll kan utnyttjas för att effektivt angripa flera olika celltyper, däribland svårt-att-transduce embryonala stamceller, hematopoetiska celler (T-celler och makrofager) och odlade och in vivo– injicerade nervceller. Protokollet är dessutom lika väl lämpad för produktion av integras-behöriga lentiviral vektorer i liknande kvantiteter.
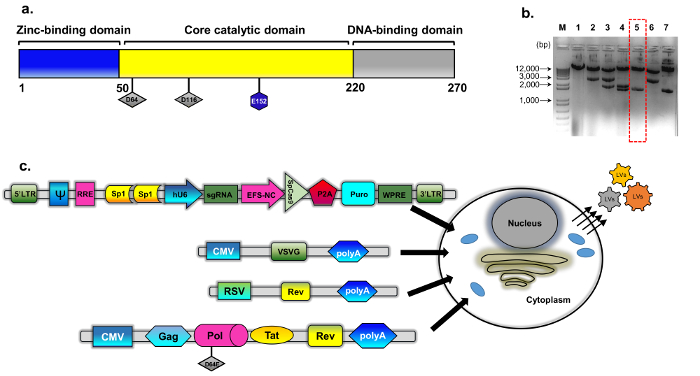
Figur 1: IDLV förpackningar. (a) Schematisk bild av vildtyp integras protein (b) modifierad plasmiden härleddes från psPAX2 (se metoder, plasmid konstruktion för detaljer). Representant agaros gel bild av kloner screenas för muterade integras kloner. DNA-prover som tillagas med ett mini-kit med standard plasmid-DNA med isolering analyserades av matsmältningen med EcoRV och SphI. Korrekt rötas klonen (nummer 5, streckad röd ruta) bekräftades ytterligare genom direkt (Sanger) sekvensering för D64E substitution i INT. Integras-bristfällig förpackning kassetten hette pBK43. (c) Schematisk av protokollet övergående transfection anställd för att generera IDLV-CRISPR/Cas9 vektorer, visar 293T celler transfekterade med VSV-G, förpackningar och transgenens kassetter (Sp1-CRISPR/Cas9 allt-i-ett plasmid). Viruspartiklar som bud ut från cellmembranet innehåller fullängds RNA av vektor (uttryckt från transgenens kassett). Den andra generationen av systemets IDLV-förpackning användes, vilket inkluderar de reglerande proteinerna Tat och Rev. Rev uttryck kompletteras vidare från en separat kassett (RSV-REV-plasmid). ABBREV: LTR-lång-terminal upprepa, VSV-G, vesikulär stomatit virus G-protein, pCMV-cytomegalovirus arrangören; Rous sarkom virus (RSV) arrangören; RRE-(Rev svar Element). Andra reglerande element på uttrycket kassetten omfattar Sp1-bindande platser, Rev Response element (RRE), murmeldjur hepatit Virus från föreskrivande Element (WPRE), en core-töjning faktor 1α promotor (EFS-NC), vektor förpackningen elementet ψ (psi), human Cytomegalovirus (hCMV) arrangören och mänskliga U6 arrangören. Klicka här för att se en större version av denna siffra.
Protocol
Representative Results
Discussion
IDLVs har börjat dyka upp som fordonet val för i vivo genredigering, särskilt inom ramen för genetiska sjukdomar, till stor del på den låga risken för mutagenicitet associerade med dessa vektorer jämfört med att integrera leverans plattformar22 , 28. i den nuvarande manuskriptet, vi försökte detalj i protokollet som är associerade med produktionen av det förbättra allt-i-ett IDLV-CRISPR/Cas9-system som har nyligen utvecklats i vårt laborat…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Vi vill tacka Institutionen för neurobiologi, Duke University School of Medicine och fakultetskansli för grundläggande vetenskap, Duke University. Vi tackar också medlemmar av hertig Viral vektor kärnan för synpunkter på manuskriptet. Plasmiden pLenti CRISPRv2 var gåva från Feng Zhang (Broad Institute). LV-förpackning systemet inklusive den plasmider psPAX2, var VSV-G, pMD2.G och pRSV-Rev en slags gåva från Didier Trono (EPFL, Schweiz). Ekonomiskt stöd för detta arbete ges av den University i South Carolina School Of Medicine, bevilja RDF18080-E202 (Bloom).
Materials
| Equipment | |||
| Optima XPN-80 Ultracentrifuge | Beckman Coulter | A99839 | |
| Allegra 25R tabletop centrifuge | Beckman Coulter | 369434 | |
| xMark Microplate Absorbance plate reader | Bio-Rad | 1681150 | |
| BD FACS | Becton Dickinson | 338960 | |
| Inverted fluorescence microscope | Leica | DM IRB2 | |
| 0.45-μm filter unit, 500mL | Corning | 430773 | |
| Conical bottom ultracentrifugation tubes | Seton Scientific | 5067 | |
| Conical tube adapters | Seton Scientific | PN 4230 | |
| SW32Ti swinging-bucket rotor | Beckman Coulter | 369650 | |
| 15 mL conical centrifuge tubes | Corning | 430791 | |
| 50mL conical centrifuge tubes | Corning | 430291 | |
| High-binding 96-well plates | Corning | 3366 | |
| 150 mm TC-Treated Cell Culture dishes with 20 mm Grid | Corning | 353025 | |
| 100mm TC-Treated Culture Dish | Corning | 430167 | |
| 0.22 μM filter unit, 1L | Corning | 430513 | |
| QIAprep Spin Miniprep Kit (50) | Qiagen | 27104 | |
| Tissue culture pipettes, 5 mL | Corning | 4487 | |
| Tissue culture pipettes, 10 mL | Corning | 4488 | |
| Tissue culture pipettes, 25 mL | Corning | 4489 | |
| Hemacytometer with cover slips | Cole-Parmer | UX-79001-00 | |
| Name | Company | Catalog Number | Comments |
| Cell culture reagents | |||
| Human embryonic kidney 293T (HEK 293T) cells | ATCC | CRL-3216 | |
| 293FT cells | Thermo Fisher Scientific | R70007 | |
| DMEM, high glucose media | Gibco | 11965 | |
| Cosmic Calf Serum | Hyclone | SH30087.04 | |
| Antibiotic-antimycotic solution, 100X | Sigma Aldrich | A5955-100ML | |
| Sodium pyruvate | Sigma Aldrich | S8636-100ML | |
| Non-Essential Amino Acid (NEAA) | Hyclone | SH30087.04 | |
| RPMI 1640 media | Thermo Fisher Scientific | 11875-085 | |
| GlutaMAX | Thermo Fisher Scientific | 35050061 | |
| Trypsin-EDTA 0.05% | Gibco | 25300054 | |
| BES (N, N-bis (2-hydroxyethyl)-2-amino-ethanesulfonic acid) | Sigma Aldrich | B9879 – BES | |
| Gelatin | Sigma Aldrich | G1800-100G | |
| Name | Company | Catalog Number | Comments |
| p24 ELISA reagents | |||
| Monoclonal anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | 3537 | |
| HIV-1 standards | NIH AIDS Research and Reference Reagent Program | SP968F | |
| Polyclonal rabbit anti-p24 antibody | NIH AIDS Research and Reference Reagent Program | SP451T | |
| Goat anti-rabbit horseradish peroxidase IgG | Sigma Aldrich | 12-348 | Working concentration 1:1500 |
| Normal mouse serum, Sterile, 500mL | Equitech-Bio | SM30-0500 | |
| Goat serum, Sterile, 10mL | Sigma | G9023 | Working concentration 1:1000 |
| TMB peroxidase substrate | KPL | 5120-0076 | Working concentration 1:10,000 |
| Name | Company | Catalog Number | Comments |
| Plasmids | |||
| psPAX2 | Addgene | 12260 | |
| pMD2.G | Addgene | 12259 | |
| pRSV-Rev | Addgene | 12253 | |
| lentiCRISPR v2 | Addgene | 52961 | |
| Name | Company | Catalog Number | Comments |
| Restriction enzymes | |||
| BsrGI | New England Biolabs | R0575S | |
| BsmBI | New England Biolabs | R0580S | |
| EcoRV | New England Biolabs | R0195S | |
| KpnI | New England Biolabs | R0142S | |
| PacI | New England Biolabs | R0547S | |
| SphI | New England Biolabs | R0182S |
References
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Marraffini, L. A., Sontheimer, E. J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea. Nat Rev Genet. 11 (3), 181-190 (2010).
- Kim, H., Kim, J. S. A guide to genome engineering with programmable nucleases. Nat Rev Genet. 15 (5), 321-334 (2014).
- Bellec, J., et al. CFTR inactivation by lentiviral vector-mediated RNA interference and CRISPR-Cas9 genome editing in human airway epithelial cells. Curr Gene Ther. 15 (5), 447-459 (2015).
- Kennedy, E. M., et al. Suppression of hepatitis B virus DNA accumulation in chronically infected cells using a bacterial CRISPR/Cas RNA-guided DNA endonuclease. Virology. 476, 196-205 (2015).
- Roehm, P. C., et al. Inhibition of HSV-1 Replication by Gene Editing Strategy. Sci Rep. 6, 23146 (2016).
- Wang, W., et al. CCR5 gene disruption via lentiviral vectors expressing Cas9 and single guided RNA renders cells resistant to HIV-1 infection. PLoS One. 9 (12), e115987 (2014).
- Kantor, B., Bailey, R. M., Wimberly, K., Kalburgi, S. N., Gray, S. J. Methods for gene transfer to the central nervous system. Adv Genet. 87, 125-197 (2014).
- Lebbink, R. J., et al. A combinational CRISPR/Cas9 gene-editing approach can halt HIV replication and prevent viral escape. Sci Rep. 7, 41968 (2017).
- Xu, L., et al. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. , (2017).
- Yiu, G., Tieu, E., Nguyen, A. T., Wong, B., Smit-McBride, Z. Genomic Disruption of VEGF-A Expression in Human Retinal Pigment Epithelial Cells Using CRISPR-Cas9 Endonuclease. Invest Ophthalmol Vis Sci. 57 (13), 5490-5497 (2016).
- Kabadi, A. M., Ousterout, D. G., Hilton, I. B., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome engineering from a single lentiviral vector. Nucleic Acids Res. 42 (19), e147 (2014).
- Fu, Y., Sander, J. D., Reyon, D., Cascio, V. M., Joung, J. K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat Biotechnol. 32 (3), 279-284 (2014).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol. 31 (9), 827-832 (2013).
- Kim, S., Kim, D., Cho, S. W., Kim, J., Kim, J. S. Highly efficient RNA-guided genome editing in human cells via delivery of purified Cas9 ribonucleoproteins. Genome Res. 24 (6), 1012-1019 (2014).
- Pattanayak, V., et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol. 31 (9), 839-843 (2013).
- Shalem, O., et al. Genome-scale CRISPR-Cas9 knockout screening in human cells. Science. 343 (6166), 84-87 (2014).
- Schaefer, K. A., et al. Unexpected mutations after CRISPR-Cas9 editing in vivo. Nat Methods. 14 (6), 547-548 (2017).
- Choi, J. G., et al. Lentivirus pre-packed with Cas9 protein for safer gene editing. Gene Ther. 23 (7), 627-633 (2016).
- Platt, R. J., et al. CRISPR-Cas9 knockin mice for genome editing and cancer modeling. Cell. 159 (2), 440-455 (2014).
- Truong, D. J., et al. Development of an intein-mediated split-Cas9 system for gene therapy. Nucleic Acids Res. 43 (13), 6450-6458 (2015).
- Nelson, C. E., Gersbach, C. A. Engineering Delivery Vehicles for Genome Editing. Annu Rev Chem Biomol Eng. 7, 637-662 (2016).
- Jaalouk, D. E., Crosato, M., Brodt, P., Galipeau, J. Inhibition of histone deacetylation in 293GPG packaging cell line improves the production of self-inactivating MLV-derived retroviral vectors. Virol J. 3, 27 (2006).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Hoban, M. D., et al. Delivery of Genome Editing Reagents to Hematopoietic Stem/Progenitor Cells. Curr Protoc Stem Cell Biol. 36, 1-10 (2016).
- Bayer, M., et al. A large U3 deletion causes increased in vivo expression from a nonintegrating lentiviral vector. Mol Ther. 16 (12), 1968-1976 (2008).
- Kantor, B., Ma, H., Webster-Cyriaque, J., Monahan, P. E., Kafri, T. Epigenetic activation of unintegrated HIV-1 genomes by gut-associated short chain fatty acids and its implications for HIV infection. Proc Natl Acad Sci U S A. 106 (44), 18786-18791 (2009).
- Ortinski, P. I., O’Donovan, B., Dong, X., Kantor, B. Integrase-Deficient Lentiviral Vector as an All-in-One Platform for Highly Efficient CRISPR/Cas9-Mediated Gene Editing. Mol Ther Methods Clin Dev. 5, 153-164 (2017).
- Kantor, B., et al. Notable reduction in illegitimate integration mediated by a PPT-deleted, nonintegrating lentiviral vector. Mol Ther. 19 (3), 547-556 (2011).
- Berkhout, B., Verhoef, K., van Wamel, J. L., Back, N. K. Genetic instability of live, attenuated human immunodeficiency virus type 1 vaccine strains. J Virol. 73 (2), 1138-1145 (1999).
- Gomez-Gonzalo, M., et al. The hepatitis B virus X protein induces HIV-1 replication and transcription in synergy with T-cell activation signals: functional roles of NF-kappaB/NF-AT and SP1-binding sites in the HIV-1 long terminal repeat promoter. J Biol Chem. 276 (38), 35435-35443 (2001).
- Kim, Y. S., et al. Artificial zinc finger fusions targeting Sp1-binding sites and the trans-activator-responsive element potently repress transcription and replication of HIV-1. J Biol Chem. 280 (22), 21545-21552 (2005).
- Ortinski, P. I., Lu, C., Takagaki, K., Fu, Z., Vicini, S. Expression of distinct alpha subunits of GABAA receptor regulates inhibitory synaptic strength. J Neurophysiol. 92 (3), 1718-1727 (2004).
- Van Lint, C., et al. Transcription factor binding sites downstream of the human immunodeficiency virus type 1 transcription start site are important for virus infectivity. J Virol. 71 (8), 6113-6127 (1997).
- Van Lint, C., Ghysdael, J., Paras, P., Burny, A., Verdin, E. A transcriptional regulatory element is associated with a nuclease-hypersensitive site in the pol gene of human immunodeficiency virus type 1. J Virol. 68 (4), 2632-2648 (1994).
- Xu, W., Russ, J. L., Eiden, M. V. Evaluation of residual promoter activity in gamma-retroviral self-inactivating (SIN) vectors. Mol Ther. 20 (1), 84-90 (2012).
- . Testing for Replication Competent Retrovirus (RCR)/Lentivirus (RCL) in Retroviral and Lentiviral Vector Based Gene Therapy Products – Revisiting Current FDA Recommendations Available from: https://sites.duke.edu/dvvc/files/2016/05/FDA-recommendation-for-RCR-testing.pdf (2017)
- . Biosafety in Microbiological and Biomedical Laboratories Available from: https://www.cdc.gov/biosafety/publications/bmbl5/bmbl.pdf (2017)
- Dull, T., et al. A third-generation lentivirus vector with a conditional packaging system. J Virol. 72 (11), 8463-8471 (1998).
- Liu, X. S., et al. Editing DNA Methylation in the Mammalian Genome. Cell. 167 (1), 233-247 (2016).
- Ran, F. A., et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature. 520 (7546), 186-191 (2015).
- Zetsche, B., et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 163 (3), 759-771 (2015).

