Oligomerization dynamik af celleoverflade receptorer i levende celler ved total intern refleksion Fluorescens mikroskopi kombineret med tal og lysstyrke analyse
Summary
Vi beskriver en billedbehandlings metode til bestemmelse af den gennemsnitlige oligomeriske tilstand af mEGFP-mærkede receptor-oligomerer induceret af ligand binding i plasma membranen af levende celler. Protokollen er baseret på total intern refleksion fluorescens (TIRF) mikroskopi kombineret med tal og lysstyrke (N & B) analyse.
Abstract
På trods af betydningen og allesteds nærhed af receptor oligomerisering, få metoder er gældende for påvisning klyngedannelse begivenheder og måling af graden af klyngedannelse. Her beskriver vi en Imaging tilgang til at bestemme den gennemsnitlige oligomeriske tilstand af mEGFP-mærkede-receptor homokomplekser i membranen af levende celler. Protokollen er baseret på total intern refleksion fluorescens (TIRF) mikroskopi kombineret med tal og lysstyrke (N & B) analyse. N & B er en metode, der svarer til fluorescens-korrelations spektroskopi (FCS) og foton-optællings histogram (PCH), som er baseret på den statistiske analyse af svingningerne i fluorescens intensitet af fluorophorer, der spredes ind og ud af en belysning volumen i løbet af observationstiden. Navnlig er N & B en forenkling af PCH for at indhente oplysninger om det gennemsnitlige antal proteiner i oligomeriske blandinger. Amplituder for intensitets udsving er beskrevet af fluoroforet molekyle lysstyrke og det gennemsnitlige antal fluorophorer inden for belysnings volumenet. N & B betragter således kun første og andet øjeblik af amplitude fordelingen, nemlig den gennemsnitlige intensitet og variansen. Dette er på samme tid, styrken og svagheden af metoden. Da der kun tages hensyn til to øjeblikke, kan N & B ikke bestemme den molære fraktion af ukendte oligomerer i en blanding, men den anslår kun den gennemsnitlige oligomeriserings tilstand for blandingen. Ikke desto mindre kan det anvendes til relativt små tidsserier (sammenlignet med andre moment metoder) af billeder af levende celler på en pixel-for-pixel basis, blot ved at overvåge tids udsving i fluorescens intensitet. Det reducerer den effektive time-per-pixel til et par mikrosekund, tillader erhvervelse i tidsinterval af sekunder til millisekunder, hvilket er nødvendigt for hurtig oligomerisering kinetik. Endelig kan store celleområder såvel som sub-cellulære rum udforskes.
Introduction
Vi beskriver en samlet intern refleksion fluorescens-antal og lysstyrke (TIRF-N & B) Imaging tilgang til bestemmelse af den gennemsnitlige oligomeriske tilstand af receptor molekyler i plasma membranen af levende celler, sigter mod at forbinde receptoren forsamling dynamik til proteinerne biologiske funktion (figur 1).
Ved ekstracellulær ligand binding initierer receptorer det intracellulære signal transduktion afhængigt af deres kropsbygning, oligomerisering, potentielle Co-receptorer og membran sammensætning. På trods af betydningen og allesteds nærende receptor oligomerisering, anerkendt som en vigtig begivenhed i cellulære signalering1,2,3,4,5,6, 7, få metoder kan detektere klyngedannelse begivenheder og måle graden af klyngedannelse eksperimentelt8,9. Den confokale volumen (x, y ≈ 300 Nm, z ≈ 900 nm) er utilstrækkeligt løst til at bevise molekylære interaktion og støkiometri, selv efter optimering af billedet restaurering algoritmer10. Under enheds sammensætningen af protein oligomerer kan ikke løses på et rent geografisk grundlag, selv ved super opløsnings metoder ved x, y-opløsning på 20-70 nm såsom PALM11, storm12og sted13. Desuden kan deres tidsmæssige opløsning (i rækkefølgen af minutter pr. billede) ikke følge kinetikken i intervallet fra sekunder. Enkelt molekyle Step-blegning løser kun støkiometri af protein oligomerer, hvis de er immobile14.
En af de mest alsidige metoder til at måle tæthed og oligomerisering af fluorescently mærkede proteiner inden for enkeltbilleder er den rumlige intensitets fordelings analyse (SpIDA), som er afhængig af rumlig prøvetagning. Det gælder både kemisk faste og levende celler, og gør det muligt at analysere flere områder af interesse for cellen samtidig ved hjælp af standard Fluorescens mikroskopi15. Alternativt, moment metoder, såsom fluorescens-korrelations spektroskopi (FCS)16, photon Counting histogram (PCH)17, og tal og lysstyrke (N & B)18,19, er egnet til kvantitativ oligomer Målinger. Disse metoder analyserer fluorescens intensitets udsving, der kan observeres i tide, når fluorophorerne diffus ind og ud af en belysnings volumen. Amplituder af intensitets svingningerne kan entydigt beskrives ved den molekylære lysstyrke af fluoroforet (ε) og det gennemsnitlige antal fluorophorer (n) i belysnings volumenet17 (figur 2). Typisk kan diffusions koefficienten for fluorophorerne og det gennemsnitlige antal molekyler (omvendt relateret til G (0)-værdien) inden for belysnings volumenet opnås af FCS20. Men da diffusions tiden kun skalerer med den kubiske rod af massen, er FCS ikke tilstrækkelig følsom til at påvise ændringer i molekylmasse21. I praksis kan en enkelt farve FCS ikke detektere dimerisering af membranreceptorer. PCH løser blandinger af forskellige oligomerer nøjagtigt. Ved hjælp af mere end to øjeblikke af amplitude fordelingen, det registrerer molekyler af forskellig lysstyrke, der indtager den samme belysning volumen. Scanning FCS22 og udviklinger, såsom den interessante par-korrelation af Molekylær lysstyrke (pcomb) tilgang23, indført for at udvide anvendelsesområdet for fluorescens korrelations metoder i biologiske systemer24 , forblive enkelt punkt metoder, der mangler evnen til hurtige målinger i et stort område af en celle, kræver mange på hinanden følgende observationer på hver pixel og data erhvervelse i rækkefølgen af sekunder.
N & B er en forenklet version af PCH, der kun betragter det første og andet øjeblik af amplituden i fluorescens fordelingen, nemlig den gennemsnitlige intensitet, , og variansen, σ2 (figur 2)18,19 og på grund af dette, kan den ikke bestemme den molære fraktion af ukendte oligomerer i en blanding, men anslår kun den gennemsnitlige oligomeriserings tilstand af blandingen. Ikke desto mindre har N & B den fordel at arbejde med relativt mindre tidsserier af billeder af levende celler end PCH på en pixel-for-pixel basis, blot ved at overvåge udsving på tidspunktet for fluorescens intensitet. Da N & B reducerer tiden pr. pixel til nogle få mikrosekunder, kan den følge en hurtig oligomeriserings kinetik over store celleområder, hvilket giver mulighed for at erhverve billeder på en tidsskala i sekunder i mikroskopi af raster scanning (f. eks. confocal, 2-photon) og millisekunder i Kamerabaseret mikroskopi (f. eks. TIRFM).
Flere rapporter har vist, at N & B er i stand til at kvantificere antallet af under enheder i protein klynger ved at afbilde udvidede celleområder. Der blev fundet paxillin-EGFP-klynger på vedhæftnings stederne i CHO-K1-cellerne25, og den intracellulære sammenlægning af det patogene Httex1p peptid blev beskrevet i cos-7-cellerne26. N & B blev ansøgt om at følge den ligand-drevne oligomerisering af ErbB-receptoren27og virkningen af ligand-FGF21 på Klothob (klb) og FGFR1c i Hela-cellerne28. Kombinationen af TIRF Imaging og N & B analyse blev brugt til at vise, at dynamin-2 primært er tetrameric i hele cellemembranen29. Vi anvendte N & B til både raster scanning og tirf billeder til at bevise ligand-drevet denne af uPAR og FGFR1 celle membranreceptorer30,31.
Fluorescens korrelations metoder, såsom N & B, FCS og PCH, er baseret på forestillingen om, at antallet af partikler i et åbent volumen følger en Poisson-fordeling. Da kun de fotoner, som fluorophorerne udsender, kan påvises, er middelværdien for en målt fluorescens intensitet versus tid i en pixel i billedet  , produktet af det gennemsnitlige antal fluorophorer i belysnings volumen n og deres Molekylær lysstyrke, ε17:
, produktet af det gennemsnitlige antal fluorophorer i belysnings volumen n og deres Molekylær lysstyrke, ε17:

hvor ε udtrykkes som antallet af fotoner, der udsendes pr. tidsenhed (konventionelt pr. sekund) pr. molekyle, når molekylet er i midten af belysnings volumenet.
Lysstyrke er en egenskab for hver fluoroforet i en given erhvervelse oprettet, mens intensitet er summen af alle bidrag fra alle fluorophorer. I biologiske konkurrencer, vil lysstyrken stige med stigningen i antallet af fluorophorer, der svinger sammen, giver oplysninger om oligomerisering tilstand af fluorescently-mærkede protein. Udsvingene amplituder ved en given pixel måles fra variansen af fluorescens signalet, σ2:

Hvor middelværdien af kvadratet af intensitet  ,, og kvadratet af middelværdien af
,, og kvadratet af middelværdien af  intensitet, er beregnet ud fra de individuelle intensitetsværdier i hver pixel i hver ramme:
intensitet, er beregnet ud fra de individuelle intensitetsværdier i hver pixel i hver ramme:
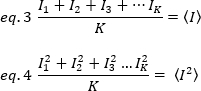
hvor K er antallet af samlede rammer i tidsserierne. Eksperimentelt er det nødvendigt at beregne variansen for hele billedserien, der beskriver spredningen af de individuelle intensitetsværdier ved hver pixel i et enkelt billede omkring middelintensitets værdien. Variansen omfatter alle udsving af forskellig oprindelse. Ved første tilnærmelse kan variansen på grund af sprede-partiklerne i belysnings volumenet, σ20, adskilles fra variansen på grund af detektor støjen, σ2d. De to afvigelser er uafhængige; således, den samlede varians er givet ved deres sum:

Variansen på grund af molekylære udsving i og ud af detekterings volumenet er lineært afhængigt af den molekylære lysstyrke og intensitet:

Omarrangering af EQ. 6 ifølge EQ. 1:

Ifølge den typiske koncept i fluorescens korrelations spektroskopi, ligning 7 hedder det, at variansen på grund af antallet af udsving afhænger af kvadratet af partikel lysstyrken.
Variansen på grund af detektor udsving er derefter en lineær funktion af den detekterede intensitet under den antagelse, at detektoren drives under dens mætningsgrænse19:

I tilfælde af foton tælle detektorer a= 1 og c= 0, således detektor variansen er lig med den gennemsnitlige intensitet:

For at anvende disse begreber til reelle målinger i levende celler, Gratton og kolleger18 definere den tilsyneladende lysstyrke, B, for hver pixel som forholdet mellem variansen over den gennemsnitlige intensitet:

B er den parameter, der måles eksperimentelt. I dette arbejde, tidsserie billeder af FGFR1 receptorer på plasma membranen af HeLa celler er fanget af TIRF mikroskopi og den gennemsnitlige tilsyneladende lysstyrke, B, bestemmes af N & B analyse. Derefter, efter tilsætning af FGF2, sammenhængende tidsserier er fanget for at følge ændringerne i den selv-samling af receptor molekyler i membranen overflade efter stimulation af receptoren med den kanoniske ligand.
Men da detektoren af TIRF mikroskop er et EMCCD-kamera, skal udtrykket for den tilsyneladende lysstyrke ændres som19:

hvor offset er den intensitets forskydning af detekterings elektronikken, der er karakteristisk for detektor indstillingerne. Variansen og den gennemsnitlige intensitet for en analog detektor angives henholdsvis ved:
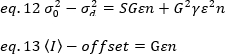
hvor G er analog gevinst i digitale niveauer (DL/photons), S, de digitale niveauer per photon19, er givet ved hældningen af en intensitet versus varians plot for en lyskilde med konstant intensitet (ingen tidsmæssige udsving). Γ-faktoren er relateret til form af pixel detekterings volumen. Ifølge Hassler et al.32er γ faktoren lig med 0,3 for tirf-billeddannelse, der arbejder ved den maksimale gevinst ved detekterings kameraet19. Parametrene offset, S og G er egenskaber for kameraet og mikroskopet. Den tilsyneladende lysstyrke, B, opnås ved at omarrangere EQ. 11 i henhold til EQ. 12 og 13:

Eksperimentelt er ε en kompleks funktion af laser intensitet og systemets detekterings effektivitet. Da B/S ikke desto mindre er lineært afhængig af ε, er det kun vigtigt at bestemme den relative værdi af ε for en given detekterings tilstand:

hvor ε ‘ er proportional med ε. Alligevel udføres en kalibrering ved hjælp af en intern reference.
Protocol
Representative Results
Discussion
N & B kræver flere forsigtighedsregler i valget af celle model og mærknings strategi. Den kan kun anvendes på levende celler, der forbliver stabilt overholdt under billed optagelsestiden. Ekstra udsving på grund af hele cellens stive forskydning kan håndteres med passende billede restaurering tilgange38. Men generelt, når en celle bevæger sig, cellemembranen også deforme, og struktur deformation, producerer store ekstra varians, introducerer alvorlig begrænsning til analyse af membran pro…
Disclosures
The authors have nothing to disclose.
Acknowledgements
CNIC støttes af Ministeriet for Ciencia, Innovacion y Universidades og Pro CNIC Foundation, og er et Severo Ochoa Center of Excellence (SEV-2015-0505). Vi støttes også af den europæiske fond for regional udvikling (FEDER) “UNA Manera de hacer Europa”. UC anerkender støtten fra Associazione Italiana Ricerca Sul Cancro, foreningen for international Cancer Research (nu kendt som Worldwide Cancer Research) og det italienske sundhedsministerium. A.T. anerkender “Fondazione Banca del Monte di Lombardia” for delvist at støtte hans arbejde med PV Fellowship “Progetto Professionkend Ivano Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
References
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemistry. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

