Dinamica di oligomerizzazione dei recettori delle superfici cellulari nelle cellule viventi mediante microscopia a fluorescenza totale di riflessione interna combinata con l'analisi del numero e della luminosità
Summary
Descriviamo un approccio di imaging per la determinazione dello stato oligomerico oligomerico medio degli oligomeri mEGFP-tagged-receptor indotti dal legame del ligando nella membrana plasmatica delle cellule viventi. Il protocollo si basa sulla microscopia TIRF (Total Internal Reflection Fluorescence) combinata con l’analisi di Number and Brightness (N&B).
Abstract
Nonostante l’importanza e l’ubiquità dell’oligomerizzazione del recettore, pochi metodi sono applicabili per rilevare gli eventi di clustering e misurare il grado di clustering. Qui, descriviamo un approccio imaging per determinare lo stato oligomerico medio degli omocomplessi del recettore mEGFP nella membrana delle cellule viventi. Il protocollo si basa sulla microscopia TIRF (Total Internal Reflection Fluorescence) combinata con l’analisi di Number and Brightness (N&B). N&B è un metodo simile alla spettroscopia fluorescenza-correlazione (FCS) e all’istogramma di conteggio dei fotoni (PCH), che si basano sull’analisi statistica delle fluttuazioni dell’intensità della fluorescenza dei fluorofori che si diffondono dentro e fuori da un’illuminazione volume durante un periodo di osservazione. In particolare, N&B è una semplificazione del PCH per ottenere informazioni sul numero medio di proteine nelle miscele oligomeriche. Le ampiezze di fluttuazione dell’intensità sono descritte dalla luminosità molecolare del fluoroforo e dal numero medio di fluorofori all’interno del volume di illuminazione. Pertanto, N&B considera solo il primo e il secondo momento della distribuzione dell’ampiezza, vale a dire l’intensità media e la varianza. Questo è, allo stesso tempo, la forza e la debolezza del metodo. Poiché vengono considerati solo due momenti, N&B non è in grado di determinare la frazione molare degli oligomeri sconosciuti in una miscela, ma stima solo lo stato medio di oligomerizzazione della miscela. Tuttavia, può essere applicato a serie temporali relativamente piccole (rispetto ad altri metodi di momento) di immagini di cellule vive su base pixel per pixel, semplicemente monitorando le fluttuazioni temporali dell’intensità della fluorescenza. Riduce il tempo effettivo per pixel a pochi microsecondi, consentendo l’acquisizione nell’intervallo di tempo di secondi a millisecondi, che è necessario per l’oligomerizzazione rapida cinetica. Infine, è possibile esplorare grandi aree cellulari e compartimenti subcellulari.
Introduction
Descriviamo un approccio di imaging TIRF-N&B (Total Internal Reflection Fluorescence-Number and Brightness) per determinare lo stato oligomerico medio delle molecole recettoriali alla membrana plasmatica delle cellule vive, con l’obiettivo di collegare l’assemblaggio del recettore funzione biologica delle proteine (Figura 1).
Al momento del legame del ligando extracellulare, i recettori avviano la trasduzione del segnale intracellulare a seconda della loro conformazione, oligomerizzazione, potenziali co-recettori e composizione della membrana. Nonostante l’importanza e l’ubiquità dell’oligomerizzazione del recettore, riconosciuto come un evento chiave nella segnalazione cellulare1,2,3,4,5,6, 7, pochi metodi possono rilevare gli eventi di clustering e misurare il grado di clustering sperimentalmente8,9. Il volume confocale (x,y : 300 nm, z ) non è sufficientemente risolto per dimostrare l’interazione molecolare e la stoichiometria, anche dopo l’ottimizzazione da parte degli algoritmi di ripristino delle immagini10. La composizione di sottounità di oligomeri proteici non può essere risolta su base puramente spaziale anche con metodi di super-risoluzione a risoluzione x,y di 20-70 nm come PALM11, STORM12e STED13. Inoltre, la loro risoluzione temporale (nell’ordine dei minuti per immagine) non può seguire la cinetica nell’intervallo di secondi. Lo sbiancamento a passo di singola molecola risolve la stoichiometria degli oligomeri proteici solo se sono immobili14.
Uno dei metodi più versatili per misurare la densità e l’oligomerizzazione delle proteine fluorescenti all’interno di singole immagini è l’analisi della distribuzione dell’intensità spaziale (SpIDA), che si basa sul campionamento spaziale. È applicabile sia alle cellule chimicamente fisse che vive e consente l’analisi di diverse regioni di interesse della cellula contemporaneamente utilizzando la microscopia a fluorescenza standard15. In alternativa, i metodi del momento, come la spettroscopia fluorescenza-correlazione (FCS)16, l’istogramma di conteggio fotone (PCH)17e il numero e la luminosità (N&B)18,19, sono adatti per oligomeri quantitativi Misure. Questi metodi analizzano le fluttuazioni di intensità della fluorescenza che possono essere osservate nel tempo in cui i fluorofori si diffondono dentro e fuori da un volume di illuminazione. Le ampiezze delle fluttuazioni di intensità possono essere descritte in modo univoco dalla luminosità molecolare del fluoroforo e dal numero medio di fluorofori (n) all’interno del volume di illuminazione17 (Figura 2). Tipicamente, il coefficiente di diffusione dei fluorofori e il numero medio di molecole (inversamente correlate al valore G(0) all’interno del volume di illuminazione possono essere ottenuti da FCS20. Tuttavia, poiché il tempo di diffusione si adatta solo alla radice cubica della massa, la FCS non è sufficientemente sensibile per rilevare i cambiamenti nella massa molecolare21. In pratica, fcS monocolore non è in grado di rilevare la dimerizzazione dei recettori della membrana. PCH risolve con precisione le miscele di diversi oligomeri. Utilizzando più di due momenti della distribuzione dell’ampiezza, rileva molecole di luminosità diversa che occupano lo stesso volume di illuminazione. Scansione FCS22 e sviluppi, come l’interessante coppia-correlazione di luminosità molecolare (pCOMB) approccio23, introdotto per estendere la gamma di applicabilità dei metodi di correlazione della fluorescenza nei sistemi biologici24 , rimangono metodi a punto singolo privi della capacità di misurazioni veloci in un’ampia area di una cella, richiedendo molte osservazioni consecutive ad ogni pixel e l’acquisizione di dati nell’ordine di secondi.
N&B è una versione semplificata di PCH che considera solo il primo e il secondo momento dell’ampiezza della distribuzione della fluorescenza, vale a dire l’intensità media, , e la varianza,2 (Figura 2)18,19 e, per questo motivo, non può determinare la frazione molare di oligomeri sconosciuti in una miscela, ma stima solo lo stato medio di oligomerizzazione della miscela. Tuttavia, N&B ha il vantaggio di lavorare con serie temporali relativamente più piccole di immagini di cellule vive rispetto al PCH su base pixel per pixel, semplicemente monitorando le fluttuazioni al momento dell’intensità di fluorescenza. Poiché N&B riduce il tempo per pixel a pochi microsecondi, può seguire cinetiche di oligomerizzazione veloce su grandi aree cellulari, consentendo l’acquisizione di immagini su una scala temporale di secondi nella microscopia a scansione raster (ad esempio, confocale, 2 fotone) e millisecondi microscopia basata su fotocamera (ad es. TIRFM).
Diversi rapporti hanno dimostrato la capacità di N&B di quantificare il numero di sottounità nei cluster proteici mediante l’imaging di regioni cellulari estese. Gli ammassi di Paxillin-EGFP sono stati rilevati nei siti di adesione nelle cellule CHO-K125e l’aggregazione intracellulare del peptide patogeno Httex1p è stata descritta nelle cellule COS-726. N&B è stato richiesto per seguire l’oligomerizzazione guidata dal ligando del recettore ErbB27e l’effetto del ligand FGF21 su Klothob (KLB) e FGFR1c nelle celle HeLa28. La combinazione di imaging TIRF e analisi N&B è stata utilizzata per dimostrare che la dinamina-2 è principalmente tetramerica in tutta la membrana cellulare29. Abbiamo applicato N&B sia alla scansione raster che alle immagini TIRF per dimostrare la dimerizzazione guidata dal ligando dei recettori della membrana cellulare uPAR e FGFR130,31.
I metodi di correlazione della fluorescenza, come N&B, FCS e PCH, si basano sull’idea che in un volume aperto il numero di occupazione delle particelle segua una distribuzione di Poisson. Poiché possono essere rilevati solo i fotoni emettiti dai fluorofori, il valore medio per un’intensità di fluorescenza misurata rispetto al tempo in un pixel dell’immagine,  è il prodotto del numero medio di fluorofori nel volume di illuminazione, n, e la loro luminosità molecolare,17:
è il prodotto del numero medio di fluorofori nel volume di illuminazione, n, e la loro luminosità molecolare,17:

dove il numero di fotoni emessi per unità di tempo (convenzionalmente al secondo) per molecola quando la molecola è al centro del volume di illuminazione.
La luminosità è una proprietà di ogni fluoroforo in una data acquisizione impostata, mentre l’intensità è la somma di tutti i contributi di tutti i fluorofori. Nei concorsi biologici, la luminosità aumenterà con l’aumento del numero di fluorofori che fluttuano insieme, fornendo informazioni sullo stato di oligomerizzazione della proteina fluorescente. Le ampiezze di fluttuazione in un determinato pixel vengono misuratedalla varianza del segnale di fluorescenza,

Dove la media del quadrato  di intensità, , e il
di intensità, , e il  quadrato della media di intensità, , vengono calcolati dai singoli valori di intensità in ogni pixel di ogni fotogramma:
quadrato della media di intensità, , vengono calcolati dai singoli valori di intensità in ogni pixel di ogni fotogramma:
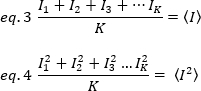
dove K è il numero di fotogrammi totali nella serie temporale. Sperimentalmente, è necessario calcolare per l’intera serie di immagini la varianza che descrive la dispersione dei singoli valori di intensità in ogni pixel di una singola immagine intorno al valore di intensità media. La varianza include tutte le fluttuazioni di origini diverse. In una prima approssimazione, la varianza dovuta alla diffazione delle particelle di fusione nel volume di illuminazione,20, può essere separata dalla varianza a causa del rumore del rivelatore,2d. Le due varianze sono indipendenti; pertanto, la varianza totale è data dalla loro somma:

La varianza, dovuta alle fluttuazioni molecolari all’esterno e all’esterno del volume di rilevamento, dipende linearmente dalla luminosità e dall’intensità molecolari:

Riorganizzazione di eq. 6 in base a eq. 1:

Secondo il concetto tipico nella spettroscopia di correlazione a fluorescenza, l’equazione 7 afferma che la varianza dovuta al numero di fluttuazioni dipende dal quadrato della luminosità della particella.
Quindi, la varianza dovuta alle fluttuazioni del rivelatore è una funzione lineare dell’intensità rilevata, partendo dal presupposto che il rivelatore sia azionato al di sotto del suo limite di saturazione19:

Nel caso dei rilevatori di conteggio dei fotoni unvalore 1 e c.0, quindi la varianza del rivelatore è uguale all’intensità media:

Per applicare questi concetti alle misurazioni reali nelle cellule vive, Gratton e colleghi18 definiscono la luminosità apparente, B, per ogni pixel come rapporto della varianza rispetto all’intensità media:

B è il parametro misurato sperimentalmente. In questo lavoro, le immagini di serie temporali dei recettori FGFR1 nella membrana plasmatica delle cellule HeLa vengono catturate dalla microscopia TIRF e la luminosità media apparente, B, è determinata dall’analisi N&B. Quindi, dopo l’aggiunta di FGF2, serie temporali consecutive vengono catturate per seguire i cambiamenti nell’auto-assemblaggio delle molecole recettoriali nella superficie della membrana dopo la stimolazione del recettore con il ligando canonico.
Tuttavia, poiché il rilevatore del microscopio TIRF è una telecamera EMCCD, l’espressione per la luminosità apparente deve essere modificata come19:

dove offset è l’offset di intensità dell’elettronica di rilevamento che è una caratteristica delle impostazioni del rivelatore. La varianza e l’intensità media per un rilevatore analogico sono date rispettivamente da:
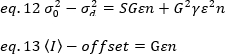
dove G è il guadagno analogico nei livelli digitali (DL/fotoni), S, i livelli digitali per fotone19, è dato dalla pendenza di un’intensità rispetto al grafico di varianza per una fonte di luce con intensità costante (senza fluttuazioni temporali). Il fattore z è correlato alla forma del volume di rilevamento dei pixel. Secondo Hassler et al.32, il fattore z è pari a 0,3 per l’imaging TIRF che lavora al massimo guadagno della telecamera di rilevamento19. I parametri offset, S e G sono caratteristiche della fotocamera e del microscopio. La luminosità apparente, B, si ottiene riorganizzando eq. 11 secondo eq. 12 e 13:

Dal punto di torto dal punto di toesa dal punto di tordo, è una funzione complessa dell’intensità del laser e dell’efficienza di rilevamento del sistema. Tuttavia, dal momento che B/S dipende linearmente da , è importante solo determinare il valore relativo di , per una determinata modalità di rilevamento:

dove è proporzionale a ” s” . Tuttavia, una calibrazione viene eseguita utilizzando un riferimento interno.
Protocol
Representative Results
Discussion
N&B richiede diverse precauzioni nella scelta del modello di cella e della strategia di etichettatura. Può essere applicato solo alle cellule vive che rimangono aderenti stabilmente durante il tempo di acquisizione dell’immagine. Le fluttuazioni extra dovute allo spostamento rigido dell’intera cellula potrebbero essere gestite con approcci appropriati per il ripristino dell’immagine38. Tuttavia, generalmente quando una cellula si muove, anche la membrana cellulare si deforma e la deformazione del…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Il CNIC è supportato dal Ministero della Ciencia, Innovacion y Universidades e dalla Pro CNIC Foundation, ed è un Severo Ochoa Center of Excellence (SEV-2015-0505). Siamo inoltre sostenuti dal Fondo europeo di sviluppo regionale (FEDER) “Una manera de hacer Europa”. UC riconosce il sostegno dell’Associazione Italiana Ricerca sul Cancro, dell’Associazione per la Ricerca Internazionale sul Cancro (ora nota come Worldwide Cancer Research) e del Ministero della Salute italiano. A.T. riconosce la “Banca banca del Monte di Lombardia” in parte sostenendo il suo lavoro con la Pv Fellowship “Progettoità Professionalità Ivano Becchi” 2011-2012.
Materials
| 3-Colour Fast TIRF Leica AM TIRF MC inverted microscope, with smi-automatic TIRF alignment. The microscope is equipped with a diode 488 nm laser, a 100×1.46 oil TIRF objective, Ex/Em Bandpass filters at 490/20 and 525/50, temperature/CO2 incubator and Andor DU 8285 VP EMCCD camera. The microscope is operated by Leica LIF software. | Leica Microsystems, Wetzlar, Germany | ||
| Albumin from Bovine Serum 98% minimun | Sigma-Aldrich, St. Louis, MI, USA | A7906-100G | |
| DMEM without Phenol Red with 25 mM HEPES | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 21063029 | Used serum free for microscopy |
| DMEM high-glucose GlutaMAX I | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10566-016 | Used for complete medium |
| Dulbecco's Phosphate Buffered Saline 10x (PBS) | Biowest, Nuaillé, France | X0515-500 | |
| Emission splitting system Photometrics DV2 | TeledynePhotometrics, Tucson, AZ, USA | ||
| Fetal Bovine Serum, qualified, Brazil | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 10270106 | 10% inactivated supplement for complete medium |
| Glass bottom 35-mm sterile 1.5 dishes | MatTek, Ashland, MA, USA | P35G-0.170-14-C | uncoated, glass thickness 0.17 microns |
| GraphPad Prism | GraphPad Software Inc., San Diego, CA, USA | ||
| Human cervical carcinoma (HeLa), serum-free animal component (AC) cells | Millipore-Sigma ECACC, Darmstadt, Germany | CB_08011102 | |
| iXonEM+ 897 EMCCD (back-illuminated) ANDOR camera controlled by ANDOR Solis software | Oxford Instruments, Andor TM Technology, Abingdon-on-Thames, UK | This camera, installed in an additional port of the microscope, is used for acquiring the N&B time series | |
| Matlab Executable N&B routine | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | download at https://www.cnic.es/en/investigacion/2/1187/tecnologia | |
| MatLab v.2018b | The MathWorks, Inc. Natick, MA, USA | download at https://www.mathworks.com/products/matlab.html | |
| Penicillin:Streptomycin for tissue culture 100x | Biowhittaker Inc. Walkersville, MD, USA | LONZA 17-602E | supplement for medium at Penicillin/Streptomycin 100U/100µg. |
| pN1-mEGFP-FGFR1 expression vector | Unit of Gynecological Oncology Research, European Institute of Oncology IRCCS, Milan, Italy | Zamai et al., 2019 | |
| pN1-N-Gly-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| pN1-N-Gly-mEGFP-mEGFP-GPI expression vector | Unit of Microscopy and Dynamic Imaging, CNIC, Madrid, Spain | Hellriegel et al., 2011 | |
| Recombinant FGF2 | PeproTech EC, Ltd., London, UK | Ligand solution: 20ng/mL of FGF2 in PBS supplemented with 0.01%BSA. | |
| Sodium pyruvate GIBCO | ThermoFisher Scientific | 11360070 | 1mM supplement for medium |
| TransIt-LT1 Transfection Reagent | MirusBio LLC, Madison, WI, USA | MIR 2300 | |
| Trypsin-EDTA (0.25%), phenol red | GIBCO Thermo Fisher Scientific,Waltham, MA, USA | 25200056 | |
| Type F Immersion liquid 10 mL | Leica Microsystems, Wetzlar, Germany | 11513 859 | |
| UltraPure BSA (50 mg/mL) | ThermoFisher Scientific | AM2618 | 0.1% supplement for medium without phenol red used for transfections |
References
- Agwuegbo, U. C., Jonas, K. C. Molecular and functional insights into gonadotropin hormone receptor dimerization and oligomerization. Minerva Ginecologica. 70 (5), 539-548 (2018).
- Ferre, S., et al. G protein-coupled receptor oligomerization revisited: functional and pharmacological perspectives. Pharmacological Reviews. 66 (2), 413-434 (2014).
- Marsango, S., Ward, R. J., Alvarez-Curto, E., Milligan, G. Muscarinic receptor oligomerization. Neuropharmacology. 136 (Pt C), 401-410 (2018).
- Oishi, A., Cecon, E., Jockers, R. Melatonin Receptor Signaling: Impact of Receptor Oligomerization on Receptor Function. International Review of Cell and Molecular Biology. 338, 59-77 (2018).
- Thelen, M., Munoz, L. M., Rodriguez-Frade, J. M., Mellado, M. Chemokine receptor oligomerization: functional considerations. Current Opinion in Pharmacology. 10 (1), 38-43 (2010).
- Van Craenenbroeck, K. GPCR oligomerization: contribution to receptor biogenesis. Subcellular Biochemistry. 63, 43-65 (2012).
- Wnorowski, A., Jozwiak, K. Homo- and hetero-oligomerization of beta2-adrenergic receptor in receptor trafficking, signaling pathways and receptor pharmacology. Cell Signaling Technology. 26 (10), 2259-2265 (2014).
- Fricke, F., Dietz, M. S., Heilemann, M. Single-molecule methods to study membrane receptor oligomerization. Chemphyschem. 16 (4), 713-721 (2015).
- Vidi, P. A., Ejendal, K. F., Przybyla, J. A., Watts, V. J. Fluorescent protein complementation assays: new tools to study G protein-coupled receptor oligomerization and GPCR-mediated signaling. Molecular and Cellular Endocrinology. 331 (2), 185-193 (2011).
- Trussell, H. J., et al., Trussell, J., et al. . Academic Press Library in Signal Processing. 4, 3-9 (2014).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313 (5793), 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nature Methods. 3 (10), 793-795 (2006).
- Nagerl, U. V., Willig, K. I., Hein, B., Hell, S. W., Bonhoeffer, T. Live-cell imaging of dendritic spines by STED microscopy. Proceedings of the National Academy of Sciences of the United States of America. 105 (48), 18982-18987 (2008).
- Tsekouras, K., Custer, T. C., Jashnsaz, H., Walter, N. G., Presse, S. A novel method to accurately locate and count large numbers of steps by photobleaching. Molecular Biology of the Cell. 27 (22), 3601-3615 (2016).
- Godin, A. G., et al. Revealing protein oligomerization and densities in situ using spatial intensity distribution analysis. Proceedings of the National Academy of Sciences of the United States of America. 108 (17), 7010-7015 (2011).
- Qian, H., Elson, E. L. Distribution of molecular aggregation by analysis of fluctuation moments. Proceedings of the National Academy of Sciences of the United States of America. 87 (14), 5479-5483 (1990).
- Chen, Y., Muller, J. D., So, P. T., Gratton, E. The photon counting histogram in fluorescence fluctuation spectroscopy. Biophysical Journal. 77 (1), 553-567 (1999).
- Dalal, R. B., Digman, M. A., Horwitz, A. F., Vetri, V., Gratton, E. Determination of particle number and brightness using a laser scanning confocal microscope operating in the analog mode. Microscopy Research and Technique. 71 (1), 69-81 (2008).
- Unruh, J. R., Gratton, E. Analysis of molecular concentration and brightness from fluorescence fluctuation data with an electron multiplied CCD camera. Biophysical Journal. 95 (11), 5385-5398 (2008).
- Hess, S. T., Huang, S., Heikal, A. A., Webb, W. W. Biological and chemical applications of fluorescence correlation spectroscopy: a review. Biochemistry. 41 (3), 697-705 (2002).
- Muller, J. D., Chen, Y., Gratton, E. Fluorescence correlation spectroscopy. Methods in Enzymology. 361, 69-92 (2003).
- Levi, V., Ruan, Q., Kis-Petikova, K., Gratton, E. Scanning FCS, a novel method for three-dimensional particle tracking. Biochemical Society Transactions. 31 (Pt 5), 997-1000 (2003).
- Hinde, E., et al. Quantifying the dynamics of the oligomeric transcription factor STAT3 by pair correlation of molecular brightness. Nature Communications. 7, 11047 (2016).
- Waithe, D., et al. Optimized processing and analysis of conventional confocal microscopy generated scanning FCS data. Methods. 140-141, 62-73 (2018).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Ossato, G., et al. A two-step path to inclusion formation of huntingtin peptides revealed by number and brightness analysis. Biophysical Journal. 98 (12), 3078-3085 (2010).
- Nagy, P., Claus, J., Jovin, T. M., Arndt-Jovin, D. J. Distribution of resting and ligand-bound ErbB1 and ErbB2 receptor tyrosine kinases in living cells using number and brightness analysis. Proceedings of the National Academy of Sciences of the United States of America. 107 (38), 16524-16529 (2010).
- Ming, A. Y., et al. Dynamics and Distribution of Klothobeta (KLB) and fibroblast growth factor receptor-1 (FGFR1) in living cells reveal the fibroblast growth factor-21 (FGF21)-induced receptor complex. Journal of Biological Chemistry. 287 (24), 19997-20006 (2012).
- Ross, J. A., et al. Oligomerization state of dynamin 2 in cell membranes using TIRF and number and brightness analysis. Biophysical Journal. 100 (3), L15-L17 (2011).
- Hellriegel, C., Caiolfa, V. R., Corti, V., Sidenius, N., Zamai, M. Number and brightness image analysis reveals ATF-induced dimerization kinetics of uPAR in the cell membrane. FASEB J. 25 (9), 2883-2897 (2011).
- Zamai, M., et al. Number and brightness analysis reveals that NCAM and FGF2 elicit different assembly and dynamics of FGFR1 in live cells. Journal of Cell Science. 132 (1), (2019).
- Hassler, K., et al. Total internal reflection fluorescence correlation spectroscopy (TIR-FCS) with low background and high count-rate per molecule. Optics Express. 13 (19), 7415-7423 (2005).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments. (92), e51994 (2014).
- Beenken, A., Mohammadi, M. The FGF family: biology, pathophysiology and therapy. Nature Reviews Drug Discovery. 8 (3), 235-253 (2009).
- Joubert, J., Sharma, D. Light microscopy digital imaging. Current Protocols in Cytometry. , (2011).
- Gell, C., Berndt, M., Enderlein, J., Diez, S. TIRF microscopy evanescent field calibration using tilted fluorescent microtubules. Journal of Microscopy. 234 (1), 38-46 (2009).
- Burghardt, T. P. Measuring incidence angle for through-the-objective total internal reflection fluorescence microscopy. Journal of Biomedical Optics. 17 (12), 126007 (2012).
- Trullo, A., Corti, V., Arza, E., Caiolfa, V. R., Zamai, M. Application limits and data correction in number of molecules and brightness analysis. Microscopy Research and Technique. 76 (11), 1135-1146 (2013).
- Caiolfa, V. R., et al. Monomer-dimer dynamics and distribution of GPI-anchored uPAR are determined by cell surface protein assemblies. Journal of Cell Biology. 179 (5), 1067-1082 (2007).
- Campbell, R. E., et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America. 99 (12), 7877-7882 (2002).
- Cutrale, F., et al. Using enhanced number and brightness to measure protein oligomerization dynamics in live cells. Nature Protocols. 14 (2), 616-638 (2019).
- Dunsing, V., Chiantia, S. A Fluorescence Fluctuation Spectroscopy Assay of Protein-Protein Interactions at Cell-Cell Contacts. Journal of Visualized Experiments. (142), (2018).

