ミトコンドリア膜動態を再構成するためのモデル膜プラットフォーム
Summary
ミトコンドリア融合は、ミトコンドリアダイナミクスの基礎となる重要なホメオスタティック反応である。ここで説明する インビトロ 再構成システムは、膜テザリング、ドッキング、ヘミフュージョン、および細孔開孔を解決できるミトコンドリア内膜融合を研究するものである。細胞膜システムを探索する際のこのアプローチの多様性について考察する。
Abstract
ミトコンドリアダイナミクスは、オルガネラの多様な機能と細胞応答に不可欠です。混雑した空間的に複雑なミトコンドリア膜は、調節要因を区別する困難な環境です。タンパク質および脂質成分の実験的制御は、調節の特定の質問に答える助けになります。しかし、これらの因子の定量的操作は、細胞アッセイでは困難です。ミトコンドリア内膜融合の分子機構を調べるには、ミトコンドリア内膜の脂質環境を模した インビトロ 再構成プラットフォームを導入しました。ここでは、脂質二重層を調製し、ミトコンドリア膜タンパク質を再構成するための詳細なステップについて説明する。このプラットフォームは、ミトコンドリア内膜融合中の中間体と、個々の遷移のための運動学を定量的に分析することを可能にした。このプロトコルは、非対称脂質組成を用いた二重層の製造について説明し、膜貫通タンパク質をクッション二層に再構成するための一般的な考慮事項を説明する。この方法は、他の膜系を研究するために適用され得る。
Introduction
膜区画化は真核細胞1の特徴である(図1A)。生体膜は、二次元溶媒以上のものとして認識され、タンパク質機能および高分子複合体集合体22,33の調節において重要な役割を果たしている環境と考えられている。天然脂質は、膜タンパク質活性33,44を調節するリガンドである。膜空間組織と多様な形状に彫刻される膜の能力は、新しい機能33,5を選択するための重要な物理的特性である。
モデル膜プラットフォームは、細胞膜構造、ダイナミクス、および機能66、7、87,8を理解するのに役立つバイオミメティックシステムです。モデル膜は、典型的には、定義された生物物理学的性質(剛性、厚さ、および弾力性)を有する、明確に定義された組成の脂質混合物を含む。蛍光イメージングに結合されて、モデル膜のプラットホームは膜の構造および機能99、10、1110の量的分析を11可能にする。脂質二重層再構成戦略は、SNARE媒介膜融合99、10、DNA10媒介膜融合12、およびウイルス融合11、13,13を研究するために用いられている。このような方法の利点は、観察可能な反応イベント14に先行する中間ステップの運動情報を得る可能性がある。
原形質膜はモデル膜を用いて広く研究されている。脂質相分離を有する二重層は、細胞シグナル伝達11、15、16,15,16において重要な脂質いかだ構造を研究するために開発された。細胞受容体の組織を調べるのにマイクロパターン化された脂質平板二重層17、18が用いられている。17,ポリマーまたはゲル支持膜は、膜細胞骨格組織、細胞シグナル伝達中の膜タンパク質分配、および細胞-細胞接触19での移動を研究するためのバイオ模倣系として使用されてきた。
人工膜システムは、細胞内小器官20の研究にも応用されている。オルガネラは、独特のサブ環境を作り出す特徴的な形態を特徴とする。小胞子(ER)ネットワークは一例である。リポソームへのレチコンの再構成の際、細胞ERと同様の特性を有する管状膜構造が形成される21.また、アプラスチンの添加により、ER融合タンパク質が、リポソームから脂質細管を誘導してネットワーク20を形成することができる。これは、プロテオリポソームがオルガネラ形態とダイナミクスに機能的洞察を提供する方法の一例です。
ミトコンドリアの膜融合と核分裂は、ミトコンドリア集団22、23、24、25,24,25の健康に不可欠である。22,ダイナミンファミリーGTPasesのセットはミトコンドリア膜融合を触媒する。Mfn 1/2は外膜融合を触媒する。Opa1は、内膜融合26を媒介する(図1B)。Opa1には、ミトコンドリア内膜に膜貫通固定された長い形態(l-Opa1)と、膜間空間に存在する「可溶性」短い形態(s-Opa1)の2つの形態がある。2つのOpa1形態の比率は、Oma1とYme1L,27、28、29、30の2つのプロテアーゼの活性によって調節される。27,2829,30Opa1規制における重要な質問は、Opa1の2つの形態、(短くおよび長い)媒介膜融合およびそれらの規制相互作用2828、29、31、32、33の方法が含まれる。,29,31,32,33
ここでは、内膜融合におけるl-およびs-Opa1の役割を明らかにするミトコンドリア内膜融合の調査に成功した再構成戦略について述べる。ポリマーテザリング脂質二重層と200nmのユニラメラ小胞を用いてミトコンドリア内膜を模倣したプラットフォームを開発した。脂質二重層の下のポリマーテザーの利点としては、以下のものが挙げられる。まず、再構成された膜貫通タンパク質を保存し、それ以外の場合はガラススライド34に近いことによって破壊され得る。第二に、脂質二重層とガラス基板との間に厚い水層を果たし、毛穴開孔9の研究を容易にし、PEGポリマーの粘弾性特性により膜湾曲変化を可能にする35。膜融合の工程を特徴付けるために3色蛍光イメージングを使用しました(図1C-F)。
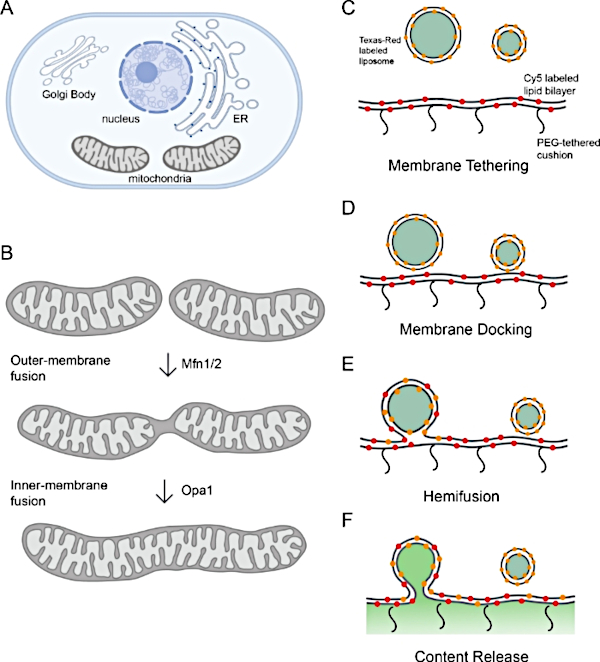
図1:ミトコンドリア膜融合のモニタリング
(A)オルガネラは細胞膜コンパートメントである。(B) ミトコンドリア膜融合の逐次的なステップ。ミトコンドリアの外膜の融合はMfn1および/またはMfn2によって触媒され、内膜融合はOpa1によって媒介される。(C-F)ミトコンドリア膜融合を研究するインビトロ再構成プラットフォームの模式図。プラットフォームには、プロテオリポソームとポリマーテザリング脂質二重層の2つの部分があり、どちらも再構成されたl-Opa1を備えています。2種類の蛍光膜色素と含量マーカーを含む蛍光標識は、膜融合中のステップを区別するのに役立ちます。2つの膜マーカー(Cy5-PE(赤)とテキサスレッドPE(オレンジ)は、閉じた膜ドッキングについて報告することができるFRETペアを作ります。プロテオリポソームを標識するテキサスレッド-PEの拡散は、脂質脱混合(ヘミフュージョン)の指標である。コンテンツの放出は、カルセイン信号のデケンチ(緑色で示す)を通して監視されます。バイオレンダーを使用して作成されたパネル A と B。この図の大きなバージョンを表示するには、ここをクリックしてください。
Protocol
Representative Results
Discussion
インビトロモデル膜システムは、明確に定義された条件下で複雑な膜プロセスを記述することができます。これらのシステムは、分子機構6、15、20、3815,20を6明らかにするために、複雑な分子プロセスに必要な最小限の成分を38区別することができる。膜タンパク質の場合、リポソームおよび平面支持二重層は一般的な再構成システムである。固形脂質二重層とは対照的に、ポリマー・テザリング二重層における基質と支持膜の間のポリマークッションは、大きな膜タンパク質の自由な移動性を可能にし、そして膜貫通タンパク質は自由に拡散し、組み立てる34。これらの特徴は、ミトコンドリア内膜融合36の運動学を調査するのに役立ちました。
ラングミュア・ブロジェット/ラングミュア・シェーファー(LB/LS)技術を用いて、ポリマーテザー化脂質二重層を用意しました。これにより、不斉脂質成分を持つ二重層を調製することができます。細胞膜は非対称リーフレット組成を有し、LB/LSアプローチは、そのような二重層の研究を可能にする。シェーファー移動により、ガラス基板全体を脂質二重層で覆うことができます。二重層調製のためにきれいな表面を準備することが重要です。さらに、シェーファー転送を正しく実行するには練習が必要です。正常なシェーファー転移は、脂質二重層に望ましくない欠陥を生じさせる可能性がある。このプロトコルにおいて、フィルムバランスに加えた圧力は、20%カーディオリピンを含む二重層に適用可能である。他のコンポーネントとの二重層については、主要コンポーネントの表面圧力面積の一元性を参照してください。別の方法は、ラングミュア・ブロジェット/ベシクル融合(LB/VF)法であり、下の脂質単層がラングミュアトラフの空気水界面からきれいな基板に移され、次いでリポソームが支持された脂質層の上部に融合し、最終二重層39を形成する。LB/VF法を用いた膜タンパク質の再構成は、プロテオリポソームの融合によって再構成が行われるため、LB/LSより簡単です。しかし、ベシクル融合には過剰なリポソームの添加が必要であり、濃度依存性タンパク質相互作用に依存する膜事象の研究が複雑になる可能性がある。
好ましい機能的指向におけるポリマーテザリング脂質二重層およびリポソームの両方への膜貫通タンパク質の再構成が成功することは重要であり、実施することは困難である。これを説明するには実験的な制御が必要です。ポリマーテザリング脂質二重層の場合、再構成中に脂質二重層の完全性を維持することも重要である。界面活性剤濃度は、脂質二重層の溶解を防ぐために比較的低く保たれなければならないが、目的のタンパク質37,40,40の変性を防ぐほど高い。ここで説明する方法は、単一分子研究のための膜タンパク質の再構成に理想的であるが、大規模な研究のために必ずしもスケーラブルではない。界面活性剤の選択も重要な考慮事項です。しばしば、精製および貯蔵に使用される界面活性剤は良い出発点である。界面活性剤の最大濃度は、通常、CMC36の約200倍少なく、界面活性剤がタンパク質の安定性を維持し、タンパク質凝集を防止する範囲で、膜36の完全性を維持する。2または3の界面活性剤を含むカクテルが考えられてもよい。リポソームへの再構成のためには、界面活性剤の低濃度は必要ありません。しかし、CMC未満の界面活性剤濃度は、リポソームに対して均一なサイズおよび形態分布を維持することが好ましい。染料の漏出を防ぐためには、染料含有緩衝液に対して透析する必要がある。
リポソーム系融合アッセイとは対照的に、我々が確立したプラットフォームは、膜融合の各ステップの運動学を調査するためのアプローチを提供する。この方法は、ほぼネイティブ条件下で膜貫通融合タンパク質を研究する能力を提供します。モデル膜プラットフォームは、ミトコンドリア内膜のような細胞内環境における膜タンパク質集合体とオリゴマー化、膜「スカルプティング」、タンパク質と脂質の相互作用を研究するために適用できます。この方法はまた、二重層組成非対称性のような膜蛋白質相互作用における重要な生理学的条件の探索を可能にする。主要なミトコンドリア脂質の役割は、カーディオリピン、リポソームおよびポリマー支持二重層の両方の二重層特性において定義され続ける。イオン強度、膜厚さ、膜の剛性、膜の曲率、膜弾性粘性特性などの特性は、タンパク質が特定の機能状態に組み立てる能力に影響を与える可能性があります。モデル膜系を創造的に応用する今後の研究は、膜タンパク質組織と機能の新たな側面を明らかにする可能性を秘めています。
Disclosures
The authors have nothing to disclose.
Acknowledgements
著者らは、チャールズ・H・フッド財団児童保健研究賞の支援と、マサチューセッツ総合病院分子生物学部門からの寛大な支援を認めている。
Materials
| 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-(Cyanine 5) | Avanti polar lipid | Cat #: 810335C1mg | membrane fluorescent markers |
| 1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-[methoxy(polyethylene glycol)-2000] (ammonium salt) | Avanti Polar lipids | Cat #: 880130P | lipid molecules |
| 1',3'-bis[1,2-dioleoyl-sn-glycero-3-phospho]-glycerol (sodium salt) | Avanti Polar lipids | Cat #: 710335P | lipid molecules |
| 18:1 (Δ9-Cis) PC (DOPC) | Avanti Polar lipids | Cat #: 850375P | lipid molecules |
| 1-palmitoyl-2-oleoyl-sn-glycero-3-phosphoethanolamine | Avanti Polar lipids | Cat #: 850757P | lipid molecules |
| Alexa Fluor 488 Antibody Labeling Kit | ThermoFisher Scientific | A20181 | |
| Amber vial with PTFE liner | Fisher scientific | 14-955-332 | sample vials to keep lipid solutions |
| Calcein | Sigma-Aldrich | Cat #: C0875; PubChem Substance ID: 24892279 | fluorescent dye |
| Chloroform | Fisher scientific | 298-500/ C295-4 | Fisher brand Chloroform is usually quite reliable for lipid works. |
| Concavity slide (1 well) | Electron Microscopy Science | 71878-05 | applied as Schaefer Slide |
| FCS analysis tool | Smith Lab, University of Akron | software tool | |
| Fiji /ImageJ | Fiji | SCR_002285 | software tool |
| Fisherbrand Cover Glasses: Circles | Fisher scientific | 12-545-102 | Cover glass for solid supported lipid bilayers, the item is now discontinued as authors prepared the manuscript. An alternative is Fisher brand premium cover glass with catalog number: 12-548-5M |
| GTP Disodium salt | SIGMA-ALDRICH INC | Cat #: 10106399001 | |
| Langmuir & Langmuir-Blodgett Trough | Biolin Scientifc | KN2002 | |
| L-α-lysophosphatidylinositol (Liver, Bovine) (sodium salt) | Avanti Polar lipids | Cat #: 850091P | lipid molecules |
| Mini Extruder | Avanti Polar lipids | 610020 | |
| n-Dodecyl-β-D-Maltopyranoside | Anatrace | Cat #: D310 25 GM | surfactant for reconstitution |
| n-Octyl-α-D-Glucopyranoside | Anatrace | Cat #: O311HA 25 GM | surfactant for reconstitution |
| PC Membranes 0.2μm | Avanti Polar Lipids | 610006 | |
| Rabbit Anti-Opa1 antibody | NOVUS BIOLOGICALS | Cat #: NBP2-59770 | antibody for Opa1 C-terminal detection |
| Slidebook | Intelligent imaging | RRID: SCR_014300 | software tool |
| Teflon threaded seal tape | Fisher Scientific | NC0636085 | taflon tape for sample storage |
| Texas Red 1,2-Dihexadecanoyl-sn-Glycero-3-Phosphoethanolamine, Triethylammonium Salt (Texas Red DHPE) | ThermoFisher Scientific | Cat #: T1395MP | membrane fluorescent markers |
References
- Sackmann, E., Lipowsky, R., Sackmann, E. Biological membranes architecture and function. Structure and Dynamics of Membranes. 1, 1-63 (1995).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118, 1099-1102 (2005).
- Schink, K. O., Tan, K. W., Stenmark, H. Phosphoinositides in Control of Membrane Dynamics. Annual Review of Cell and Developmental Biology. 32, 143-171 (2016).
- Gu, R. X., Ingolfsson, H. I., de Vries, A. H., Marrink, S. J., Tieleman, D. P. Ganglioside-Lipid and Ganglioside-Protein Interactions Revealed by Coarse-Grained and Atomistic Molecular Dynamics Simulations. Journal of Physical Chemistry B. 121 (15), 3262-3275 (2017).
- Schafer, D. A. Coupling actin dynamics and membrane dynamics during endocytosis. Current Opinion in Cell Biology. 14 (1), 76-81 (2002).
- Chan, Y. H., Boxer, S. G. Model membrane systems and their applications. Current Opinion in Chemical Biology. 11 (6), 581-587 (2007).
- Biswas, K. H., Groves, J. T. Hybrid Live Cell-Supported Membrane Interfaces for Signaling Studies. Annual Reviews in Biophysics. 48, 537-562 (2019).
- Pick, H., Alves, A. C., Vogel, H. Single-Vesicle Assays Using Liposomes and Cell-Derived Vesicles: From Modeling Complex Membrane Processes to Synthetic Biology and Biomedical Applications. Chemical Reviews. 118 (18), 8598-8654 (2018).
- Kiessling, V., Domanska, M. K., Tamm, L. K. Single SNARE-mediated vesicle fusion observed in vitro by polarized TIRFM. Biophysical Journal. 99 (12), 4047-4055 (2010).
- Kiessling, V., et al. Rapid fusion of synaptic vesicles with reconstituted target SNARE membranes. Biophysical Journal. 104 (9), 1950-1958 (2013).
- Yang, S. T., Kiessling, V., Tamm, L. K. Line tension at lipid phase boundaries as driving force for HIV fusion peptide-mediated fusion. Nature Communication. 7, 11401 (2016).
- Rawle, R. J., van Lengerich, B., Chung, M., Bendix, P. M., Boxer, S. G. Vesicle fusion observed by content transfer across a tethered lipid bilayer. Biophysical Journal. 101 (8), 37 (2011).
- Chao, L. H., Klein, D. E., Schmidt, A. G., Pena, J. M., Harrison, S. C. Sequential conformational rearrangements in flavivirus membrane fusion. Elife. 3, 04389 (2014).
- Floyd, D. L., Harrison, S. C., van Oijen, A. M. Analysis of kinetic intermediates in single-particle dwell-time distributions. Biophysical Journal. 99 (2), 360-366 (2010).
- Sezgin, E., Schwille, P. Model membrane platforms to study protein-membrane interactions. Molecular Membrane Biology. 29 (5), 144-154 (2012).
- Ge, Y., Gao, J., Jordan, R., Naumann, C. A. Changes in Cholesterol Level Alter Integrin Sequestration in Raft-Mimicking Lipid Mixtures. Biophysical Journal. 114 (1), 158-167 (2018).
- Dutta, D., Kam, L. C. Micropatterned, multicomponent supported lipid bilayers for cellular systems. Methods in Cell Biology. 120, 53-67 (2014).
- Groves, J., Ferrari, M., Desai, T., Bhatia, S. Supported Lipid Bilayers as Mimics for Cell Surfaces and as Tools in Biotechnology. BioMEMS and Biomedical Nanotechnology. , (2006).
- Shoaib, T., Nalam, P. C., He, Y., Chen, Y., Espinosa-Marzal, R. M. Assembly, Morphology, Diffusivity, and Indentation of Hydrogel-Supported Lipid Bilayers. Langmuir. 33 (28), 7105-7117 (2017).
- Wang, N., Rapoport, T. A. Reconstituting the reticular ER network – mechanistic implications and open questions. Journal of Cell Science. 132 (4), 227611 (2019).
- Powers, R. E., Wang, S., Liu, T. Y., Rapoport, T. A. Reconstitution of the tubular endoplasmic reticulum network with purified components. Nature. 543 (7644), 257-260 (2017).
- Chan, D. C. Fusion and fission: interlinked processes critical for mitochondrial health. Annual Reviews of Genetics. 46, 265-287 (2012).
- Nunnari, J., Suomalainen, A. Mitochondria: in sickness and in health. Cell. 148 (6), 1145-1159 (2012).
- Wai, T., Langer, T. Mitochondrial Dynamics and Metabolic Regulation. Trends in Endocrinology Metabolism. 27 (2), 105-117 (2016).
- Westermann, B. Mitochondrial fusion and fission in cell life and death. Nature Reviews Molecular Cell Biology. 11 (12), 872-884 (2010).
- Chen, H., et al. Mitofusins Mfn1 and Mfn2 coordinately regulate mitochondrial fusion and are essential for embryonic development. Journal of Cell Biology. 160 (2), 189-200 (2003).
- Ishihara, N., Fujita, Y., Oka, T., Mihara, K. Regulation of mitochondrial morphology through proteolytic cleavage of OPA1. EMBO Journal. 25 (13), 2966-2977 (2006).
- Anand, R., et al. The i-AAA protease YME1L and OMA1 cleave OPA1 to balance mitochondrial fusion and fission. Journal of Cell Biology. 204 (6), 919-929 (2014).
- Mishra, P., Carelli, V., Manfredi, G., Chan, D. C. Proteolytic cleavage of Opa1 stimulates mitochondrial inner membrane fusion and couples fusion to oxidative phosphorylation. Cell Metabolism. 19 (4), 630-641 (2014).
- Baker, M. J., et al. Stress-induced OMA1 activation and autocatalytic turnover regulate OPA1-dependent mitochondrial dynamics. EMBO Journal. 33 (6), 578-593 (2014).
- DeVay, R. M., et al. Coassembly of Mgm1 isoforms requires cardiolipin and mediates mitochondrial inner membrane fusion. Journal of Cell Biology. 186 (6), 793-803 (2009).
- Rainbolt, T. K., Lebeau, J., Puchades, C., Wiseman, R. L. Reciprocal Degradation of YME1L and OMA1 Adapts Mitochondrial Proteolytic Activity during Stress. Cell Reports. 14 (9), 2041-2049 (2016).
- Ban, T., et al. Molecular basis of selective mitochondrial fusion by heterotypic action between OPA1 and cardiolipin. Nature Cell Biology. 19 (7), 856-863 (2017).
- Tanaka, M., Sackmann, E. Polymer-supported membranes as models of the cell surface. Nature. 437 (7059), 656-663 (2005).
- Shilts, K., Naumann, C. A. Tunable cell-surface mimetics as engineered cell substrates. Biochimica Biophysica Acta Biomembrane. 1860 (10), 2076-2093 (2018).
- Ge, Y., et al. Two forms of Opa1 cooperate to complete fusion of the mitochondrial inner-membrane. Elife. 9, 50973 (2020).
- Ge, Y., Siegel, A. P., Jordan, R., Naumann, C. A. Ligand binding alters dimerization and sequestering of urokinase receptors in raft-mimicking lipid mixtures. Biophysical Journal. 107 (9), 2101-2111 (2014).
- Simons, K., Vaz, W. L. Model systems, lipid rafts, and cell membranes. Annual Review of Biophysics and Biomolecular Structure. 33, 269-295 (2004).
- Kiessling, V., Liang, B., Tamm, L. K. Reconstituting SNARE-mediated membrane fusion at the single liposome level. Methods in Cell Biology. 128, 339-363 (2015).
- Siegel, A. P., Kimble-Hill, A., Garg, S., Jordan, R., Naumann, C. A. Native ligands change integrin sequestering but not oligomerization in raft-mimicking lipid mixtures. Biophysics Journal. 101 (7), 1642-1650 (2011).

