Imagerie à cellules vivantes de réseaux unicellulaires (LISCA) - une technique polyvalente pour quantifier la cinétique cellulaire
Summary
Nous présentons une méthode pour l’acquisition des cours de temps de rapporteur de fluorescence à partir de cellules simples utilisant des réseaux micropatterned. Le protocole décrit la préparation de réseaux unicellulaires, la mise en place et le fonctionnement de la microscopie time-lapse à balayage de cellules vivantes et un outil d’analyse d’image open source pour la présélection automatisée, le contrôle visuel et le suivi des cours de temps de fluorescence intégrés aux cellules par site d’adhésion.
Abstract
L’imagerie par cellules vivantes de réseaux unicellulaires (LISCA) est une méthode polyvalente pour collecter des cours temporels de signaux de fluorescence à partir de cellules individuelles à haut débit. En général, l’acquisition de cours de temps unicellulaires à partir de cellules cultivées est entravée par la motilité cellulaire et la diversité des formes cellulaires. Les microréseaux adhésifs standardisent les conditions unicellulaires et facilitent l’analyse d’images. LISCA combine des puces à ADN unicellulaires avec une microscopie time-lapse à balayage et un traitement d’image automatisé. Ici, nous décrivons les étapes expérimentales de la prise de cours de temps de fluorescence unicellulaire dans un format LISCA. Nous transfectons les cellules adhérentes à un réseau micropatterned utilisant le codage d’ARNm pour la protéine fluorescente verte améliorée (eGFP) et surveillons la cinétique d’expression eGFP de centaines de cellules en parallèle via la microscopie time-lapse de balayage. Les piles de données d’image sont automatiquement traitées par un logiciel nouvellement développé qui intègre l’intensité de fluorescence sur les contours cellulaires sélectionnés pour générer des cours de temps de fluorescence unicellulaires. Nous démontrons que les cours de temps d’expression d’eGFP après transfection d’ADN messagère sont bien décrits par un modèle de traduction cinétique simple qui révèle des taux d’expression et de dégradation de l’ARNm. D’autres applications de LISCA pour des corrélations de temps d’événement des marqueurs multiples dans le cadre de l’apoptosis de signalisation sont discutées.
Introduction
Ces dernières années, l’importance des expériences unicellulaires est devenue évidente. Les données des cellules individuelles permettent d’enquêter sur la variabilité de cellule à cellule, la résolution des corrélations de paramètres intracellulaires et la détection de la cinétique cellulaire qui reste cachée dans les mesures d’ensemble1,2,3. Afin d’étudier la cinétique cellulaire de milliers de cellules individuelles en parallèle, de nouvelles approches sont nécessaires qui permettent de surveiller les cellules dans des conditions normalisées sur une période de temps de plusieurs heures à plusieurs jours suivie d’une analyse quantitative des données 4. Ici, nous présentons l’imagerie à cellules vivantes de réseaux unicellulaires (LISCA), qui combine l’utilisation de réseaux microstructurés avec la microscopie time-lapse et l’analyse automatisée d’images.
Plusieurs méthodes de génération de réseaux unicellulaires microstructurés ont été établies et publiées dans la littérature5,6. Ici, nous décrivons brièvement le modelage des protéines initiées par plasma à micro-échelle (μPIPP). Un protocole détaillé de la fabrication de réseaux unicellulaires à l’aide de μPIPP se trouve également dans la référence7. L’utilisation de réseaux unicellulaires permet l’alignement de milliers de cellules sur des points d’adhésion standardisés présentant des microenvironnements définis pour chaque cellule et réduit ainsi une source de variabilité expérimentale (Figure 1A). Les réseaux unicellulaires sont utilisés pour surveiller les cours de temps des marqueurs fluorescents destinés à indiquer une variété de processus cellulaires. La microscopie à long terme en mode time-lapse à balayage permet de surveiller une grande partie des réseaux unicellulaires et donc d’échantillonner des données unicellulaires à haut débit sur un temps d’observation de plusieurs heures, voire plusieurs jours. Cela génère des piles de lignes de temps d’images à partir de chaque position du tableau(Figure 1B). Afin de réduire la grande quantité de données d’image et d’extraire les cours de temps de fluorescence unicellulaire souhaités de manière efficace, un traitement d’image automatisé est nécessaire qui tire parti du positionnement des cellules(Figure 1C).
Le défi de LISCA est d’adapter les protocoles expérimentaux et les outils de calcul pour former un test à haut débit qui génère des données quantitatives et reproductibles de la cinétique cellulaire. Dans cet article, nous fournissons une description étape par étape des méthodes individuelles et de la façon dont elles sont combinées dans un test LISCA. À titre d’exemple, nous discutons le cours de temps de l’expression verte augmentée de la protéine fluorescente (eGFP) après la livraison artificielle d’ADN messagère. L’expression de l’eGFP après l’administration de l’ARNm est décrite par des équations de vitesse de réaction modélisant la traduction et la dégradation de l’ARNm. L’ajustement de la fonction du modèle pour l’évolution dans le temps de la concentration eGFP à la lecture LISCA de l’intensité de fluorescence pour chaque cellule individuelle au fil du temps donne les meilleures estimations des paramètres du modèle tels que le taux de dégradation de l’ARNm. Comme résultat représentatif nous discutons l’efficacité de livraison d’ADN messagère de deux agents lipide-basés différents de transfection et comment leurs distributions de paramètre diffèrent.
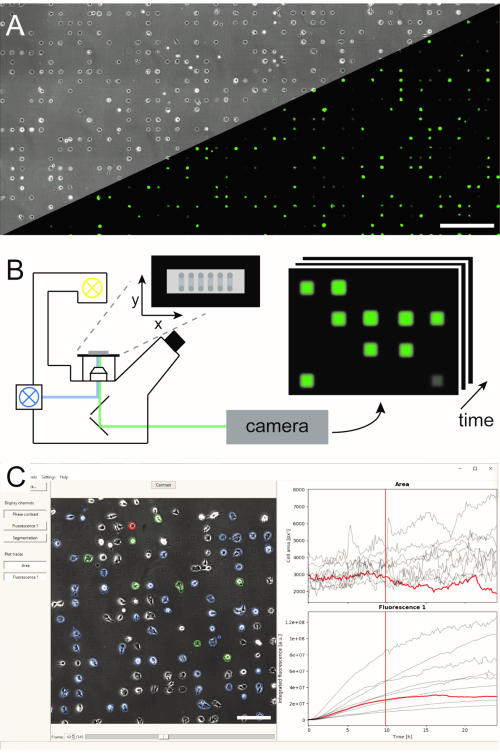
Figure 1: Représentation du flux de travail LISCA combinant (A) des réseaux unicellulaires micro-modelés (B) une microscopie time-lapse à balayage et (C) une analyse d’image automatisée de séries d’images enregistrées. Les réseaux unicellulaires sont constitués d’un motif bidimensionnel de carrés adhésifs cellulaires avec un interespace répulsif pour les cellules conduisant à un arrangement des cellules sur le micropattern, comme on peut le voir dans l’image à contraste de phase ainsi que l’image de fluorescence des cellules exprimant l’eGFP (A). L’ensemble de la zone microstructurée est entité en mode time-lapse de numérisation à plusieurs reprises en prenant des images à une séquence de positions(B). Les séries d’images enregistrées sont traitées pour lire l’intensité de fluorescence par cellule au fil du temps (C). Barres d’échelle: 500 μm(A),200 μm (C). Veuillez cliquer ici pour voir une version plus grande de cette figure.
Protocol
Representative Results
Discussion
Ici nous avons décrit LISCA comme technique polyvalente pour suivre la cinétique cellulaire des étiquettes fluorescentes intracellulaires au niveau unicellulaire. Afin d’effectuer une expérience LISCA réussie, chacune des étapes décrites de la section de protocole doit être établie individuellement, puis toutes les étapes doivent être combinées. Chacun des trois aspects majeurs de LISCA comporte des étapes cruciales.
Fabrication de puces à ADN unicellulaires
La qual…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Ce travail a été soutenu par des subventions de la Fondation allemande pour la science (DFG) au Centre de recherche collaborative (SFB) 1032. Le soutien du ministère fédéral allemand de l’Éducation, de la Recherche et de la Technologie (BMBF) dans le cadre du projet coopératif 05K2018-2017-06716 Medisoft ainsi qu’une subvention de la Bayerische Forschungsstiftung sont vivement appréciés. Anita Reiser a été soutenue par une bourse DFG de la Graduate School of Quantitative Biosciences Munich (QBM).
Materials
| Adtech Polymer Engineering PTFE Microtubing | Fisher Scientific | 10178071 | |
| baking oven | Binder | 9010-0190 | |
| CFI Plan Fluor DL 10x | Nikon | MRH20100 | |
| Desiccator | Roth | NX07.1 | |
| Eclipse Ti-E | Nikon | ||
| eGFP mRNA | Trilink | L-7601 | |
| Female Luer to Tube Connector | MEDNET | FTL210-6005 | |
| Fetal bovine serum | Thermo Fisher | 10270106 | |
| Fibronectin | Yo Proteins | 663 | |
| Filter set eGFP | AHF | F46-002 | |
| Fisherbrand Translucent Platinum-Cured Silicone Tubing | Fisher Scientific | 11768088 | |
| HEPES (1 M) | Thermo Fisher | 15630080 | |
| Incubation Box | Okolab | OKO-H201 | |
| incubator | Binder | 9040-0012 | |
| L-15 without phenol red | Thermo Fisher | 21083027 | |
| Lipofectamine 2000 | Thermo Fisher | 11668027 | |
| Male Luer | in-house fabricated consisting of teflon | ||
| Male Luer to Tube Connector | MEDNET | MTLS210-6005 | alternative to in-house fabricated male luers |
| NaCl (5 M) | Thermo Fisher | AM9760G | |
| Needleless Valve to Male Luer Connector | MEDNET | NVFMLLPC | |
| NIS Elements | Nikon | Imaging software Version 5.02.00 | |
| NOA81 | Thorlabs | NOA81 | Fast Curing Optical Adhesive for tube system assembly |
| Opti-MEM | Thermo Fisher | 31985062 | |
| PCO edge 4.2 M-USB-HQ-PCO | pco | ||
| Phosphate buffered saline (PBS) | in-house prepared | ||
| Plasma Cleaner | Diener Femto | Pico-BRS | |
| PLL(20 kDa)-g[3.5]-PEG(2 kDa) | SuSoS AG | ||
| silicon wafer mit mircorstructures | in-house fabricated | ||
| Sola Light Engine | Lumencor | ||
| sticky slide VI 0.4 | ibidi | 80608 | |
| Sylgard 184 Silicone Elastomer Kit | Dow Corning | 1673921 | |
| Tango 2 | Märzhäuser | 00-24-626-0000 | |
| Ultrapure water | in-house prepared | ||
| uncoated coverslips | ibidi | 10813 | |
| Injekt-F Solo, 1 mL | Omilab | 9166017V | with replacement sporn |
References
- Altschuler, S. J., Wu, L. F. Cellular heterogeneity: do differences make a difference. Cell. 141 (4), 559-563 (2010).
- Locke, J. C., Elowitz, M. B. Using movies to analyse gene circuit dynamics in single cells. Nature Reviews Microbiology. 7 (5), 383-392 (2009).
- Spencer, S. L., Gaudet, S., Albeck, J. G., Burke, J. M., Sorger, P. K. Non-genetic origins of cell-to-cell variability in TRAIL-induced apoptosis. Nature. 459 (7245), 428-432 (2009).
- El-Ali, J., Sorger, P. K., Jensen, K. F. Cells on chips. Nature. 442 (7101), 403-411 (2006).
- Segerer, F. J., et al. Versatile method to generate multiple types of micropatterns. Biointerphases. 11 (1), 011005 (2016).
- Piel, M., Théry, M. . Micropatterning in cell biology, part A/B/C. , (2014).
- Reiser, A., Zorn, M. L., Murschhauser, A., Rädler, J. O., Ertl, P., Rothbauer, M. . Cell-Based Microarrays: Methods and Protocols. , 41-54 (2018).
- Picone, R., Baum, B., McKendry, R. . Methods in cell biology. 119, 73-90 (2014).
- Reiser, A. . Single-cell time courses of mRNA transport and translation kinetics. , (2019).
- . Softmatter LMU-Raedler Group Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- . MIAAB, 29262 Available from: https://github.com/SoftmatterLMU-RaedlerGroup/pyama (2020)
- Reiser, A., et al. Correlation of mRNA delivery timing and protein expression in lipid-based transfection. Integrative Biology. 11 (9), 362-371 (2019).
- Reiser, A., Woschée, D., Mehrotra, N., Krzysztoń, R. S., Strey, H. H., Rädler, J. O. Supplementing data and code for: “Correlation of mRNA delivery timing and protein expression in lipid-based transfection”. Zenodo. , (2019).
- Ferizi, M., et al. Stability analysis of chemically modified mRNA using micropattern-based single-cell arrays. Lab on a Chip. 15 (17), 3561-3571 (2015).
- Fröhlich, F., et al. Multi-experiment nonlinear mixed effect modeling of single-cell translation kinetics after transfection. NPJ systems biology and applications. 4 (1), 1-12 (2018).
- Krzysztoń, R., et al. Single-cell kinetics of siRNA-mediated mRNA degradation. Nanomedicine: Nanotechnology, Biology and Medicine. 21, 102077 (2019).
- Röttgermann, P. J., Alberola, A. P., Rädler, J. O. Cellular self-organization on micro-structured surfaces. Soft Matter. 10 (14), 2397-2404 (2014).
- Röttgermann, P. J., Dawson, K. A., Rädler, J. O. Time-resolved study of nanoparticle induced apoptosis using microfabricated single cell arrays. Microarrays. 5 (2), 8 (2016).
- Murschhauser, A., et al. A high-throughput microscopy method for single-cell analysis of event-time correlations in nanoparticle-induced cell death. Communications Biology. 2 (1), 1-11 (2019).
- Chatzopoulou, E. I., et al. Chip-based platform for dynamic analysis of NK cell cytolysis mediated by a triplebody. Analyst. 141 (7), 2284-2295 (2016).
- Sztilkovics, M., et al. Single-cell adhesion force kinetics of cell populations from combined label-free optical biosensor and robotic fluidic force microscopy. Scientific Reports. 10 (1), 1-13 (2020).

