Picometer-Precision Rastreamento de posição atômica através de microscopia eletrônica
Summary
Este trabalho apresenta um fluxo de trabalho para rastreamento de posição atômica em imagens de microscopia eletrônica de transmissão de resolução atômica. Este fluxo de trabalho é realizado usando um aplicativo Matlab de código aberto (EASY-STEM).
Abstract
Os modernos microscópios eletrônicos de transmissão de transmissão de varredura corrigidos pela aberração (AC-STEM) conseguiram visualizar com sucesso a visualização direta de colunas atômicas com resolução sub-angstrom. Com esse progresso significativo, a quantificação e análise avançada de imagem ainda estão nos estágios iniciais. Neste trabalho, apresentamos o caminho completo para a metrologia das imagens de microscopia eletrônica de transmissão de resolução atômica (STEM). Isso inclui (1) dicas para aquisição de imagens STEM de alta qualidade; (2) denoização e correção à deriva para melhorar a precisão da medição; (3) obtenção de posições atômicas iniciais; (4) indexar os átomos com base em vetores de células unitárias; (5) quantificar as posições da coluna atômica com o encaixe de pico único 2D-Gaussiano ou (6) rotinas de montagem multi-pico para colunas atômicas ligeiramente sobrepostas; (7) quantificação da distorção/tensão da rede dentro das estruturas cristalinas ou nos defeitos/interfaces em que a periodicidade da rede é interrompida; e (8) alguns métodos comuns para visualizar e apresentar a análise.
Além disso, será apresentado um simples aplicativo MATLAB gratuito autodesenvolvido (EASY-STEM) com uma interface gráfica de usuário (GUI). A GUI pode auxiliar na análise de imagens STEM sem a necessidade de escrever código de análise ou software dedicado. Os métodos avançados de análise de dados aqui apresentados podem ser aplicados para a quantificação local de relaxamentos de defeitos, distorções estruturais locais, transformações de fases locais e não-centrosimetria em uma ampla gama de materiais.
Introduction
O desenvolvimento da correção de aberração esférica no microscópio eletrônico de transmissão de varredura moderna (STEM) permitiu que microscopistas sondassem cristais com feixes de elétrons do tamanho de sub-angstrom1,2. Isso permitiu a imagem de colunas atômicas individuais em uma grande variedade de cristais com imagens de resolução atômica interpretáveis para elementos pesados e leves3,4. Desenvolvimentos recentes em detectores de elétrons diretos pixelados e algoritmos de análise de dados permitiram técnicas de imagem de reconstrução de fase, como a ptychografia, com melhorias adicionais na resolução espacial por volta das 30 pm5,6,7. Além disso, o recente progresso na tomografia STEM permitiu até mesmo a reconstrução da resolução atômica tridimensional da única nanopartícula8. O microscópio eletrônico tornou-se, assim, uma ferramenta extraordinariamente poderosa para quantificar propriedades estruturais em materiais com alta precisão e especificidade do local.
Com as imagens STEM de ultra-alta resolução como entrada de dados, foram realizadas medições diretas de distorções estruturais para extrair informações físicas de cristais na escala atômica9,10. Por exemplo, o acoplamento de defeito entre um dopant Mo na monocamada WS2 e uma única vaga S foi diretamente visualizado medindo as posições atômicas e, em seguida, calculando os comprimentos de ligação projetados11. Além disso, a medição em interfaces de cristal, como os limites de grãos coalescidos na monocamada WS2,pode exibir o arranjo atômico local12. A análise interfacial realizada nas paredes de domínio ferroelétrico em LiNbO3 revelou que a parede de domínio era uma combinação dos estados Ising e Neel13. Outro exemplo é a visualização das estruturas de vórtice polar alcançadas nas superlattices SrTiO3-PbTiO3, obtidas através do cálculo dos deslocamentos da coluna atômica de titânio em relação às posições de estrôncio e coluna de chumbo14. Finalmente, os avanços nos algoritmos de visão computacional, como a denoização de imagens com análise de componentes de princípio não local15, a desconvolução de Richardson e Lucy16,a correção de deriva com o registro não linear17, e o reconhecimento de padrões com aprendizagem profunda, reforçaram significativamente a precisão da medição para a precisão sub-picometro18. Um exemplo é o alinhamento e o registro de imagem de múltiplas imagens criogênicas-STEM para melhorar a relação sinal-ruído. Posteriormente, a técnica de mascaramento de Fourier foi aplicada para analisar as ondas de densidade de carga em cristais, visualizando diretamente a distorção periódica da rede19. Embora a incrível instrumentação STEM corrigida pela aberração seja cada vez mais acessível a pesquisadores em todo o mundo, os procedimentos e métodos avançados de análise de dados permanecem incomuns e uma enorme barreira para um sem experiência em análise de dados.
No presente trabalho, mostramos o caminho completo para a metrologia das imagens STEM de resolução atômica. Este processo inclui, em primeiro lugar, a aquisição das imagens STEM com um microscópio corrigido pela aberração seguido pela realização de denoização/correção pós-aquisição para maior precisão de medição. Discutiremos ainda mais os métodos existentes para resolver e quantificar com precisão as posições da coluna atômica com o encaixe de pico único 2D-Gaussiano ou rotinas de montagem de vários picos para colunas atômicas ligeiramente sobrepostas20,21. Por fim, este tutorial discutirá métodos para quantificação da distorção/tensão da rede dentro das estruturas cristalinas ou nos defeitos/interfaces onde a periodicidade da rede é interrompida. Também introduziremos um simples aplicativo MATLAB gratuito auto-desenvolvido (EASY-STEM) com uma interface gráfica de usuário (GUI) que pode ajudar na análise de imagens STEM sem a necessidade de escrever código de análise ou software dedicado. Os métodos avançados de análise de dados aqui apresentados podem ser aplicados para a quantificação local de relaxamentos de defeitos, distorções estruturais locais, transformações de fases locais e não-centrosimetria em uma ampla gama de materiais.
Protocol
Representative Results
Discussion
Ao trabalhar no processamento pós-aquisição, alguns cuidados também precisam ser tomados. Para começar, durante a correção de deriva da imagem, o algoritmo assume que a imagem 0° tem a direção horizontal de digitalização rápida, então verifique novamente a direção antes do cálculo. Se a direção de digitalização não estiver corretamente definida, o algoritmo de correção de deriva falhará e poderá até introduzir artefatos na saída17. Em seguida, durante a denoização da imagem, certos métodos podem introduzir um artefato; por exemplo, a filtragem Fourier pode criar contraste de coluna atômica nos locais de vacância ou remover recursos finos nas imagens, se a resolução espacial não for limitada corretamente. Como resultado, é de vital importância verificar se as imagens denoizadas se assemelham muito às imagens de entrada bruta originais.
Em seguida, ao determinar as posições atômicas iniciais com base no máximo/mínimo local, tente ajustar a distância mínima de restrição entre os picos para evitar criar posições redundantes entre colunas atômicas. Estas posições redundantes são artefatos gerados devido ao algoritmo reconhecer erroneamente o maxima/minima local na imagem como colunas atômicas. Além disso, pode-se ajustar o valor limiar para encontrar a maioria das posições se houver grandes diferenças de contraste entre várias espécies atômicas na imagem (por exemplo, em imagens ADF-STEM de WS2). Depois de obter a maioria das posições atômicas iniciais na imagem, tente adicionar manualmente as faltas ou remover as extras com o melhor esforço. Além disso, o método de indexação dos átomos é o mais eficaz quando não há grandes interrupções nas periodicidades dentro da imagem. Quando há interrupções, como limites de grãos ou limites de fase apresentados na imagem, a indexação pode falhar. A solução para este problema é definir as áreas de interesse na imagem (clicando no botão Definir Área de Interesse no aplicativo EASY-STEM) e, em seguida, indexar e refinar as posições dentro de cada área separadamente. Depois, pode-se facilmente combinar conjuntos de dados de diferentes áreas na mesma imagem em um conjunto de dados e trabalhar na análise.
Finalmente, depois de aplicar os encaixes de pico 2D-Gaussian, espalhe os pontos de posições refinados na imagem de entrada para verificar os resultados de montagem para ver se as posições refinadas se desviam das colunas atômicas. A precisão fornecida pelo único algoritmo de montagem gaussiano é suficiente na maioria dos experimentos STEM; no entanto, se a posição se desviar devido à intensidade de um átomo vizinho, use o algoritmo de montagem multi-pico (mpfit) em vez de isolar a intensidade das colunas atômicas adjacentes21. Caso contrário, se a posição se desviar devido ao problema de qualidade da imagem ou à baixa intensidade das colunas atômicas específicas, é sugerido descartar a posição instalada naquele local.
Existem vários algoritmos existentes e especializados para a medição da posição atômica, por exemplo, o software de catador de octaedra de oxigênio22, atomap python pacote23e pacote StatSTEM Matlab24. No entanto, esses algoritmos têm algumas limitações em certos aspectos. Por exemplo, o catador de octahedra de oxigênio requer a entrada de imagens STEM para conter apenas colunas atômicas claramente resolvidas e, portanto, não conseguiu abordar o problema nas imagens com colunas atômicas sobrepostas intensidades21. Por outro lado, embora a Atomap possa calcular as posições das colunas atômicas “semelhantes a halteres”, o processo não é muito simples. Além disso, o StatSTEM é um ótimo algoritmo para quantificar as intensidades sobrepostas, mas seu processo de montagem baseado em modelo iterativo é computacionalmente caro21. Em contraste, nossa abordagem, introduzida neste trabalho juntamente com o aplicativo Matlab EASY-STEM, que é integrado com o algoritmo mpfit avançado, pode resolver o problema da intensidade sobreposta e é menos computacionalmente caro que o StatSTEM, ao mesmo tempo em que oferece precisão de medição competitiva. Além disso, a análise da Atomap e dos pacotes de software de captador de oxigênio octahedra são projetados e especializados para analisar os dados dos cristais perovskite ABO3, enquanto o sistema de indexação mostrado neste trabalho é muito mais flexível sobre diferentes sistemas de materiais. Com o método neste trabalho, os usuários podem projetar e personalizar totalmente a análise de dados para seus sistemas materiais exclusivos com base nos resultados de saída que contêm tanto posições atômicas refinadas quanto a indexação vetorial da célula unitária.
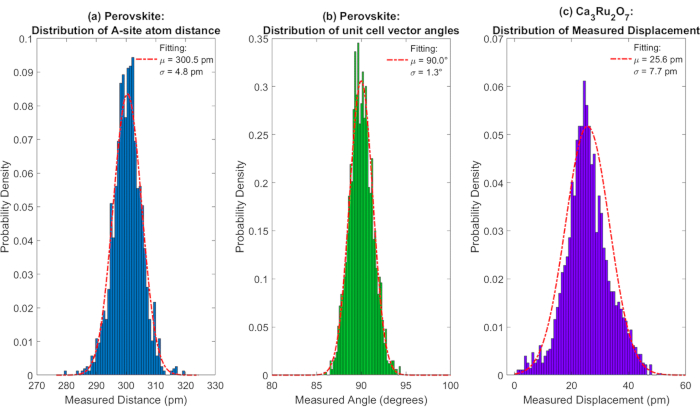
Figura 6: Quantificação estatística do achado da posição atômica. (a) A distribuição do perovskite A-local à distância do local A apresentada em um histograma. O encaixe de distribuição normal é plotado e sobreposto como a linha tracejada vermelha mostrando a média de 300,5 pm e o desvio padrão de 16:8 pm. (b) A quantificação estatística da medição do ângulo vetorial da unidade perovskite é apresentada como um histograma. O encaixe de distribuição normal é plotado e sobreposto como a linha vermelha tracejada mostrando a média de 90,0° e o desvio padrão de 1,3°. (c) A quantificação estatística da medição do deslocamento polar em Ca3Ru2O7 (CRO) é apresentada como histograma. O encaixe de distribuição normal é plotado e sobreposto como a linha vermelha tracejada mostrando a média das 25:6 pm e o desvio padrão das 19:7 pm. Clique aqui para ver uma versão maior desta figura.
O método aqui introduzido fornece precisão em nível de picometro e simplicidade para implementação. Para demonstrar a precisão da medição, a quantificação estatística do achado da posição atômica é apresentada na Figura 6. As medidas da distribuição de distânciaperovskite perovskite A-local e distribuição de ângulo vetorial de células unitárias são traçadas usando histograma na Figura 6a e Figura 6b, respectivamente. Ao encaixar a curva de distribuição normal às distribuições, a distribuição de distância do local A mostra uma média de 300,5 pm e desvio padrão de 4,8 pm e distribuição do ângulo vetorial da célula unitária mostra uma média de 90,0° e desvio padrão de 1,3°. A quantificação estatística indica que o método aqui proposto permite a precisão no nível do picometro e pode aliviar muito a distorção devido à deriva durante a imagem. Este resultado sugere que essa medida é confiável quando as informações físicas a serem medidas são maiores ou iguais a cerca de 22h. Por exemplo, no caso dos cristais cro acima mencionados, a medição da magnitude do deslocamento polar é apresentada na Figura 6c. A medição mostra uma média de 25,6 pm, um desvio padrão de 7,7 pm, e mostra que a medição de deslocamento polar em imagens DE STEM CRO é sólida. Além disso, é preciso ter mais cautela em caso de limitações experimentais, como baixa relação sinal-ruído quando amostras sensíveis ao feixe de imagem. Nesses casos, as posições atômicas medidas precisam ser examinadas de perto contra as imagens brutas para garantir a validade da medição. Consequentemente, o método de análise introduzido aqui tem limitações à precisão de medição quando comparado com algoritmos mais recentes e avançados. Nosso método é insuficiente quando a precisão é necessária no nível do sub-picometro, por isso uma rotina de análise mais avançada é necessária se o recurso a ser extraído na imagem estiver abaixo de um certo limiar. Por exemplo, o algoritmo de registro não rígido mostrou medição de precisão sub-picometer no silício e permite uma medição precisa da variação do comprimento do título em uma única nanopartícula Pt25. Mais recentemente, o algoritmo de aprendizagem profunda foi empregado para identificar vários tipos de defeitos de ponto em monocamadas de dichalcogenidas metálicas de transição 2D a partir de uma enorme quantidade de dados de imagem STEM. Posteriormente, a medição foi realizada na imagem média de diferentes tipos de defeitos e este método também demonstrou precisão de nível sub-picometro na distorção em torno desses defeitos18. Consequentemente, como um plano futuro para aumentar a capacidade de análise, estamos no progresso do desenvolvimento e implementação de algoritmos mais avançados, como o deep learning. Também tentaremos integrá-los às futuras atualizações da ferramenta de análise de dados.
Disclosures
The authors have nothing to disclose.
Acknowledgements
O trabalho de L.M. e N.A. apoiado pelo Penn State Center for Nanoscale Sciences, um MRSEC da NSF sob o número de subvenção DMR-2011839 (2020 – 2026). D.M. foi apoiado pelo Programa de Pesquisa e Desenvolvimento Direcionado ao Laboratório (LDRD) da ORNL, que é gerenciado pela UT-Battelle, LLC, para o Departamento de Energia dos EUA (DOE). A.C. e N.A. reconhecem o programa FA9550-18-0277 da Força Aérea, bem como o programa GAME MURI, 10059059-PENN para apoio.
Materials
| EASY-STEM | Nasim Alem Group, Pennsylvania State University | Matlab app for STEM image processing; Download link: https://github.com/miaoleixin1994/EASY-STEM.git | |
| JoVE article example script | Nasim Alem Group, Pennsylvania State University | Example Script for sorting atoms in unit cells | |
| Matlab Optimization Tool Box | MathWorks | Optimization add-on packge in Matlab | |
| Matlab | MathWorks | Numerical calculation software | |
| Matlab: Image Processing Tool Box | MathWorks | Image processing add-on packge in Matlab |
References
- Batson, P. E., Dellby, N., Krivanek, O. L. Sub-aångstrom resolution using aberration corrected electron optics. Nature. , (2002).
- Haider, M., et al. Electron microscopy image enhanced. Nature. , (1998).
- Muller, D. A., Nakagawa, N., Ohtomo, A., Grazul, J. L., Hwang, H. Y. Atomic-scale imaging of nanoengineered oxygen vacancy profiles in SrTiO3. Nature. , (2004).
- Findlay, S. D., et al. Robust atomic resolution imaging of light elements using scanning transmission electron microscopy. Applied Physics Letters. , (2009).
- Tate, M. W., et al. High Dynamic Range Pixel Array Detector for Scanning Transmission Electron Microscopy. Microscopy and Microanalysis. , (2016).
- Rodenburg, J. M., McCallum, B. C., Nellist, P. D. Experimental tests on double-resolution coherent imaging via STEM. Ultramicroscopy. 48, 304-314 (1993).
- Jiang, Y., et al. Electron ptychography of 2D materials to deep sub-ångström resolution. Nature. 559, 343-349 (2018).
- Yang, Y., et al. Deciphering chemical order/disorder and material properties at the single-atom level. Nature. , (2017).
- Bals, S., Van Aert, S., Van Tendeloo, G., Ávila-Brande, D. Statistical estimation of atomic positions from exit wave reconstruction with a precision in the picometer range. Physics Review Letters. , (2006).
- Kim, Y. M., He, J., Biegalski, M., et al. Probing oxygen vacancy concentration and homogeneity in solid-oxide fuel-cell cathode materials on the subunit-cell level. Nature Mater. 11, (2012).
- Azizi, A., et al. Defect Coupling and Sub-Angstrom Structural Distortions in W1-xMoxS2 Monolayers. Nano Letters. , (2017).
- Reifsnyder Hickey, D., et al. Illuminating Invisible Grain Boundaries in Coalesced Single-Orientation WS2 Monolayer Films. arXiv. , (2020).
- Mukherjee, D., et al. Atomic-scale measurement of polar entropy. Physics Review B. 100, 1-21 (2019).
- Yadav, A. K., et al. Observation of polar vortices in oxide superlattices. Nature. , (2016).
- Yankovich, A. B., et al. Non-rigid registration and non-local principle component analysis to improve electron microscopy spectrum images. Nanotechnology. , (2016).
- Ishizuka, K., Abe, E. Improvement of Spatial Resolution of STEM-HAADF Image by Maximum-Entropy and Richardson-Lucy Deconvolution. EMC. , (2004).
- Ophus, C., Ciston, J., Nelson, C. T. Correcting nonlinear drift distortion of scanning probe and scanning transmission electron microscopies from image pairs with orthogonal scan directions. Ultramicroscopy. , (2016).
- Lee, C. H., et al. Deep learning enabled strain mapping of single-atom defects in two-dimensional transition metal dichalcogenides with sub-picometer precision. Nano Letters. , (2020).
- Savitzky, B. H., et al. Bending and breaking of stripes in a charge ordered manganite. Nature Communications. 8, 1-6 (2017).
- Stone, G., et al. Atomic scale imaging of competing polar states in a Ruddlesden-Popper layered oxide. Natature Communications. 7, 1-9 (2016).
- Mukherjee, D., Miao, L., Stone, G., Alem, N. mpfit: a robust method for fitting atomic resolution images with multiple Gaussian peaks. Advanced Structural and Chemical Imaging. , (2020).
- Wang, Y., Salzberger, U., Sigle, W., Eren Suyolcu, Y., van Aken, P. A. Oxygen octahedra picker: A software tool to extract quantitative information from STEM images. Ultramicroscopy. 168, 46-52 (2016).
- Nord, M., Vullum, P. E., MacLaren, I., Tybell, T., Holmestad, R. Atomap: a new software tool for the automated analysis of atomic resolution images using two-dimensional Gaussian fitting. Advanced Structral and Chemical Imaging. 3, 9 (2017).
- De Backer, A., vanden Bos, K. H. W., Vanden Broek, W., Sijbers, J., Van Aert, S. StatSTEM: An efficient approach for accurate and precise model-based quantification of atomic resolution electron microscopy images. Ultramicroscopy. 171, 104-116 (2016).
- Yankovich, A. B., et al. Picometre-precision analysis of scanning transmission electron microscopy images of platinum nanocatalysts. Nature Communications. , (2014).

