ओपन-सोर्स smfBox का उपयोग कर सटीक और सटीक एकल-अणु FRET माप बनाना
Summary
यह आलेख व्यक्तिगत पर पूरी तरह से सही सटीक FRET माप बनाने के लिए चरण-दर-चरण निर्देश प्रदान करता है, स्वतंत्र रूप से खुले स्रोत, सस्ती smfBox का उपयोग करके बायोमोलेक्यूल्स को अलग करता है, स्विच ऑन से, संरेखण के माध्यम से और ध्यान केंद्रित करने के लिए, डेटा संग्रह और विश्लेषण के लिए।
Abstract
SmfBox एकल-अणु Förster अनुनाद ऊर्जा हस्तांतरण (smFRET) के लिए हाल ही में विकसित लागत प्रभावी, ओपन-सोर्स उपकरण है, जो स्वतंत्र रूप से बायोमोलेक्यूल्स को अधिक सुलभ बनाता है। इस अवलोकन में डुप्लेक्स डीएनए नमूनों में सटीक FRET दक्षताओं के माप करने के लिए इस उपकरण का उपयोग करने के लिए एक चरण-दर-चरण प्रोटोकॉल शामिल है, जिसमें नमूना तैयारी, उपकरण सेटअप और संरेखण, डेटा अधिग्रहण और पूर्ण विश्लेषण दिनचर्या का विवरण शामिल है। प्रस्तुत दृष्टिकोण, जिसमें सटीक FRET-व्युत्पन्न दूरी माप के लिए आवश्यक सभी सुधार कारकों को निर्धारित करना शामिल है, FRET समुदाय में हाल के सहयोगी कार्य के एक बड़े शरीर पर बनाता है, जिसका उद्देश्य मानक प्रोटोकॉल और विश्लेषण दृष्टिकोण स्थापित करना है। यह प्रोटोकॉल, जो आसानी से बायोमोलेक्यूलर सिस्टम की एक श्रृंखला के लिए अनुकूलहै, व्यापक वैज्ञानिक समुदाय के लिए smFRET को लोकतांत्रिक बनाने में बढ़ते प्रयासों को जोड़ता है।
Introduction
एकल-अणु Förster अनुनाद ऊर्जा हस्तांतरण (smFRET) एक ऐसी तकनीक है जो व्यक्तिगत अणुओं के स्तर पर दो रंगों-एक दाता और एक स्वीकर्ता के बीच FRET दक्षता को मापता है। FRET दो रंगों के अतिव्यापी ऊर्जा स्पेक्ट्रा से उत्पन्न होने वाली एक फोटोफिजिकल प्रक्रिया है: दाता एक विशिष्ट तरंग दैर्ध्य के प्रकाश से उत्साहित होता है और स्वीकर्ता को गैर-विकिरणीय रूप से ऊर्जा स्थानांतरित करता है, जिसके परिणामस्वरूप स्वीकर्ता से उत्सर्जन होता है। इस हस्तांतरण की दक्षता दो रंगों के बीच की दूरी की छठी शक्ति के व्युत्क्रमानुपाती है, इसलिए स्थानांतरण दक्षता दूरी 1 के साथ भिन्न होती है। इस प्रकार, इस FRET दक्षता का उपयोग अणु (ओं) 2 के बारे में स्थानिक जानकारी निर्धारित करने के लिए किया जा सकता है, जिसमें रंजक जुड़े हुए हैं, 3-10 एनएम की सीमा के भीतर। यह पैमाने पर, और तथ्य यह है कि FRET दक्षता में परिवर्तन Angstrom आणविक आंदोलनों 3 के प्रति संवेदनशील हैं, तकनीक को बायोमोलेक्यूल्स के बारे में संरचनात्मक जानकारी की जांच करने के लिए अच्छी तरह से अनुकूल बनाता है- जैसे न्यूक्लिक एसिड और प्रोटीन-पहनावा औसत की जटिलताओं के बिना 4,5,6। जबकि सापेक्ष FRET दक्षताओं में परिवर्तन का उपयोग बायोमोलेक्यूलर इंटरैक्शन और संरचनात्मक गतिशीलता की निगरानी के लिए किया जा सकता है, प्रोटीन (un) तह, प्रतिलेखन, और डीएनए प्रतिकृति और मरम्मत जैसी प्रमुख सेलुलर प्रक्रियाओं पर प्रकाश डाला जा सकता है, पूर्ण FRET दक्षता का उपयोग बायोमोलेक्यूलर संरचना निर्धारण के लिए सटीक दूरी निर्धारित करने के लिए किया गया है7,8,9,10,11 , क्रिस्टलीकरण या ठंड की आवश्यकता पर काबू पाने के रूप में कुछ अन्य संरचनात्मक तरीकों के लिए आवश्यक है4,12.
smFRET प्रयोगों सबसे अधिक दो रूपों, confocal या कुल आंतरिक प्रतिबिंब प्रतिदीप्ति (TIRF) माइक्रोस्कोपी ले लो. दोनों दृष्टिकोणों के बीच बायोमोलेक्यूल्स की आणविक गतिशीलता की जांच आमतौर पर पिको से मिलीसेकंड (कॉन्फोकल, स्वतंत्र रूप से अलग अणुओं) से मिलीसेकंड से लेकर घंटों तक (टीआईआरएफ, सतह स्थिर अणुओं) तक टाइमस्केल पर की जा सकती है। यह प्रत्येक तकनीक में शामिल विभिन्न सेटअपों के कारण है। TIRF माइक्रोस्कोपी में, अणुओं को स्लाइड की सतह पर स्थिर किया जाता है और एक तरंग (चित्रा 1 ए) द्वारा उत्तेजित किया जाता है। यहां, हालांकि, फोकस कॉन्फोकल माइक्रोस्कोपी पर है क्योंकि यह smfBox का प्रारूप है। Confocal माइक्रोस्कोपी में, अणुओं को स्थिर नहीं किया जाता है और इसके बजाय स्वतंत्र रूप से confocal मात्रा (~ 1 fL) के माध्यम से ब्राउनियन गति के माध्यम से फैलाया जाता है, जो समाधान के भीतर कुछ निर्दिष्ट गहराई पर एक स्थान पर एक उच्च संख्यात्मक एपर्चर लेंस के माध्यम से एक लेजर बीम पर ध्यान केंद्रित करके बनता है (चित्रा 1 बी)। परिणामी उत्सर्जन को एक ही एपर्चर के माध्यम से वापस केंद्रित किया जाता है और एक डाइक्रोइक दर्पण के माध्यम से फ़िल्टर किया जाता है (पूर्ण योजनाबद्ध के लिए चित्रा 1 सी )। यह तब किसी भी आउट-ऑफ-फोकस प्रकाश को हटाने के लिए और एक हिमस्खलन फोटोडायोड (एपीडी) पर पिनहोल के माध्यम से केंद्रित होता है। जब एपीडी एक फोटॉन का पता लगाता है, तो यह एक टीटीएल पल्स आउटपुट करता है, जिसका समय पिकोसेकंड रिज़ॉल्यूशन तक रिकॉर्ड किया जा सकता है। कॉन्फोकल वॉल्यूम के आसपास के क्षेत्र के भीतर इन स्वतंत्र रूप से अलग अणुओं का अवलोकन समय आमतौर पर मिलीसेकंड के क्रम के भीतर होता है।
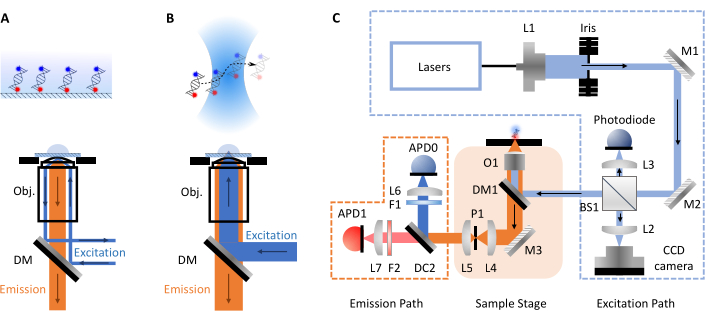
चित्र1: माइक्रोस्कोपी और smfBox सेटअप के सिद्धांतों को दिखाने वाली योजनाबद्ध योजनाएं। (A) कुल आंतरिक प्रतिबिंब प्रतिदीप्ति (TIRF) माइक्रोस्कोपी सिद्धांत: उत्तेजना प्रकाश को उद्देश्य (Obj.) के किनारे में निर्देशित किया जाता है और कवरस्लिप-बफर इंटरफ़ेस पर कुल आंतरिक प्रतिबिंब से गुजरता है जो सतह से जुड़े अणुओं को उत्तेजित करने के लिए एक तेजी से क्षयकारी evanescence क्षेत्र उत्पन्न करता है। (बी) कॉन्फोकल माइक्रोस्कोपी: स्वतंत्र रूप से विवर्तन करने वाले अणु नमूने में केंद्रित एक निकट विवर्तन-सीमित स्थान से उत्साहित होते हैं। (सी) इस प्रोटोकॉल में उपयोग किया जाने वाला smfBox सेटअप, सभी प्रमुख घटकों को दर्शाता है: हिमस्खलन फोटोडायोड (एपीडी), बीम स्प्लिटर (बीएस), डाइक्रोइक दर्पण (डीएम), फिल्टर (एफ), दर्पण (एम), उद्देश्य (ओ) और पिनहोल (पी)। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहाँ क्लिक करें.
हाल ही में, smFRET तकनीकों में दो रंग उत्तेजना शामिल हैं, जहां दाता और स्वीकर्ता उत्तेजना तरंग दैर्ध्य से मेल खाने वाले लेजर को वैकल्पिक किया जाता है5। यह दो तरीकों में से एक में किया जा सकता है, पहला KHz टाइमस्केल पर निरंतर तरंग लेजर को मॉड्युलेट करके, जिसे वैकल्पिक लेजर उत्तेजना (एलेक्स) 13,14 के रूप में जाना जाता है। दूसरी विधि MHz टाइमस्केल पर तेजी से दालों interleaves; यह nanosecond-ALEX15 या स्पंदित interleaved उत्तेजना (PIE)16 है. इन सभी दृष्टिकोणों में, स्वीकर्ता लेजर से जानकारी तथाकथित स्टोइकोमेट्री की गणना की ओर ले जाती है, जो कम FRET दक्षता वाले अणुओं और स्वीकर्ता की कमी वाले अणुओं के बीच भेदभाव कर सकती है (या तो अपूर्ण लेबलिंग या फोटोब्लीचिंग के माध्यम से)। पीआईई / एनएस-एलेक्स का उपयोग करना अतिरिक्त रूप से एकल-अणु स्तर पर फ्लोरोसेंट जीवनकाल तक पहुंच प्रदान करता है, और ध्रुवीकरण प्रकाशिकी के साथ युग्मित होने पर अनिसोट्रॉपी को मापा जा सकता है। माप के इस संयोजन को मल्टीपैरामीटर प्रतिदीप्ति का पता लगाने (MFD)9 के रूप में जाना जाता है।
SMFRET के कई फायदों के बावजूद, वाणिज्यिक उपकरणों की उच्च लागत और सरल, स्व-निर्माण विकल्पों की कमी के कारण विशेषज्ञ प्रयोगशालाओं के बाहर इसका व्यापक रूप से उपयोग नहीं किया जाता है। कम लागत वाले ओपनसोर्स माइक्रोस्कोपी के विकास की दिशा में एक बढ़ती प्रवृत्ति हो रही है और अन्य प्लेटफार्म हाल ही में उभरे हैं, जिनमें प्लैंकटनस्कोप 17, ओपनफ्लेक्स्योर माइक्रोस्कोप 18, फ्लेक्सिस्कोप 19, miCube20, liteTIRF21 और Squid22 शामिल हैं। इसमें अध्ययन smfBox का उपयोग करने के लिए प्रोटोकॉल का वर्णन करता है, एक हाल ही में विकसित लागत प्रभावी confocal सेट-अप स्वतंत्र रूप से एकल अणुओं diffusing पर दो रंगों के बीच FRET दक्षता को मापने में सक्षम है। विस्तृत निर्माण निर्देश और सभी आवश्यक परिचालन सॉफ़्टवेयर स्वतंत्र रूप से उपलब्ध हैं: https://craggslab.github.io/smfBox/ 23। SmfBox की ऑप्टिकल व्यवस्था को सस्ती और व्यापक रूप से सुलभ निर्माताओं से खरीदे गए आसानी से उपलब्ध घटकों से इकट्ठा किया जाता है, जबकि माइक्रोस्कोप बॉडी (एक मानक कॉन्फोकल सेट-अप में अधिकांश खर्च के लिए जिम्मेदार) को एक कस्टम लाइट-टाइट एनोडाइज्ड-एल्यूमीनियम बॉक्स द्वारा प्रतिस्थापित किया गया है (परिवेश प्रकाश परिस्थितियों के तहत माप करने की अनुमति देता है)। इस बॉक्स में प्रमुख ऑप्टिकल घटक हैं, जिनमें उत्तेजना डाइक्रोइक, उद्देश्य और पिनहोल, और एक यांत्रिक लेजर इंटरलॉक शामिल हैं, जो कक्षा I लेजर उत्पाद के रूप में अपने सुरक्षित संचालन को सक्षम करते हैं (एक पूर्ण योजनाबद्ध के लिए चित्रा 1 सी देखें)। SmfBox एलेक्स का उपयोग करता है डाई stoichiometry मान्य करने के लिए और सटीक FRET सुधार कारकों को निर्धारित करने के लिए. यह कस्टम-लिखित, ओपन-सोर्स सॉफ़्टवेयर (smOTTER) का उपयोग करके संचालित किया जाता है, जो डेटा अधिग्रहण के सभी पहलुओं को नियंत्रित करता है और कई तृतीय-पक्ष विश्लेषण उपकरणों के साथ संगत ओपन-सोर्स फोटॉन-HDF5 format24 में डेटा आउटपुट करता है। SmfBox और अधिग्रहण और डेटा विश्लेषण प्रोटोकॉल हाल ही में एक बहु-प्रयोगशाला अंधा अध्ययन 25 में >20 अन्य उपकरणों (confocal और TIRF दोनों) के खिलाफ परीक्षण किया गया था। प्राप्त FRET दक्षता अन्य सभी उपकरणों के साथ उत्कृष्ट समझौते में थे, smfBox व्यावसायिक रूप से उपलब्ध सेटअप की कीमत का केवल एक अंश लागत के बावजूद।
यहां, एक चरण-दर-चरण प्रोटोकॉल को प्राप्त करने और विश्लेषण करने के लिए रेखांकित किया गया है, जो कि smfBox का उपयोग करके डीएनए डुप्लेक्स को स्वतंत्र रूप से अलग करने पर सटीक, पूर्ण FRET दक्षताओं को प्राप्त करने और विश्लेषण करने के लिए, सभी तरह से स्विच ऑन से, संरेखण और ध्यान केंद्रित करने के माध्यम से, डेटा संग्रह और विश्लेषण के लिए। यहां उपयोग किए जाने वाले नमूने तीन डुप्लेक्स डीएनए हैं (उच्च, मध्य और निम्न-FRET दक्षताओं को प्रदर्शित करते हुए, तालिका 1 देखें) जो दुनिया भर में अंधे अध्ययन 25 में मूल्यांकन किए गए थे; हालांकि, विधि प्रोटीन और अन्य न्यूक्लिक एसिड सहित कई आणविक प्रणालियों के लिए अनुकूलहै। आशा है कि इस तरह के एक विस्तृत प्रोटोकॉल, smfBox23 के लिए पहले से ही मौजूदा निर्माण निर्देशों के साथ, इस शक्तिशाली तकनीक को प्रयोगशालाओं की एक विस्तृत श्रृंखला के लिए और भी अधिक सुलभ बनाने में मदद करेगा।
Protocol
Representative Results
Discussion
प्रोटोकॉल में सबसे महत्वपूर्ण कदम माइक्रोस्कोप का संरेखण और नमूना एकाग्रता को सही कमजोर पड़ने के लिए समायोजित कर रहे हैं। यदि संरेखण बंद है, तो फटने और प्लॉट हिस्टोग्राम की पहचान करने के लिए अपर्याप्त संकेत हो सकता है, और यदि नमूनों के बीच misalignment होता है तो सटीक FRET सुधार रिसाव और पता लगाने / उत्तेजना क्षमता में परिवर्तन के कारण विफल हो सकता है। एक उपयुक्त एकाग्रता का उपयोग भी महत्वपूर्ण है, बहुत अधिक एकाग्रता संयोग फटने देगी, जिसमें संभावित रूप से अलग-अलग फ्रेट दक्षताओं या लेबलिंग स्टोइकोमेट्री के साथ कई अणु शामिल हैं। बहुत कम एकाग्रता मजबूत डेटा विश्लेषण के लिए बहुत कम फटने देगी।
यहां वर्णित प्रोटोकॉल स्थैतिक एकल FRET प्रजातियों में दूरी को मापने के लिए है। यदि नमूने में FRET दक्षता हिस्टोग्राम में एक से अधिक चोटी है, या चोटियां व्यापक दिखाई देती हैं (जो गतिशील प्रजातियों के साथ हो सकती हैं), तो हिस्टोग्राम को सटीकता की एक ही डिग्री में फिट करने के लिए अधिक फटने की आवश्यकता हो सकती है। दो अच्छी तरह से अलग चोटियों के लिए तो लगभग दो बार के रूप में ज्यादा डेटा की आवश्यकता होगी, लेकिन अगर आबादी थोड़ा ओवरलैप तो और भी अधिक डेटा की आवश्यकता है.
यदि दो आबादी प्रयोग के समय पैमाने पर परस्पर परिवर्तित होती है, तो सिस्टम की गतिशीलता और कैनेटीक्स संभावित रूप से निर्धारित किया जा सकता है। BVA27 और 2CDE28 जैसे परीक्षण इस बात की पुष्टि कर सकते हैं कि मध्यवर्ती फटने प्रकृति में गतिशील हैं, जबकि dPDA29,30 या H2MM31 सहित विश्लेषण अंतर-रूपांतरण की दरों को निर्धारित कर सकते हैं। BVA और 2CDE के लिए Jupyter नोटबुक FRETBursts26 वेबसाइट पर उपलब्ध हैं, और MATLab आधारित सॉफ़्टवेयर PAM32 BVA, 2CDE और PDA विश्लेषण चला सकते हैं।
Confocal एकल अणु FRET आसानी से राज्यों को टीआईआरएफ की तुलना में बहुत अधिक अल्पकालिक (~ 1 एमएस) का निरीक्षण कर सकता है; हालांकि, प्रसार द्वारा सीमित लघु अवलोकन समय, कोई आणविक इतिहास नहीं देते हैं, और इसलिए लंबे समय तक रहने के समय, या जटिल संक्रमण नेटवर्क को उस तरह से निर्धारित नहीं कर सकते हैं जिस तरह से सतह स्थिर प्रयोग कर सकते हैं।
जैसा कि प्रोटोकॉल बहुत कम सांद्रता पर अणुओं को स्वतंत्र रूप से अलग करने के उपाय करता है, यह एक ही अणु पर इंट्रामोलेक्यूलर दूरी को मापते समय सबसे अच्छा काम करता है। क्षणिक रूप से बाध्य अणुओं के बीच अंतर-आणविक दूरी को मापा जा सकता है बशर्ते कि दो अणुओं का केडी पर्याप्त रूप से कम हो कि परिसर प्रयोग द्वारा आवश्यक कम काम करने वाली एकाग्रता (~ 100 पीएम) पर एक महत्वपूर्ण मात्रा में मौजूद हो। यदि केडी इससे बहुत अधिक है, तो केवल एकल लेबल वाले अणुओं को देखा जाएगा। इस समस्या को माइक्रोफ्लुइडिक्स का उपयोग करके दो लेबल घटकों को एक साथ एक उच्च एकाग्रता पर मिश्रण करने के लिए दूर किया जा सकता है और फिर जटिल विघटित होने से पहले उद्देश्य पर तेजी से पतला और बहरहा है33,34।
एकल-अणु स्तर पर FRET दक्षताओं को मापने का पहनावा तकनीकों पर एक महत्वपूर्ण लाभ है, क्योंकि यह विषम उप-आबादी पर सूचित करता है, जो एक पहनावा प्रयोग में औसत होगा। इसके अलावा, एलेक्स के साथ एकल-अणु FRET सटीक FRET दक्षता तक पहुंच प्रदान करता है, जिसे सटीक दूरी में परिवर्तित किया जा सकता है। यह केवल सापेक्ष दूरी परिवर्तनों की जांच करने के बजाय अधिक विस्तृत संरचनात्मक जानकारी के निर्धारण को सक्षम बनाता है। SmfBox इन सभी लाभों और क्षमताओं को वहन करता है, लेकिन confocal smFRET23 के लिए सक्षम तुलनीय व्यावसायिक रूप से उपलब्ध माइक्रोस्कोप की तुलना में बहुत कम बजट पर बनाया जा सकता है।
SmfBox smFRET तकनीकों के लिए प्रवेश करने के लिए एक बहुत कम बाधा का प्रतिनिधित्व करता है, जिससे शोधकर्ताओं को संरचनात्मक परिवर्तनों को मापने की अनुमति मिलती है, और प्रोटीन और न्यूक्लिक एसिड के भीतर और उनके बीच सटीक दूरी 7,8,9,10,11,35।
Disclosures
The authors have nothing to disclose.
Acknowledgements
लेखकों ने निम्नलिखित वित्त पोषण स्रोतों को कृतज्ञतापूर्वक स्वीकार किया है: BBSRC (BB/ T008032/1); EPSRC (बीए के लिए Studentship) और MRC (ए. आर.-टी. के लिए Studentship)।
Materials
| Amino modified oligonucleotide | Eurogentec | N/A | May be ordered from various suppliers or synthesised; amino modification enables labeling with NHS-ester modified dyes |
| Avalanche photodiode (APD) | Excelitas | SPCM-AQRH-14 | Two APDs are required for the smfBox setup |
| Bovine Serum Albumin (BSA) | Merck | A2153 | System dependant; imaging buffer component (0.1 mg/mL in buffer) |
| Compact Laser Combiner | OMICRON | LightHUB-2 | 515 nm (80 mW) and 638 nm (100 mW) lasers |
| Coverglass | VWR | 630-2742 | Thickness: 0.17 ± 0.01 mm, LxW: 22×22 mm |
| Cy3B | Cytiva | PA63101 | 1 mg, PA63100 (5 mg), PA96106 (25 mg) |
| FRETBursts Python Package | N/A | N/A | Open-source python package for burst analysis of freely-diffusing single-molecule FRET data: https://fretbursts.readthedocs.io |
| Imaging Buffer | N/A | System dependant; 5 mM NaCl, 20 mM MgCl2, 5 mM Tris pH 7.5 and 0.1 mg/mL BSA | |
| Immersion Oil | Olympus | IMMOIL-F30CC | |
| Jupyter notebooks | Project Jupyter | N/A | Open-source web application to create and share documents that contain live code, equations, visualizations and text; data analysis notebooks for smfBox can be found in the SI |
| Lens Tissue | ThorLabs | MC-5 | MC-50E is same item in bulk |
| Magnesium Chloride | Merck | M2670 | System dependant; imaging buffer component (20 mM in buffer) |
| MilliQ/Ultrapure water | N/A | ||
| Nanopoistioner | Piezoconcept | FOC300 | Nanopositioner for accurate positioning of microscope objective |
| NHS-ester modified ATTO-550 | ATTO-TEC | AD 550-31 | 1 mg, AD 550-35 (5 mg) |
| NHS-ester modified ATTO-647N | ATTO-TEC | AD 647N-31 | 1 mg, AD 647N-35 (5 mg) |
| Objective lens | Olympus | N1480700 | Olympus objective series from orignal smfBox discontinued; replaced by N5702300 |
| OMICRON Control Center (OCC)- laser control center | OMICRON | N/A | v3.5.34 – OMICRON laser driver software |
| Press-To-Seal silicone isolator | Grace Bio-Labs | 664201 | 8-9 mm Diameter x 1.7 mm Depth |
| smOTTER | N/A | N/A | Open-source acquisition software for the Craggs Lab smfBox: https://github.com/craggslab/smOTTER |
| Sodium Chloride | Merck | S7653 | System dependant; imaging buffer component (5 mM in buffer) |
| Tris base | Merck | 93362 | System dependant; imaging buffer component (5 mM, pH 7.5 in buffer) |
| Type I ultrapure water | Merck | ZIQ7000T0 | Milli-Q® IQ 7000 Ultrapure Water System |
References
- Forster, T. Intermolecular energy migration and fluorescence. Annals of Physics. 437 (1-2), 55-75 (1948).
- Stryer, L., Haugland, R. P. Energy transfer: a spectroscopic ruler. Proceedings of the National Academy of Sciences. 58 (2), 719-726 (1967).
- Hohlbein, J., et al. Conformational landscapes of DNA polymerase I and mutator derivatives establish fidelity checkpoints for nucleotide insertion. Nature Communications. 4 (1), 2131 (2013).
- Lerner, E., et al. Toward dynamic structural biology: Two decades of single-molecule Förster resonance energy transfer. Science. 359 (6373), (2018).
- Hohlbein, J., Craggs, T. D., Cordes, T. Alternating-laser excitation: single-molecule FRET and beyond. Chemical Society Reviews. 43 (4), 1156-1171 (2014).
- Lerner, E., et al. The FRET-based structural dynamics challenge — community contributions to consistent and open science practices. arXiv. , (2020).
- Hellenkamp, B., Wortmann, P., Kandzia, F., Zacharias, M., Hugel, T. Multidomain structure and correlated dynamics determined by self-consistent FRET networks. Nature Methods. 14 (2), 174-180 (2017).
- Craggs, T. D., et al. Substrate conformational dynamics facilitate structure-specific recognition of gapped DNA by DNA polymerase. Nucleic Acids Research. 47 (20), 10788-10800 (2019).
- Tsytlonok, M., et al. Dynamic anticipation by Cdk2/Cyclin A-bound p27 mediates signal integration in cell cycle regulation. Nature Communications. 10 (1), 1676 (2019).
- Nagy, J., et al. Complete architecture of the archaeal RNA polymerase open complex from single-molecule FRET and NPS. Nature Communications. 6 (1), 6161 (2015).
- LeBlanc, S. J., et al. Coordinated protein and DNA conformational changes govern mismatch repair initiation by MutS. Nucleic Acids Research. 46 (20), 10782-10795 (2018).
- Segal, M., et al. High-throughput smFRET analysis of freely diffusing nucleic acid molecules and associated proteins. Methods. 169, 21-45 (2019).
- Kapanidis, A. N., et al. Fluorescence-aided molecule sorting: Analysis of structure and interactions by alternating-laser excitation of single molecules. Proceedings of the National Academy of Sciences. 101 (24), 8936-8941 (2004).
- Kapanidis, A. N., et al. Alternating-laser excitation of single molecules. Accounts of Chemical Research. 38 (7), 523-533 (2005).
- Müller, B. K., Zaychikov, E., Brauchle, C., Lamb, D. C. Pulsed interleaved excitation. Biophysical Journal. 89 (5), 3508-3522 (2005).
- Laurence, T. A., Kong, X., Jager, M., Weiss, S. Probing structural heterogeneities and fluctuations of nucleic acids and denatured proteins. Proceedings of the National Academy of Sciences of the United States of America. 102 (48), 17348-17353 (2005).
- Pollina, T., et al. PlanktonScope: Affordable modular imaging platform for citizen oceanography. bioRxiv. , 056978 (2020).
- Collins, J. T., et al. Robotic microscopy for everyone: the OpenFlexure microscope. Biomedical Optics Express. 11 (5), 2447-2460 (2020).
- Courtney, A., Alvey, L. M., Merces, G. O. T., Burke, N., Pickering, M. The Flexiscope: a low cost, flexible, convertible and modular microscope with automated scanning and micromanipulation. Royal Society Open Science. 7 (3), 191949 (2020).
- Martens, K. J. A., et al. Visualisation of dCas9 target search in vivo using an open-microscopy framework. Nature Communications. 10 (1), 3552 (2019).
- Auer, A., et al. Nanometer-scale multiplexed super-resolution imaging with an economic 3D-DNA-PAINT microscope. ChemPhysChem. 19 (22), 3024-3034 (2018).
- Li, H., et al. Squid: Simplifying quantitative imaging platform development and deployment. bioRxiv. , 424613 (2020).
- Ambrose, B., et al. The smfBox is an open-source platform for single-molecule FRET. Nature Communications. 11 (1), 5641 (2020).
- Ingargiola, A., Laurence, T., Boutelle, R., Weiss, S., Michalet, X. Photon-HDF5: An open file format for timestamp-based single-molecule fluorescence experiments. Biophysical Journal. 110 (1), 26-33 (2016).
- Hellenkamp, B., et al. Precision and accuracy of single-molecule FRET measurements-a multi-laboratory benchmark study. Nature Methods. 15 (9), 669-676 (2018).
- Ingargiola, A., Lerner, E., Chung, S., Weiss, S., Michalet, X. FRETBursts: An open source toolkit for analysis of freely-diffusing single-molecule FRET. PLOS One. 11 (8), 0160716 (2016).
- Torella, J. P., Holden, S. J., Santoso, Y., Hohlbein, J., Kapanidis, A. N. Identifying molecular dynamics in single-molecule FRET experiments with burst variance analysis. Biophysical Journal. 100 (6), 1568-1577 (2011).
- Tomov, T. E., et al. Disentangling subpopulations in single-molecule FRET and ALEX experiments with photon distribution analysis. Biophysical Journal. 102 (5), 1163-1173 (2012).
- Santoso, Y., Torella, J. P., Kapanidis, A. N. Characterizing single-molecule FRET dynamics with probability distribution analysis. ChemPhysChem. 11 (10), 2209-2219 (2010).
- Kalinin, S., Valeri, A., Antonik, M., Felekyan, S., Seidel, C. A. M. Detection of structural dynamics by FRET: A photon distribution and fluorescence lifetime analysis of systems with multiple states. The Journal of Physical Chemistry B. 114 (23), 7983-7995 (2010).
- Pirchi, M., et al. Photon-by-photon hidden Markov model analysis for microsecond single-molecule FRET kinetics. The Journal of Physical Chemistry B. 120 (51), 13065-13075 (2016).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. PAM: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophysical Journal. 114 (7), 1518-1528 (2018).
- Zijlstra, N., et al. Rapid microfluidic dilution for single-molecule spectroscopy of low-affinity biomolecular complexes. Angewandte Chemie International Edition. 56 (25), 7126-7129 (2017).
- Hellenkamp, B., Thurn, J., Stadlmeier, M., Hugel, T. Kinetics of transient protein complexes determined via diffusion-independent microfluidic mixing and fluorescence stoichiometry. The Journal of Physical Chemistry B. 122 (49), 11554-11560 (2018).
- Bennet, I. A., et al. Regional conformational flexibility couples substrate specificity and scissile phosphate diester selectivity in human flap endonuclease 1. Nucleic Acids Research. 46 (11), 5618-5633 (2018).

