Driedimensionaal in vitro biomimetisch model van neuroblastoom met behulp van op collageen gebaseerde steigers
Summary
Dit artikel somt de stappen op die nodig zijn om neuroblastoomcellijnen te zaaien op eerder beschreven driedimensionale op collageen gebaseerde steigers, de celgroei gedurende een vooraf bepaald tijdsbestek te behouden en steigers op te halen voor verschillende celgroei- en celgedragsanalyses en stroomafwaartse toepassingen, aanpasbaar om te voldoen aan een reeks experimentele doelen.
Abstract
Neuroblastoom is de meest voorkomende extracraniële solide tumor bij kinderen, goed voor 15% van de totale sterfgevallen door kinderkanker. Het oorspronkelijke tumorweefsel is een complexe driedimensionale (3D) micro-omgeving met lagen kankercellen en niet-kankercellen omgeven door een extracellulaire matrix (ECM). De ECM biedt fysieke en biologische ondersteuning en draagt bij aan ziekteprogressie, prognose van de patiënt en therapeutische respons.
Dit artikel beschrijft een protocol voor het samenstellen van een op 3D-steigers gebaseerd systeem om de micro-omgeving van neuroblastoom na te bootsen met behulp van neuroblastoomcellijnen en op collageen gebaseerde steigers. De steigers worden aangevuld met nanohydroxyapatiet (nHA) of glycosaminoglycanen (GAG’s), die van nature in hoge concentraties worden aangetroffen in het bot en het beenmerg, de meest voorkomende metastatische plaatsen van neuroblastoom. De 3D poreuze structuur van deze steigers maakt de hechting, proliferatie en migratie van neuroblastoomcellen en de vorming van celclusters mogelijk. In deze 3D-matrix is de celrespons op therapieën meer een afspiegeling van de in vivo situatie.
Het op steigers gebaseerde kweeksysteem kan hogere celdichtheden handhaven dan conventionele tweedimensionale (2D) celkweek. Daarom zijn optimalisatieprotocollen voor het initiële zaaien van celaantallen afhankelijk van de gewenste experimentele tijdschema’s. Het model wordt gemonitord door celgroei te beoordelen via DNA-kwantificering, cellevensvatbaarheid via metabole assays en celdistributie binnen de steigers via histologische kleuring.
De toepassingen van dit model omvatten de beoordeling van gen- en eiwitexpressieprofielen en cytotoxiciteitstesten met behulp van conventionele geneesmiddelen en miRNA’s. Het 3D-kweeksysteem maakt de nauwkeurige manipulatie van cel- en ECM-componenten mogelijk, waardoor een omgeving wordt gecreëerd die fysiologisch meer lijkt op het oorspronkelijke tumorweefsel. Daarom zal dit 3D in vitro model het begrip van de pathogenese van de ziekte vergroten en de correlatie verbeteren tussen resultaten verkregen in vitro, in vivo in diermodellen en menselijke proefpersonen.
Introduction
Neuroblastoom is een pediatrische kanker van het sympathische zenuwstelsel die ontstaat tijdens de embryonale ontwikkeling of het vroege postnatale leven als gevolg van de transformatie van neurale topcellen1. Het is de meest voorkomende solide extracraniale tumor bij kinderen, die 8% van de maligniteiten vertegenwoordigt die worden gediagnosticeerd bij patiënten jonger dan 15 jaar en is verantwoordelijk voor 15% van alle sterfgevallen door kanker bij kinderen. De ziekte vertoont zeer heterogeen klinisch gedrag als gevolg van specifieke chromosomale, genetische en epigenetische veranderingen, en histopathologische kenmerken.
Deze veranderingen dragen bij aan de agressiviteit van neuroblastoom en slechte resultaten bij pediatrische patiënten. Vandaar dat de huidige therapieën op de lange termijn niet effectief blijken te zijn voor bijna 80% van de patiënten met de klinisch agressieve ziekte2, wat benadrukt dat de behandeling voor deze groep patiënten een uitdaging blijft. Dit is waarschijnlijk te wijten aan het feit dat de mechanismen van neuroblastoomheterogeniteit en metastasen nog steeds niet volledig worden begrepen. Er wordt nu echter algemeen aangenomen dat de tumormicro-omgeving (TME) een rol speelt bij de progressie van veel kankers; Toch blijft het onderbelicht in neuroblastoom 3,4.
De natuurlijke TME is een complexe 3D-micro-omgeving met kankercellen en niet-kankercellen omgeven door een ECM. De ECM verwijst naar de acellulaire component van een weefsel die structurele en biochemische ondersteuning biedt aan de cellulaire bewoners en bijdraagt aan ziekteprogressie, prognose van de patiënt en therapeutische respons5. Deze bevordering van ziekteprogressie is te wijten aan “dynamische wederkerigheid” of voortdurende bidirectionele communicatie tussen cellen en de ECM 6,7,8. Naarmate kanker vordert, wordt stromaal collageen gereorganiseerd, vaak in lineaire patronen loodrecht op de stroma-kankerinterface, die kankercellen gebruiken als een migratieroute naar metastase 9,10,11.
De belangrijkste componenten van deze natuurlijke functionele biologische steiger zijn een vezelig netwerk van collagenen type I en II en andere eiwitten, waaronder elastine, glycoproteïnen zoals laminine, evenals een reeks proteoglycanen en andere oplosbare componenten12,13. Deze eiwitten van het natuurlijke ECM zijn nu aantrekkelijke natuurlijke biomoleculen geworden voor het ontwikkelen van 3D in vitro modellen3. De toepassing van 3D-steigers voor in vitro celkweek wordt steeds populairder vanwege de grotere fysiologische weergave van de TME in vergelijking met traditionele 2D-monolaagcultuur. De gefabriceerde 3D-steigers helpen bij celhechting, proliferatie, migratie, metabolisme en reactie op stimuli die worden gezien in in vivo biologische systemen.
Het hoofdbestanddeel van deze 3D-steigers is collageen, dat een belangrijke speler is in veel normale biologische processen, waaronder weefselherstel, angiogenese, weefselmorfogenese, celadhesie enmigratie11. Op collageen gebaseerde 3D-matrices hebben hun robuuste functionaliteit getoond om ECM te modelleren, en dienen als een in vitro biomimetische micro-omgeving en tegelijkertijd cel-ECM-interacties mogelijk maken, evenals celmigratie en -invasie. Deze 3D-matrices bieden ook een nauwkeurigere analyse van de celrespons op chemotherapeutische geneesmiddelen dan traditionele 2D- of “platte” cultuur in veel kankermodellen 14,15,16, waaronder neuroblastoom 17,18. Genetische analyse van 3D-celculturen heeft een hogere correlatie met het menselijke weefselprofiel gerapporteerd, zelfs in vergelijking met diermodellen19. Over het algemeen is de hoeksteen van deze 3D-steigers om cellen een geschikte in vitro-omgeving te bieden, die de oorspronkelijke weefselarchitectuur recapituleert en bidirectionele moleculaire overspraak vergemakkelijkt8.
Om de complexiteit van op collageen gebaseerde modellen te vergroten, worden andere gemeenschappelijke ECM-componenten opgenomen in het weefselmanipulatieproces, waardoor meer fysiologisch relevante modellen worden gecreëerd om niche-TME’s van verschillende weefsels weer te geven. GAG’s, negatief geladen polysacchariden die aanwezig zijn in alle zoogdierweefsels20, vergemakkelijken bijvoorbeeld celhechting, migratie, proliferatie en differentiatie. Chondroïtinesulfaat is een specifiek type GAG dat wordt aangetroffen in het bot en kraakbeen, dat eerder is gebruikt in weefselmanipulatietoepassingen voor botherstel 21,22,23,24,25. Nanohydroxyapatiet (nHA) is het belangrijkste anorganische bestanddeel van de minerale samenstelling van menselijk botweefsel, dat tot 65 gewichtsprocent van het bot uitmaakt26 en wordt daarom veel gebruikt voor botvervanging en -regeneratie27. GAG’s en nHA zijn dus aantrekkelijke composieten voor het reconstrueren van de primaire neuroblastoom-ECM en het modelleren van de meest voorkomende metastatische plaatsen van neuroblastoom, beenmerg (70,5%) en bot (55,7%)28.
Scaffolds waarin deze ECM-componenten zijn verwerkt, zijn oorspronkelijk ontwikkeld voor toepassingen op het gebied van botweefselengineering met uitgebreide analyse van hun biocompatibiliteit, toxiciteit en osteoconductieve en osteoinductieve eigenschappen29,30. Het zijn poreuze, op collageen gebaseerde matrices die worden geproduceerd met behulp van vriesdroogtechnieken om hun fysische en biologische eigenschappen te controleren. De collageensteigers aangevuld met nHA (Coll-I-nHA) of chondroïtine-6-sulfaat (Coll-I-GAG) toonden succes aan bij het nabootsen van de primaire TME bij borstkanker31 en metastase naar bot bij prostaatkanker15 en neuroblastoom17. De vriesdroogtechniek die wordt gebruikt om deze composietsteigers te vervaardigen, levert reproduceerbare homogeniteit in poriegrootte en porositeit op binnen de steigers22,23,24. In het kort wordt een collageenslurry (0,5 gew.%) vervaardigd door fibrillair collageen te mengen met 0,05 M azijnzuur. Voor Coll-I-GAG wordt tijdens het mengen 0,05 gew.% chrondoitine-6-sulfaat geïsoleerd uit haaienkraakbeen toegevoegd aan de collageensuspensie. Voor de composiet Coll-I-nHA-steigers worden hydroxyapatietdeeltjes van nanoformaat gesynthetiseerd zoals eerder beschreven27 en toegevoegd aan de collageensuspensie in een verhouding van 2:1 tot het gewicht van het collageen tijdens het mengproces. Alle steigers worden fysiek verknoopt en gesteriliseerd met behulp van een dehydrothermale behandeling bij 105 °C gedurende 24 uuren 25 minuten. Cilindrische steigers (6 mm diameter, 4 mm hoogte) worden verkregen met behulp van een biopsiepons en kunnen chemisch worden verknoopt met 3 mM N-(3-dimethylaminopropyl)-N’-ethylcarbodiimidehydrochloride en 5,5 mM N-hydroxysuccinimide (EDAC/NHS) in gedestilleerd water (dH2O) om de mechanische eigenschappen van de constructies te verbeteren30. Dit goed geoptimaliseerde productieproces van twee collageensteigers creëert steigers met reproduceerbare mechanische eigenschappen, waaronder poriegrootte, porositeit en stijfheid (kPa). Zowel Coll-I-GAG- als Coll-I-nHA-steigers hebben verschillende fysische eigenschappen, waardoor verschillende omgevingsomstandigheden ontstaan. De eigenschappen van elke steiger worden weergegeven in tabel 1.
| Coll-I-GAG | Coll-I-nHA | |
| Steiger Grootte (diameter [mm] x hoogte [mm]) |
6 x 4 17 | 6 x 4 17 |
| Collageenconcentratie (gew. %) | 0,5 17 | 0,5 17 |
| Substraatconcentratie (gew. %) [op basis van het gewicht van collageen] |
0,05 15,17 | 200 17 |
| Gemiddelde poriegrootte (mm) | 96 22 | 96 – 120 29 |
| Porositeit (%) | 99,5 23 | 98,9 – 99,4 27 |
| Stijfheid (kPa) | 1.5 27 | 5,5 – 8,63 29 |
Tabel 1: Overzicht van de mechanische eigenschappen van de twee steigers die zijn gebruikt voor het bestuderen van de biologie van neuroblastoom.
Dit artikel beschrijft een protocol voor het samenstellen van een op 3D-steigers gebaseerd systeem om de micro-omgeving van neuroblastoom beter na te bootsen met behulp van neuroblastoomcellijnen en eerder beschreven op collageen gebaseerde steigers aangevuld met nHA (Coll-I-nHA) of chondroïtine-6-sulfaat (Coll-I-GAG). Het protocol omvat stroomafwaartse methoden om de groeimechanismen van de neuroblastoomcellen in een meer fysiologisch relevante omgeving te analyseren met behulp van eerder geoptimaliseerde goedkope methoden die zijn aangepast aan 2D-monolaagkweek Figuur 1.
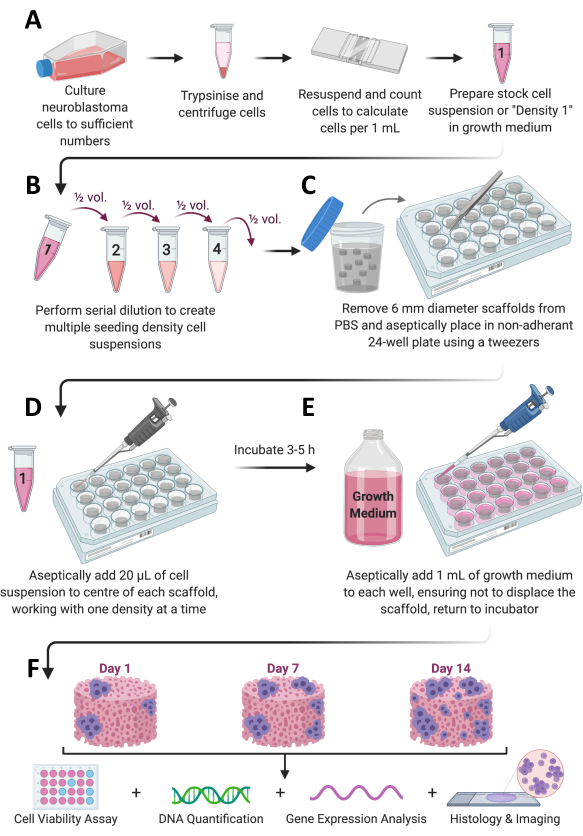
Figuur 1: Algemene protocolworkflow. (A) Cellen worden tot voldoende aantallen gekweekt, gesplitst, geteld en opnieuw gesuspendeerd in een geschikte hoeveelheid medium. (B) Dit celmateriaal ondergaat vervolgens seriële verdunning om in totaal 4 celsuspensies van verschillende dichtheden te bereiden. (C) Op collageen gebaseerde steigers worden steriel geplateerd in niet-klevende platen met 24 putjes, en (D) 20 μL celsuspensie wordt toegevoegd aan het midden van elke steiger en gedurende 3-5 uur laten incuberen bij 37 °C, 5% CO2 en 95% vochtigheid. (E) Volledig groeimedium (1 ml) wordt vervolgens langzaam aan elke steiger toegevoegd en de platen worden teruggeplaatst in de incubator om celgroei gedurende het gewenste tijdsbestek mogelijk te maken. (F) Op elk vooraf bepaald tijdstip worden verschillende steigers opgehaald voor beoordeling van de levensvatbaarheid en groei van cellen, analyse van genexpressie en histologische kleuring. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
Het 3D-scaffold-kankercelmodel heeft bewezen een waardevol en veelzijdig hulpmiddel te zijn voor het verkrijgen van mechanistisch inzicht in de groei, levensvatbaarheid en infiltratie van neuroblastoomcellen in een vereenvoudigde TME32. Het hier beschreven 3D-neuroblastoommodel bootst de minimale TME na en levert meer fysiologisch relevante gegevens op dan een 2D-monolaagcultuur. Een groot nadeel van 3D-celkweek is de toegenomen experimentele complexiteit en langere tijdsbestekken. Hier wordt een geoptimaliseerd protocol beschreven voor het zaaien, groeien en onderhouden van neuroblastoomcellen op op collageen gebaseerde steigers, gevolgd door stroomafwaartse analyses en toepassingen, wat een robuuste karakterisering van celgroei oplevert. We wilden inzicht krijgen in de optimale celseeding-dichtheid voor de steigers om een voorspelbare en controleerbare omgeving te creëren voor het beoordelen van behandelingen tegen kanker in een snel experimenteel venster van 14 dagen. De combinatie van al deze beschreven eenvoudige protocollen biedt een goed afgeronde beoordeling van de groei van neuroblastoomcellen in het op steigers gebaseerde in-vitrokweeksysteem .
De kritieke punten in de opzet van het protocol zijn benadrukt om wetenschappers in staat te stellen dit snel in hun laboratoria vast te stellen. De aangegeven incubatietijden voor betere prestaties van de colorimetrische cellevensvatbaarheidstest maken bijvoorbeeld een diepere penetratie van het reagens in de poriën van de steiger mogelijk om alle cellen te bereiken. Bovendien is de fluorescerende dsDNA-kleuringstechniek robuust en eenvoudig; het vrijkomen van DNA uit de steigers vereist echter krachtige cellyse, omdat de cellen ‘gevangen’ zitten in collageenvezels.
Met behulp van de beschreven eenvoudige DNA-kwantificeringstest kunnen we met behulp van dit model de loggroeifase identificeren op op collageen gebaseerde steigers voor screening op geneesmiddelen tegen kanker. In de beschreven experimentele setting werden 4 initiële celzaaidichtheden gebruikt met een totale periode van 14 dagen en analysetijdstippen op dag 1, 7 en 14. We hebben vastgesteld dat KellyLuc-cellen die op 4 × 105 cellen/scaffold zijn gezaaid, het meest significant actieve proliferatievenster hebben tussen dag 7 en 14. Deze groeigegevens van de logfase zullen een betrouwbare interpretatie van verschillende celcytotoxiciteitsexperimenten mogelijk maken. Het elimineert speculatie over een afname van de groei of celdood als gevolg van onderdrukte groei op het poreuze 3D-platform in plaats van door toxiciteit van geneesmiddelen. De levensvatbaarheid van cellen is ook een veelgebruikte beoordeling voor de geschiktheid van 3D-platforms om de groei van verschillende celtypen te ondersteunen33,34. Hoewel er veel tests zijn om de levensvatbaarheid van cellen te meten, waaronder levende/dode kleuring, ATP-meting, proliferatietests, vonden we het gebruik van de Alamar Blue colorimetrische cellevensvatbaarheidstest een eenvoudige en effectieve techniek om DNA-kwantificeringsgegevens te ondersteunen.
Het gecombineerde gebruik van DNA-kwantificering en cellevensvatbaarheid leverde complementair bewijs dat de optimale dichtheid om cellen op het schavot te zaaien om gedurende een periode van 14 dagen verdere groei te bereiken, gemiddeld 2-4 × 105 cellen/steiger is. Dit protocol kan echter eenvoudig worden aangepast om te voldoen aan verschillende experimentele tijdschema’s, analysetijdstippen en stroomafwaartse toepassingen. Hoewel dit protocol de evaluatie van monocultuurcelgroei van neuroblastoomcellen op steigers beschrijft, zijn de steigers gemakkelijk aanpasbaar voor gebruik als platform voor co-cultuur, beschreven door do Amaral et al., die collageen-GAG-steigers gebruikten om keratinocyten en fibroblasten samen te kweken in een onderzoek naar wondgenezing35.
Het beschreven 3D-model maakt het mogelijk om celgroei en -infiltratie te visualiseren met behulp van verschillende bekende technieken, zoals immunofluorescentie en standaard H&E. Het is belangrijk om de cellen samen met de karakterisering van de groei te visualiseren met behulp van biochemische assays vanwege de diversiteit van de celmorfologie en groeipatronen op steigers. Inzicht in het groeipatroon kan inzicht geven in groeigedrag en toekomstige respons op geneesmiddelen tegen kanker. IMR32-groei met behulp van DNA-kwantificering levert bijvoorbeeld vergelijkbare patronen op als Kelly, hoewel IMR32 bij visualisatie met behulp van H&E in grotere clusters groeit dan Kelly, die meer verspreide groei vertoonde (Figuur 9). Deze gevarieerde groeipatronen van cellijnen in steigers weerspiegelen het klinische scenario van tumorheterogeniteit. Het onderzoeken van de respons op geneesmiddelen tegen kanker met behulp van een panel van cellijnen met verschillende morfologieën in 3D-steigers zal de voorspellende waarde voor de respons van de patiënt op dezelfde geneesmiddelen vergroten.
Detectie van gen- of eiwitexpressie kan ook worden uitgevoerd met behulp van andere benaderingen zoals RT-qPCR of ELISA als het eiwit van belang wordt uitgescheiden. Een surrogaatmarker van neuroblastoomprogressie, chromogranine A (CgA)36, werd gebruikt om de groei van neuroblastoomcellen in 3D extra te karakteriseren. Zoals beschreven in eerder werk17, nam de CgA-secretie toe naarmate cellen zich vermenigvuldigden (Figuur 10). Hoewel monolaagse celkweek deze toename niet kon opvangen, omdat proliferatie betekende dat cellen volledige samenvloeiing bereikten in de kweekschalen, maakte het gebruik van de 3D-collageensteigers een langdurige beoordeling van de CgA-secretie mogelijk.
Dit 3D in vitro model is mogelijk niet geschikt voor alle onderzoeksvragen om de biologie van neuroblastoom en de respons op therapieën te bestuderen. Een van de beperkingen is ongelijke celpenetratie in steigers en de vorming van celclusters van verschillende grootte, die afhankelijk is van een bepaalde cellijn en kan leiden tot oncontroleerbare diffusie van voedingsstoffen en testmedicijnen. Deze eigenschap beïnvloedt de robuustheid van therapeutische screening. Ondanks deze beperking is het echter belangrijk om te bedenken dat inheemse tumoren ook heterogeen zijn in grootte en verdeling van kankercellen en vele andere celtypen in het tumorweefsel bevatten. Om deze beperking te overwinnen, stellen we voor om elke met cellen bevolkte steiger te gebruiken als een enkel microweefsel waarvoor de volgende parameters zullen worden geoptimaliseerd: (a) incubatietijden voor het cellevensvatbaarheidsreagens om de cellen en celclusters te bereiken, en (b) lysing van de cellen in Triton X-100-buffer door voorbewerking van cellen op steigers met een weefsellyser om het DNA van de cellen diep in de steiger vrij te maken.
Een andere technische beperking van dit protocol is het ontbreken van mechanische tests van elke batch nieuw vervaardigde steigers voor dit model. Het gebruik van het robuuste productieproces van de steigers, die uitgebreid zijn gekarakteriseerd met betrekking tot fysische en chemische eigenschappen van de steigers, zoals druk- en trekmodulus, porositeit en visuele poriënstructuur en homogeniteit, zorgt er echter voor dat de kwaliteiten van de steiger behouden blijven door batches 21,24,27,30,37.
Samenvattend presenteert dit artikel een reeks eenvoudige methoden voor de analyse van cellulaire groei op op collageen gebaseerde steigers. Zowel de experimentele tijdlijn als de analysepunten kunnen worden uitgewisseld, afhankelijk van de specifieke onderzoeksvragen. Dit protocol is ook aanpasbaar aan andere celtypen. De hierboven getoonde resultaten leveren bewijs over hoe deze compilatie van methoden inzicht gaf in de optimale zaaidichtheid voor verschillende neuroblastoomcellijnen om continue groei gedurende 14 dagen te creëren. De samenvoeging van resultaten verkregen uit alle methoden in dit protocol levert een superieur begrip op van celgroei binnen de 3D-collageenmatrix. Toekomstig gebruik van dit model zal waarschijnlijk co-cultuursystemen omvatten die specifiek zijn voor het neuroblastoom TME en het testen van verschillende nieuwe geneesmiddelen tegen kanker.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Dit werk werd ondersteund door het National Children’s Research Centre (NCRC), Irish Research Council (IRC) en Neuroblastoma UK. De illustraties zijn gemaakt met behulp van BioRender.
Materials
| Cells | |||
| IMR-32 | ATCC | CCL-127 | |
| Kelly | ECACC | 82110411 | |
| KellyCis83 | Made in lab – derived from Kelly (Piskareva et al., 2015) | – | Increasing exposure to cisplatin. Cross resistance acquired |
| SH-SY5Y | ATCC | CRL-2266 | |
| Disposable | |||
| 0.22 µm syringe filter | Millex | SLHP033RS | |
| 1.5 mL Eppendorf tube | Eppendorf | 0030 120.086 | |
| 100 mL sterile Pot | Starstedt | – | |
| 10 mL plastic pipette | Cellstar | 607 180 | |
| 15 mL Falcon tube | Starstedt | 62.554.502 | |
| 25 mL plastic pipette | Cellstar | 760 180 | |
| 50 mL Falcon tube | Starstedt | 62.547.254 | |
| 5 mL plastic pipette | Cellstar | 606 180 | |
| 6 mm Biopsy punches | Kai Medical | BP-60F | |
| Aluminium foil | – | – | |
| Cover Slip | Menzel-Glaser | – | |
| HYPERflask | Corning | CLS10030 | |
| Microscope slides | Thermo Scientific | J1840AMNT | |
| Opaque black 96-well plate | Costar | 3915 | |
| Sterile P10 tips | Starlab | S1121-3810 | |
| Sterile P1000 tips | Starlab | S1122-1830 | |
| Sterile P20 tips | Starlab | S1123-1810 | |
| Sterile P200 tips | Starlab | S1120-8810 | |
| T-175 (175 cm2 flask) | Sarstedt | 83.3912 | |
| T-75 (75 cm2 flask) | Sarstedt | 83.3911.302 | |
| Translucent clear 96 well plate | Cellstar | 655180 | |
| Translucent non-adherent 24 well plates | Cellstar | 83.3922.500 | |
| Equipment | |||
| Autoclave | Astell | – | |
| Automatic tissue processor | Leica | TP1020 | |
| Centrifuge 5804 | Eppendorf | – | |
| Hemocytometer | Hausser Scientific | – | |
| Incubator | ThermoScientific | – | |
| Microtome | Leica | RM2255 | |
| Oven | Memmert | Calibrated by: Cruinn diagnostics Ltd | |
| P10 pipette | Gilson | ||
| P100 pipette | Gilson | ||
| P1000 pipette | Gilson | ||
| P20 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| P200 pipette | Gilson | Calibrated by: Cruinn diagnostics Ltd | |
| Paraffin section flotation bath | Electrothermal | MH8517 | Calibrated by: Cruinn diagnostics Ltd |
| Pipette electronic dispenser | Corning | StripipetterUltra | Calibrated by: Cruinn diagnostics Ltd |
| Plate cooler | Leica | EG1140C | Calibrated by: Cruinn diagnostics Ltd |
| Refrigerator -20 °C | Liebherr | – | |
| Refrigerator -80 °C | Liebherr | – | |
| Refrigerator 4 °C | Liebherr | – | |
| Seesaw Rocker | DLAb | SK-D1807-E | |
| Spectrophotometer – Victor3V Platereader | PerkinElmer | 1420 | |
| Tissue culture hood/Laminar flow hood | GMI | 8038-30-1044 | |
| Tissue Lyser | Qiagen | TissueLyser LT | |
| Tweezers | – | – | |
| Water bath | Grant | – | |
| Wax embedder | Leica | EG1140H | |
| Materials | |||
| 1 L Water | Adrona – Biosciences | 568 | |
| 1% Triton-X | Sigma Aldrich | 9002-93-1 | |
| 10x PBS tablets | Sigma Aldrich | P4417-100TAB | |
| 37% paraformaldehyde | Sigma-Aldrich | F8775 | |
| Alamar Blue Cell Viability Reagent | Invitrogen | DAL1100 | |
| Collagen- glycosaminoglycan scaffold | Tissue engineering research group (TERG) | ||
| Collagen-nanohydroxyapatite scaffold | Tissue engineering research group (TERG) | ||
| dH20 | Adrona – Biosciences | 568 | |
| Eosin | Sigma-Aldrich | E4009 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| EtOH | Sigma-Aldrich | 1.00983.2500 | Made as per: (Cunniffe et al., 2010, Fitzgerald et al., 2015; O’Brien et al., 2005) |
| F12 | Gibco | 21765-029 | |
| FBS | Gibco | 10270-106 | |
| Hemaytoxylin | Sigma-Aldrich | HHS32-1L | |
| L-Glutamine | Gibco | 25030-024 | |
| MEM | Gibco | 21090-022 | |
| miRNA easy Kit | Qiagen | 217004 | |
| MNEAA’s | Gibco | 11140-035 | |
| Penicillin/streptomycin | Gibco | 015140-122 | |
| Qiazol | Qiagen | 79306 | |
| Quant-iT PicoGreen dsDNA Assay Kit | Invitrogen | P11496 | |
| RPMI | Gibco | 21875-034 | |
| Sodium bicarbonate | Sigma Aldrich | S7795-500G | |
| Tissue embedding Medium | Sigma | A6330-4LB | |
| Trypsin-EDTA | Gibco | 25300-054 | |
| Software | |||
| Excel | – | Excel 2016 | |
| ImageJ | – | – | |
| Prism | – | Version 9 | |
References
- Davidoff, A. M. Neuroblastoma. Seminars in Pediatric Surgery. 21 (1), 2-14 (2012).
- Matthay, K. K., et al. Neuroblastoma. Nature Reviews Disease Primers. 2, 16078 (2016).
- Costard, L. S., Hosn, R. R., Ramanayake, H., O’Brien, F. J., Curtin, C. M. Influences of the 3D microenvironment on cancer cell behaviour and treatment responsiveness: a recent update on lung, breast and prostate cancer models. Acta Biomaterialia. , (2021).
- Borriello, L., Seeger, R. C., Asgharzadeh, S., Declerck, Y. A. More than the genes, the tumor microenvironment in neuroblastoma. Cancer Letters. 380 (1), 304-318 (2016).
- Walker, C., Mojares, E., Del Río Hernández, A. Role of extracellular matrix in development and cancer progression. International Journal of Molecular Sciences. 19 (10), 3028 (2018).
- Bissell, M. J., Hall, H. G., Parry, G. How does the extracellular matrix direct gene expression. Journal of Theoretical Biology. 99 (1), 31-68 (1982).
- Schultz, G. S., Davidson, J. M., Kirsner, R. S., Bornstein, P., Herman, I. M. Dynamic reciprocity in the wound microenvironment. Wound Repair and Regeneration. 19 (2), 134-148 (2011).
- Brancato, V., Oliveira, J. M., Correlo, V. M., Reis, R. L., Kundu, S. C. Could 3D models of cancer enhance drug screening. Biomaterials. 232, 119744 (2020).
- Provenzano, P. P., et al. Collagen density promotes mammary tumor initiation and progression. BMC Medicine. 6, 11 (2008).
- Provenzano, P. P., et al. Collagen reorganization at the tumor-stromal interface facilitates local invasion. BMC Medicine. 4, 38 (2006).
- Ouellette, J. N., et al. Navigating the collagen jungle: The biomedical potential of fiber organization in cancer. Bioengineering. 8 (2), 1-19 (2021).
- Kreger, S. T., Voytik-Harbin, S. L. Hyaluronan concentration within a 3D collagen matrix modulates matrix viscoelasticity, but not fibroblast response. Matrix Biology. 28 (6), 336-346 (2009).
- Frantz, C., Stewart, K. M., Weaver, V. M. The extracellular matrix at a glance. Journal of Cell Science. 123 (24), 4195-4200 (2010).
- Hume, R. D., et al. Tumour cell invasiveness and response to chemotherapeutics in adipocyte invested 3D engineered anisotropic collagen scaffolds. Scientific Reports. 8 (1), 12658 (2018).
- Fitzgerald, K. A., et al. The use of collagen-based scaffolds to simulate prostate cancer bone metastases with potential for evaluating delivery of nanoparticulate gene therapeutics. Biomaterials. 66, 53-66 (2015).
- Sapudom, J., Pompe, T. Biomimetic tumor microenvironments based on collagen matrices. Biomaterials Science. 6 (8), 2009-2024 (2018).
- Curtin, C., et al. A physiologically relevant 3D collagen-based scaffold-neuroblastoma cell system exhibits chemosensitivity similar to orthotopic xenograft models. Acta Biomaterialia. 70, 84-97 (2018).
- Gavin, C., et al. Neuroblastoma invasion strategies are regulated by the extracellular matrix. Cancers. 13 (4), 1-23 (2021).
- Ridky, T. W., Chow, J. M., Wong, D. J., Khavari, P. A. Invasive three-dimensional organotypic neoplasia from multiple normal human epithelia. Nature Medicine. 16 (12), 1450-1456 (2010).
- Casal, e. J., Crane, J. S. . Biochemistry, Glycosaminoglycans. , (2019).
- O’Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. J. The effect of pore size on cell adhesion in collagen-GAG scaffolds. Biomaterials. 26 (4), 433-441 (2005).
- O’Brien, F. J., Harley, B. A., Yannas, I. V., Gibson, L. Influence of freezing rate on pore structure in freeze-dried collagen-GAG scaffolds. Biomaterials. 25 (6), 1077-1086 (2004).
- Haugh, M. G., Murphy, C. M., McKiernan, R. C., Altenbuchner, C., O’Brien, F. J. Crosslinking and mechanical properties significantly influence cell attachment, proliferation, and migration within collagen glycosaminoglycan scaffolds. Tissue Engineering. Part A. 17 (9-10), 1201-1208 (2011).
- Murphy, C. M., Haugh, M. G., O’Brien, F. J. The effect of mean pore size on cell attachment, proliferation and migration in collagen-glycosaminoglycan scaffolds for bone tissue engineering. Biomaterials. 31 (3), 461-466 (2010).
- Haugh, M. G., Jaasma, M. J., O’Brien, F. J. The effect of dehydrothermal treatment on the mechanical and structural properties of collagen-GAG scaffolds. Journal of Biomedical Materials Research – Part A. 89 (2), 363-369 (2009).
- Lowe, B., Hardy, J. G., Walsh, L. J. Optimizing nanohydroxyapatite nanocomposites for bone tissue engineering. ACS Omega. 5 (1), 1-9 (2020).
- Cunniffe, G. M., Dickson, G. R., Partap, S., Stanton, K. T., O’Brien, F. J. Development and characterisation of a collagen nano-hydroxyapatite composite scaffold for bone tissue engineering. Journal of Materials Science. Materials in Medicine. 21 (8), 2293-2298 (2010).
- DuBois, S. G., et al. Metastatic sites in stage IV and IVS neuroblastoma correlate with age, tumor biology, and survival. Journal of Pediatric Hematology/Oncology. 21 (3), 181-189 (1999).
- Ryan, A. J., Gleeson, J. P., Matsiko, A., Thompson, E. M., O’Brien, F. J. Effect of different hydroxyapatite incorporation methods on the structural and biological properties of porous collagen scaffolds for bone repair. Journal of Anatomy. 227 (6), 732-745 (2015).
- Tierney, C. M., et al. The effects of collagen concentration and crosslink density on the biological, structural and mechanical properties of collagen-GAG scaffolds for bone tissue engineering. Journal of the Mechanical Behavior of Biomedical Materials. 2 (2), 202-209 (2009).
- Cox, R. F., Jenkinson, A., Pohl, K., O’Brien, F. J., Morgan, M. P. Osteomimicry of mammary adenocarcinoma cells in vitro; increased expression of bone matrix proteins and proliferation within a 3D collagen environment. PLoS One. 7 (7), 41679 (2012).
- Nolan, J. C., et al. Preclinical models for neuroblastoma: advances and challenges. Cancer Letters. 474, 53-62 (2020).
- Sirivisoot, S., Pareta, R., Harrison, B. S. Protocol and cell responses in threedimensional conductive collagen gel scaffolds with conductive polymer nanofibres for tissue regeneration. Interface Focus. 4 (1), 20130050 (2014).
- Thevenot, P., Nair, A., Dey, J., Yang, J., Tang, L. Method to analyze three-dimensional cell distribution and infiltration in degradable scaffolds. Tissue Engineering. Part C-Methods. 14 (4), 319-331 (2008).
- do Amaral, R. J. F. C., et al. Functionalising collagen-based scaffolds with platelet-rich plasma for enhanced skin wound healing potential. Frontiers in Bioengineering and Biotechnology. 7, 371 (2019).
- Gkolfinopoulos, S., Tsapakidis, K., Papadimitriou, K., Papamichael, D., Kountourakis, P. Chromogranin A as a valid marker in oncology: Clinical application or false hopes. World Journal of Methodology. 7 (1), 9-15 (2017).
- O’Brien, F. J., et al. The effect of pore size on permeability and cell attachment in collagen scaffolds for tissue engineering. Technology and Health Care. 15 (1), 3-17 (2007).

