Microscopie à feuille de lumière de la dynamique cardiaque rapide chez les embryons de poisson zèbre
Summary
Nous décrivons des outils optimisés pour étudier le cœur du poisson-zèbre in vivo avec la microscopie à fluorescence à feuille de lumière. Plus précisément, nous suggérons des lignées transgéniques cardiaques brillantes et présentons de nouvelles techniques d’intégration et d’immobilisation douces qui évitent les malformations développementales et cardiaques. Un éventuel pipeline d’acquisition et d’analyse de données adapté à l’imagerie cardiaque est également fourni.
Abstract
La recherche cardiaque embryonnaire a grandement bénéficié des progrès de la microscopie à fluorescence à feuille de lumière in vivo rapide (LSFM). Combiné au développement externe rapide, à la génétique traitable et à la translucidité du poisson-zèbre, Danio rerio, le LSFM a fourni des informations sur la forme et la fonction cardiaques à haute résolution spatiale et temporelle sans phototoxicité ou photoblanchiment significatif. L’imagerie des cœurs battants remet en question les techniques existantes de préparation des échantillons et de microscopie. Il faut maintenir un échantillon sain dans un champ de vision restreint et acquérir des images ultrarapides pour résoudre le rythme cardiaque. Nous décrivons ici des outils et des solutions optimisés pour étudier le cœur du poisson-zèbre in vivo. Nous démontrons les applications de lignées transgéniques brillantes pour l’étiquetage des constituants cardiaques et présentons de nouvelles techniques d’intégration et d’immobilisation douces qui évitent les défauts de développement et les changements de fréquence cardiaque. Nous proposons également un pipeline d’acquisition et d’analyse de données adapté à l’imagerie cardiaque. L’ensemble du flux de travail présenté ici se concentre sur l’imagerie cardiaque embryonnaire du poisson-zèbre, mais peut également être appliqué à divers autres échantillons et expériences.
Introduction
Pour découvrir les événements complexes et les interactions dans le cœur battant tôt, l’imagerie in vivo de l’ensemble de l’organe est nécessaire. Avec sa phototoxicité minimale1,2,3, son faible photoblanchiment4 et sa grande vitesse, la microscopie à feuille de lumière est devenue le principal outil d’imagerie pour le développement embryonnaire et cardiaque5,6. Il a fourni des informations sur la forme et la fonction cardiaques à une résolution spatiale et temporelle élevée7,8,9 et a permis aux chercheurs d’imager et de suivre à grande vitesse des parties du cœur marquées par fluorescence, d’étudier les forces hémodynamiques et de suivre le développement cardiaque directement à l’intérieur du corps des embryons en développement6,10,11,12.
Pour contraindre avec précision et de manière reproductible le poisson-zèbre dans le champ de vision, il existe une variété de protocoles d’intégration pour la feuille lumineuse, à court et à long terme, ainsi qu’un ou plusieurs échantillons13,14,15,16,17,18,19. Le protocole le plus courant implique l’immobilisation de la tricaïne et le montage de l’agarose à l’intérieur d’un tube en verre ou en plastique. Cependant, comme la fréquence cardiaque peut changer en raison de la température, des anesthésiques et du matériel d’intégration utilisé20,21,22, l’imagerie cardiaque du poisson-zèbre nécessite des protocoles spécifiques et doux pour assurer la santé de l’échantillon6,8,11,12,20,21,22,23 . Pour l’imagerie à court terme (jusqu’à une heure), on peut anesthésier le poisson dans 130 mg/L de tricaïne et l’incorporer dans des tubes en éthylène propylène fluoré (FEP) avec une solution d’agarose à 0,1% et un bouchon, comme décrit dans Weber et al. 201416. Cependant, comme la tricaïne peut entraîner des défauts de développement20,22, différents protocoles doivent être utilisés pour l’imagerie à long terme.
Nous décrivons ici une nouvelle stratégie pour l’imagerie cardiaque à long terme. Bien qu’il existe de nombreuses implémentations de feuilles lumineuses24, nous vous recommandons d’utiliser un échantillon suspendu dans un microscope T-SPIM (une lentille de détection et deux lentilles d’éclairage dans un plan horizontal avec l’échantillon suspendu verticalement dans la mise au point commune). Cela donne la liberté de mouvement et de rotation nécessaire pour le positionnement précis de l’échantillon. Les poissons sont immobilisés en injectant 30 pg α-bungarotoxine ARNm au stade à une ou deux cellules. α-bungarotoxine est un venin de serpent qui paralyse les muscles sans affecter le développement cardiovasculaire ou la physiologie22. Pour une imagerie précise et sans distorsion, nous recommandons de monter les poissons dans des tubes en FEP, un polymère avec un indice de réfraction presque identique à l’eau. Nous discutons de la meilleure façon de préparer les tubes FEP en les redressant et en les nettoyant avant l’imagerie. Les poissons sont ensuite montés dans ces tubes, tête en bas, dans un milieu, et le fond du tube est scellé avec un bouchon d’agarose à 2%, sur lequel reposent les têtes de poisson. La coupe de trous dans le tube FEP facilite les échanges gazeux et assure la croissance des poissons. Les poissons incorporés peuvent être conservés dans un support jusqu’à ce qu’ils soient montés sur un porte-échantillon juste avant l’imagerie. Nous suggérons également un pipeline d’acquisition et d’analyse de données pour une imagerie haute vitesse reproductible. En outre, nous discutons de l’utilisation de lignées transgéniques cytoplasmiques par rapport aux marqueurs membranaires pour le marquage robuste des cellules cardiaques, ainsi que de différentes options pour arrêter le cœur. Ces techniques de montage assurent la santé de l’échantillon tout en permettant de contraindre le cœur de manière précise et reproductible dans le champ de vision.
Protocol
Representative Results
Discussion
Lignes transgéniques pour imager le cœur
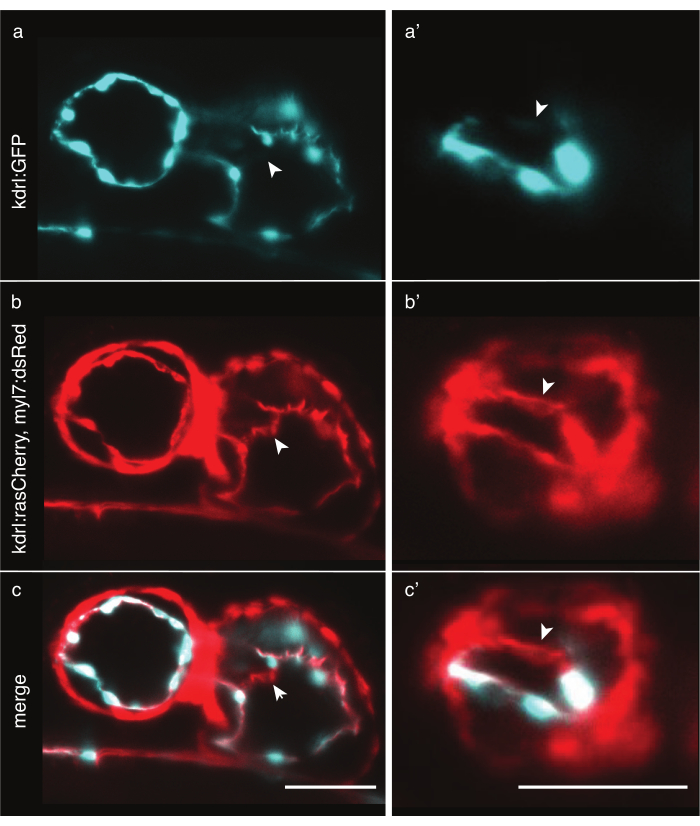
Figure 6 : Comparaison des lignées transgéniques de poisson-zèbre marqueur cytoplasmique et membranaire. Vue antérieure-ventrale de cœurs de poisson zèbre de 48 hpf imagés avec LSFM. Les flèches blanches indiquent des structures visibles uniquement avec une ligne transgénique marqueur membranaire. a) Signal Tg(kdrl:EGFP)32 en cyan dans le cœur et (a’) dans le ventricule. b) Signal Tg(kdrl:Hsa.HRAS-mCherry; myl7:dsRed)33 en rouge dans le cœur et (b’) dans le ventricule. ( c,c’) fusion des signaux Tg(kdrl:Hsa.HRAS-mCherry; myl7:dsRed) et Tg(kdrl:EGFP). Barre d’échelle 50 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
L’imagerie du cœur du poisson-zèbre nécessite un marquage précis des cellules cardiaques. Alors que l’épaisseur du myocarde est relativement constante dans toutes les cellules, les cellules endocardiques sont épaisses autour du noyau mais ont de minces protubérances membranaires, dans certaines régions plus minces que 2 μm. Les lignées transgéniques cytoplasmiques telles que Tg(kdrl:EGFP)32 marquent efficacement les régions autour des noyaux endocardiques, mais plus loin, le cytoplasme mince pourrait ne pas émettre suffisamment de photons pour être détecté avec des temps d’exposition aussi courts, ce qui entraînerait des trous artificiels dans les données (Figure 6a). En revanche, les lignées transgéniques marqueurs membranaires telles que Tg(kdrl:Hsa.HRAS-mCherry)33 peuvent étiqueter efficacement l’endocarde et révéler plus de détails (Figure 6b,c). Pour chaque expérience, choisissez soigneusement la lignée transgénique la plus appropriée.
Immobilisation du poisson-zèbre
Le choix de la technique d’immobilisation dépend de la durée de l’expérience et de l’âge du poisson à imager. La tricaïne a été couramment utilisée pour l’immobilisation du poisson-zèbre, principalement en raison de sa facilité d’utilisation. En effet, le simple fait d’ajouter 130 mg/L de tricaïne aux milieux piscicoles permet d’obtenir leur anesthésie en 10 min. Comme il peut entraîner des défauts de développement et affecter la physiologie cardiaque20,22, nous recommandons d’utiliser la tricaïne uniquement pour de courtes expériences (moins de 30 min). Pour une imagerie plus longue, les injections d’ARNm α-bungarotoxine au stade à une ou deux cellules paralysent les poissons jusqu’à 3 jours après la fécondation (dpf) sans affecter le développement cardiovasculaire ou la physiologie22.
Choisir les bons tubes FEP
Les tubes FEP sont disponibles en différents diamètres et épaisseurs. Pour imager 0-5 dpf poisson, 0,8 mm est un bon diamètre intérieur; choisissez soit des tubes à paroi épaisse de 0,8 x 1,6 mm, soit des tubes à paroi mince de 0,8 x 1,2 mm. Nous recommandons les tubes à paroi mince; cependant, les parois plus épaisses offrent une stabilité et une rigidité accrues, ce qui peut être important si la chambre d’échantillonnage a un fluide fluide qui pourrait perturber et déplacer un tube mince. Pour les échantillons plus grands, 1,6 x 2,4 mm et 2 x 3 mm peuvent être utilisés.
Échanges de température et de gaz
Un aspect essentiel du bien-être de l’embryon de poisson-zèbre est la température. Idéalement, maintenez le poisson à 28,5 °C pendant l’imagerie, car la température de l’environnement affecte le développement et la fréquence cardiaque34.
D’après notre expérience, l’échange d’oxygène à travers le bouchon d’agarose à 2% ne maintient qu’une fréquence cardiaque stable jusqu’à 3-4 dpf. Par conséquent, la coupe de trous dans le tube assure la diffusion de l’oxygène. Il peut également être nécessaire pour l’administration de médicaments à l’échantillon si vous le souhaitez.
Suspension du rythme cardiaque.
Les vitesses d’acquisition rapides des microscopes à feuille de lumière équipés de manière appropriée permettent d’enregistrer le cœur battant in vivo. Cependant, pour acquérir une pile z non perturbée, on peut ralentir ou arrêter le cœur. Cependant, l’arrêt du cœur entraîne une relaxation du muscle cardiaque et peut entraîner l’effondrement du cœur6. La suspension du rythme cardiaque peut être effectuée en utilisant des morpholinos, des basses températures, un inhibiteur de la contraction musculaire ou de l’optogénétique. Ces méthodes ont chacune leurs inconvénients et doivent être soigneusement évaluées pour chaque expérience.
L’injection de 4 ng de morpholino cardiaque silencieux (sih) au stade d’une cellule peut arrêter le rythme cardiaque en ciblant le gène tnnt2a crucial pour la formation de sarcomères35. Les poissons-zèbres sih n’ont pas de battement de cœur et ne survivent que jusqu’à 7 dpf, lorsque les embryons commencent à dépendre du sang circulant pour l’oxygénation. Comme la morphogenèse cardiaque est entraînée par des forces génétiques et biomécaniques36, ces poissons présentent des malformations cardiaques autour de 3 dpf.
Comme le flux de Ca2+ est sensible à la température, la température influence la fréquence cardiaque chez les poissons-zèbres embryonnaires21. Par conséquent, l’abaissement de la température dans la chambre d’imagerie ralentit le rythme cardiaque. L’arrêt du rythme cardiaque nécessite des températures inférieures à 15 °C. Comme les poissons-zèbres sont généralement maintenus à 28,5 ° C, ces températures basses ne peuvent être maintenues que pendant de brèves périodes (moins de 10 min).
Des médicaments tels que les inhibiteurs chimiques des contractions musculaires, le 2,3-Bu-tanedione 2-monoxime (BDM), peuvent être ajoutés au milieu du poisson-zèbre (50 nM37,38) pour suspendre temporairement le rythme cardiaque. BDM est pratique à utiliser car il arrête la contraction cardiaque en moins de 15 minutes et peut être lavé pour restaurer la fonction cardiaque. Cependant, comme le BDM modifie le potentiel d’action cardiaque, il doit être utilisé avec prudence37.
Enfin, le cœur des poissons-zèbres transgéniques exprimant des canaux ioniques à faible luminosité ou des pompes telles que la channelrhodopsine ou l’halorhodopsine dans leur myocarde peut être manipulé et arrêté en éclairant le stimulateur cardiaque au niveau du tractus d’entrée avec de la lumière39,7,40,41,9.
Perspective
Les outils et solutions optimisés présentés pour étudier le cœur du poisson-zèbre in vivo permettent une imagerie douce et à long terme de la dynamique cardiaque ultrarapide. L’intégration de l’échantillon peut être adaptée à différentes modalités d’imagerie, telles que la microscopie confocale, la microscopie à deux photons ou la tomographie par projection optique (OPT). La microscopie à feuille de lumière, cependant, est probablement la technique préférée qui offre une section optique à une vitesse suffisante pour capturer la dynamique du cœur. Bien que ce protocole se concentre sur l’imagerie cardiaque embryonnaire du poisson-zèbre, nous pensons qu’il pourrait également être appliqué à divers autres échantillons et expériences. Il sera intéressant de voir à l’avenir si des techniques d’intégration et d’imagerie similaires peuvent également être utilisées à des stades ultérieurs du développement, lorsque le cœur est plus caché et la larve moins translucide.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Nous remercions Madelyn Neufeld pour l’illustration de la figure 2h. Ce travail a été soutenu par la Max Planck Society, le Morgridge Institute for Research, la Chan Zuckerberg Initiative et le Human Frontier Science Program (HFSP).
Materials
| 1.5mL Eppendorf | Eppendorf | 22364111 | To carry embedded samples |
| 15mL Falcon tubes | Falcon | 352095 | To carry embedded samples |
| 50mL centrifuge tubes | Falcon | 352070 | 50ml tubes for sonication step, and storing cleaned, straightened FEP tubes |
| 50mL syringe | BD | 309654 | 50ml syringe for FEP cleaning |
| Agarose, low gelling temperature | Sigma Aldrich | 39346-81-1 | To make plug |
| Blunt Tip Needles, 21 gauge | VWR | 89500-304 | Blunt end needle for 0.8 inner diameter FEP tube |
| Borosilicate glass tube | McMaster-Carr | 8729K33 | Tubing for FEP tube straightening 9.5mm outer diameter, 5.6 inner diameter, 30cm long, other sizes available |
| Borosilicate glass tube | McMaster-Carr | 8729K31 | Tubing for FEP tube straightening 6.35mm outer diameter, 4mm inner diameter, 30cm long, other sizes available |
| Conventional needles, 21 Gauge | BD | 305165 | Conventional needle for 0.8 inner diameter FEP tube |
| Disposable glass pipette | Grainger | 52NK56 | To transfer fish, use with pipette pump |
| E3 medium for zebrafish embryos | |||
| Ethanol | Sigma Aldrich | 64-17-5 | Ethanol for FEP cleaning |
| FEP tube, 0.8 / 1.2 mm | ProLiquid | 2001048 | FEP tube with thick wall, other sizes available |
| FEP tube, 0.8 / 1.6 mm | Bola | S1815-04 | FEP tube with thin wall, other sizes available |
| FEP tube, 2/3mm | BGB | 211581 | Large FEP tube with thick wall, other sizes available |
| Hot Hand Rubber Mitt | Cole-Parmer | 691000 | To carry hot equipment after autoclaving |
| Omnifix 1mL Syringes | B Braun | 9161406V | 1ml syringe for embedding |
| Petri dish, small | Dot Scientific | PD-94050 | To make agarose plug |
| Pipette pumps | Argos Technologies | 04395-05 | To transfer fish, use with disposable glass pipette |
| PTU | Sigma Aldrich | 103-85-5 | Also known as: N-Phenylthiourea, 1-Phenyl-2-thiourea, Phenylthiocarbamide |
| Razor blades | Azpack | 11904325 | To cut FEP tubes |
| Sodium Hydroxide | Dot Scientific | DSS24000 | NaOH for FEP cleaning |
| Tricaine | Sigma Aldrich | E10521 | Also known as: MS-222, Ethyl 3-aminobenzoate methanesulfonate, Tricaine |
References
- Hoebe, R. A., et al. Controlled light-exposure microscopy reduces photobleaching and phototoxicity in fluorescence live-cell imaging. Nature Biotechnology. 25 (2), 249-253 (2007).
- Jenielita, M., Taormina, M. J., Delaurier, A., Kimmel, C. B., Parthasarathy, R. Comparing phototoxicity during the development of a zebrafish craniofacial bone using confocal and light sheet fluorescence microscopy techniques. Journal of Biophotonics. 6 (11-12), 920-928 (2013).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. BioEssays. 39 (8), 1700003 (2017).
- Reynaud, E. G., Kržič, U., Greger, K., Stelzer, E. H. K. Light sheet-based fluorescence microscopy: more dimensions, more photons, and less photodamage. Human Frontier Science Program Journal. 2 (5), 266-275 (2008).
- Weber, M., Huisken, J. Light sheet microscopy for real-time developmental biology. Current Opinion in Genetics & Development. 21 (5), 566-572 (2011).
- Mickoleit, M., et al. High-resolution reconstruction of the beating zebrafish heart. Nat Methods. 11 (9), 919-922 (2014).
- Arrenberg, A. B., Stainier, D. Y., Baier, H., Huisken, J. Optogenetic control of cardiac function. Science. 330 (6006), 971-974 (2010).
- Trivedi, V., et al. Dynamic structure and protein expression of the live embryonic heart captured by 2-photon light sheet microscopy and retrospective registration. Biomed Optics Express. 6 (6), 2056-2066 (2015).
- Weber, M., Yelon, D., et al. Cell-accurate optical mapping across the entire developing heart. eLife. 6, 28307 (2017).
- Weber, M., Huisken, J. In vivo imaging of cardiac development and function in zebrafish using light sheet microscopy. Swiss Medical Weekly. 145, 14227 (2015).
- Fei, P., et al. Cardiac light-sheet fluorescent microscopy for multi-scale and rapid imaging of architecture and function. Science Reports. 6 (1), 1-12 (2016).
- Felker, A., et al. Continuous addition of progenitors forms the cardiac ventricle in zebrafish. Nature Communication. 9 (1), 1-14 (2018).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. K. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305 (5686), 1007-1009 (2004).
- Keller, P. J., Pampaloni, F., Stelzer, E. H. Life sciences require the third dimension. Current Opinion in Cell Biology. 18 (1), 117-124 (2006).
- Keller, P. J., Schmidt, A. D., Wittbrodt, J., Stelzer, E. H. K. Reconstruction of zebrafish early embryonic development by scanned light sheet microscopy. Science. 322 (5904), 1065-1069 (2008).
- Weber, M., Mickoleit, M., Huisken, J. Multilayer mounting for long-term light sheet microscopy of zebrafish. Journal of Visualized Experiments. (84), e51119 (2014).
- Berndt, F., Shah, G., Power, R. M., Brugués, J., Huisken, J. Dynamic and non-contact 3D sample rotation for microscopy. Nature Communications. 9 (1), 1-7 (2018).
- Daetwyler, S., Günther, U., Modes, C. D., Harrington, K., Huisken, J. Multi-sample SPIM image acquisition, processing. and analysis of vascular growth in zebrafish. Development. , 173757 (2019).
- Keomanee-Dizon, K., Fraser, S. E., Truong, T. V. A versatile, multi-laser twin-microscope system for light-sheet imaging. Review of Scientific Instruments. 91 (5), 053703 (2020).
- Kaufmann, A., Mickoleit, M., Weber, M., Huisken, J. Multilayer mounting enables long-term imaging of zebrafish development in a light sheet microscope. Development. 139 (17), 3242-3247 (2012).
- Gierten, J., et al. Automated high-throughput heartbeat quantification in medaka and zebrafish embryos under physiological conditions. Science Reports. 10 (1), 1-12 (2020).
- Swinburne, I. A., Mosaliganti, K. R., Green, A. A., Megason, S. G. Improved long-term imaging of embryos with genetically encoded α-bungarotoxin. PLOS ONE. 10 (8), 0134005-0134015 (2015).
- Taylor, J. M., et al. Adaptive prospective optical gating enables day-long 3D time-lapse imaging of the beating embryonic zebrafish heart. Nature Communication. 10 (1), 1-15 (2019).
- Power, R. M., Huisken, J. A guide to light-sheet fluorescence microscopy for multiscale imaging. Nature Methods. 14 (4), 360-373 (2017).
- Nüsslein-Volhard, C., Dahm, R. . Zebrafish: A Practical Approach. , (2002).
- Avdesh, A., et al. Regular care and maintenance of a zebrafish (Danio rerio) laboratory: An introduction. Journal of Visualized Experiments. (69), e4196 (2012).
- Cold Spring Harbor. E3 medium (for zebrafish embryos). Cold Spring Harbor Protocols. 2011, 66449 (2011).
- Yuan, S., Sun, Z. Microinjection of mRNA and morpholino antisense oligonucleotides in zebrafish embryos. Journal of Visualized Experiments. (27), e1113 (2009).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Rueden, C. T., et al. ImageJ2: ImageJ for the next generation of scientific image data. BMC Bioinformatics. 18 (1), 529 (2017).
- Jin, S. -. W., Beis, D., Mitchell, T., Chen, J. -. N., Stainier, D. Y. Cellular and molecular analyses of vascular tube and lumen formation in zebrafish. Development. 132 (23), 5199-5209 (2005).
- Chi, N. C., et al. Foxn4 directly regulates tbx2b expression and atrioventricular canal formation. Genes and Development. 22 (6), 734-739 (2008).
- Scott, G. R., Johnston, I. A. Temperature during embryonic development has persistent effects on thermal acclimation capacity in zebrafish. Proceedings of the National Academy of Sciences. 109 (35), 14247-14252 (2012).
- Sehnert, A. J., et al. Cardiac troponin T is essential in sarcomere assembly and cardiac contractility. Nature Genetics. 31 (1), 106-110 (2002).
- Sidhwani, P., Yelon, D., Wellik, D. M. Fluid forces shape the embryonic heart: Insights from zebrafish. Current Topics in Developmental Biology. 132, 395-416 (2019).
- Jou, C. J., Spitzer, K. W., Tristani-Firouzi, M. Blebbistatin effectively uncouples the excitation-contraction process in zebrafish embryonic heart. Cell Physiology and Biochemistry. 25 (4-5), 419-424 (2010).
- Chow, R. W. -. Y., Lamperti, P., Steed, E., Boselli, F., Vermot, J. following endocardial tissue movements via cell photoconversion in the zebrafish embryo. Journal of Visualized Experiments. (132), e57290 (2018).
- Nagel, G., et al. Channelrhodopsin-2, a directly light-gated cation-selective membrane channel. Proccedings of the National Academy of Sciences. 100 (24), 13940-13945 (2003).
- Knollmann, B. C. Pacing lightly: optogenetics gets to the heart. Nature Methods. 7 (11), 889-891 (2010).
- Bruegmann, T., et al. Optogenetic control of heart muscle in vitro and in vivo. Nature Methods. 7 (11), 897-900 (2010).

