S. cerevisiae 세포로부터 정제된 양방향 Kinesin-5 Cin8의 단일 분자 및 클러스터의 이동성
Summary
양방향 유사분열 키네신-5 Cin8은 운동성 동안 분열되고 병합되는 클러스터에 축적됩니다. 클러스터에 축적되면 Cin8의 속도와 방향성도 변경됩니다. 여기에서, 정제된 Cin8-GFP를 사용한 운동성 분석 및 Cin8의 단일 분자 및 클러스터의 모타일 특성의 분석을 위한 프로토콜이 기재되어 있다.
Abstract
유사분열성 양극성 키네신-5 모터는 스핀들 역학에서 필수적인 기능을 수행합니다. 이 모터는 활성 복합체의 반대쪽 끝에 위치한 두 쌍의 촉매 모터 도메인을 가진 호모 사량체 구조를 나타냅니다. 이 독특한 아키텍처는 kinesin-5 모터가 반평행 스핀들 미세소관(MT)을 가교 및 분리할 수 있게 해줌으로써 스핀들 극을 분리하는 외향 지향적인 힘을 제공합니다. 이전에는 kinesin-5 모터가 독점적으로 플러스 엔드 지향으로 여겨졌습니다. 그러나 최근 연구에 따르면 여러 곰팡이 키네신 -5 모터는 단일 분자 수준에서 향하는 마이너스 엔드이며 다양한 실험 조건에서 방향성을 전환 할 수 있습니다. Saccharomyces cerevisiae kinesin-5 Cin8은 이러한 양방향 운동 단백질의 예입니다 : 높은 이온 강도 조건에서 Cin8의 단일 분자가 MT의 마이너스 – 끝 방향으로 움직입니다. 또한 Cin8은 주로 MT의 마이너스 말단에 모타일 클러스터를 형성하며, 이러한 클러스터링을 통해 Cin8은 방향성을 전환하고 느리고 플러스 엔드 지향 운동성을 겪을 수 있습니다. 이 기사는 S. cerevisiae 세포에서의 단백질 과발현 및 그의 정제에서부터 시험관내 단일 분자 운동성 분석에 이르기까지 GFP 태깅된 키네신-5 Cin8로 작업하는 모든 단계에 대한 상세한 프로토콜을 제공한다. 여기에 설명 된 새로 개발 된 방법은 형광 강도에 따라 Cin8의 단일 분자와 클러스터를 구별하는 데 도움이됩니다. 이 방법은 Cin8의 단일 분자 및 클러스터의 운동성에 대한 별도의 분석을 가능하게하여 클러스터 크기에 대한 Cin8 운동성의 의존성의 특성을 제공합니다.
Introduction
진핵 세포 내의 많은 운동성 사건은 분자 운동 단백질의 기능에 의해 매개된다. 이 모터는 세포 골격 필라멘트, 액틴 필라멘트 및 미세 소관 (MTs)을 따라 움직이며 ATP 가수분해의 화학 에너지를 세포 내에서 생물학적 운동성을 유도하는 데 필요한 운동 및 기계적 힘으로 변환합니다. MT 기반 S. cerevisiae Cin8은 양극성, 호모테트라머 키네신-5 모터 단백질로, 스핀들 MT를 교차 연결하고슬라이드합니다 1. Cin8은 유사분열 동안, 스핀들 어셈블리 2,3,4 및 아나페이즈 5,6,7 동안 스핀들 신장에서 필수 기능을 수행합니다. 이전에는 Cin8이 다양한 실험 조건에서 방향성을 전환하는 양방향 모터라는 것이 입증되었습니다. 예를 들어, 높은 이온 강도 조건에서 단일 Cin8 모터는 MT의 마이너스 끝쪽으로 이동하지만 클러스터, 다중 모터 MT 글라이딩 분석 및 병렬 MT 사이에서 Cin8 모터는 주로 MT 8,9,10,11,12의 플러스 엔드쪽으로 이동합니다. . 이러한 발견은 여러 가지 이유로 인해 매우 예상치 못한 결과였습니다. 첫째, Cin8은 아미노 말단에서 촉매 모터 도메인을 운반하며 그러한 모터는 이전에 독점적으로 플러스 엔드 지향으로 여겨졌지만 Cin8은 단일 분자 수준에서 마이너스 말단으로 향하는 것으로 나타났습니다. 둘째, 키네신 모터는 마이너스 엔드 또는 플러스 엔드 지향 단방향인 단방향인 것으로 여겨졌지만, Cin8은 실험 조건에 따라 양방향인 것으로 나타났다. 마지막으로, 유사 분열 스핀들에서의 MT 방향 때문에, 스핀들 조립 및 아나 페이즈 B 동안 스핀들 폴의 분리에서 kinesin-5 모터의 고전적인 역할은1,13을 교차 연결하는 MT에 대한 플러스 엔드 지향 운동성으로 만 설명 될 수 있습니다. Cin8의 양방향성에 대한 첫 번째 보고에 따라, 몇 가지 다른 키네신 모터가 양방향 14,15,16인 것으로 입증되었으며, 이는 키네신 모터의 양방향 운동성이 이전에 생각했던 것보다 더 일반적일 수 있음을 나타냅니다.
세포에서 Cin8은 또한 양방향 방식으로 움직인다8, 일부 kinesin-5 모터의 양방향 운동성이 세포 내 기능에 중요하다는 개념을 뒷받침하는 것으로 이전에 보고되었다. 또한, 양방향성으로 보고되었던 세 개의 키네신-5 모터가 진균 세포로부터 왔기 때문에, 키네신-5 모터의 양방향성에 대한 가능한 역할이 최근 이러한 세포(10)에서 제안되고 있다. 이 모델에 따르면, 유사분열 중에 핵 외피가 분해되지 않는 곰팡이 세포의 폐쇄 된 유사분열에서 kinesin-5 모터는 스핀들 조립 전에 스핀들 극을 분리하는 초기 힘을 제공합니다. 이 작업을 수행하기 위해 스핀들 폴 분리 전에 kinesin-5 모터는 단일 핵 MT에서 마이너스 엔드 지향 운동성에 의해 스핀들 폴 근처에 국한됩니다. 이 위치에 도착하면 kinesin-5 모터가 클러스터링되고, 방향성을 전환하고, 캡처하고, 인접 스핀들 폴에서 MT를 교차 연결합니다. 그 후, kinesin-5 모터는 가교 연결된 MT에서 플러스 엔드 지향 운동성으로 극의 초기 분리를 제공합니다. 이 모델에 따르면, 단일 MT의 마이너스 엔드 지향 운동성과 반평행 슬라이딩 중 가교 MT의 플러스 엔드 지향 운동성은 곰팡이 키네신-5 모터가 스핀들 어셈블리1,13에서 역할을 수행하는 데 필요합니다.
기술된 방법의 전반적인 목표는 고순도 진균성 GFP-태깅된 키네신-5 Cin8을 수득하고 Cin8의 단일 분자 및 클러스터의 운동성을 개별적으로 분석하면서 단일 분자 운동성 분석(도 1)을 수행하는 것이다. 단일 분자와 클러스터 사이의 분리는 Cin8의 방향성에 영향을 미치는 것으로 입증된 인자 중 하나가 MT10,12 상의 클러스터에 축적되기 때문에 중요하다. MT 표면 글라이딩 및 MT 슬라이딩 분석과 같은 대안적인 운동성 분석은 단일 운동 단백질17,18의 활성에 관한 정보를 제공하지 않는다. 여기에 설명된 강력한 단일 분자 운동성 분석 및 분석 방법은 키네신-5 모터, Cin8 및 Kip1 10,11,12,14,19,20의 다양한 측면을 특성화하기 위해 성공적으로 적용되었습니다.
여기에서, Cin8 과발현 및 정제, MTs의 중합, 및 단일 분자 운동성 검정을 위한 상세한 프로토콜이 제시된다. 또한, Cin8의 단일 분자와 클러스터를 구별하고, 평균 변위 (MD) 및 평균 제곱 변위 (MSD) 분석에 의해 단일 모터 및 클러스터 속도를 결정하는 분석도 설명된다. 이 프로토콜은 연구자가 절차의 모든 단계를 시각화하고 이러한 유형의 분석 문제를 해결하는 데 도움을주는 것을 목표로합니다.
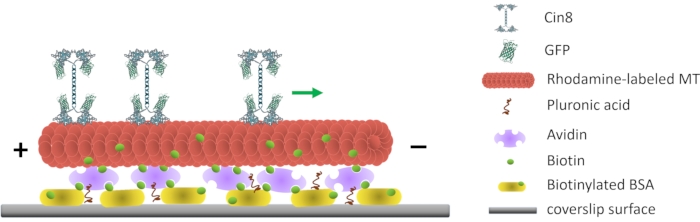
도 1: 단일 분자 운동성 분석의 개략적 표현. 비오티닐화 형광 MTs는 유리 표면에 부착되고, 표면 부착된 비오티닐화-BSA와 상호작용하는 아비딘으로 코팅된다. 녹색 화살표는 높은 이온 강도 조건 하에서 단일 Cin8 분자의 이동 방향을 나타낸다. +/- MT의 극성을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Protocol
Representative Results
Discussion
이 연구에서는 양방향 키네신-5 Cin8을 사용한 단일 분자 운동성 분석 및 운동성 분석을 위한 프로토콜이 제시된다. C 말단에서 천연 핵 국재화 신호(NLS)를 포함하는 전장Cin8(18 )은 천연 숙주 S. 세레비시아로부터 정제되었다. Cin8은 핵 운동 단백질이기 때문에 액체 질소 하에서 S. cerevisiae 세포를 분쇄하는 것이 세포 용해를위한 가장 효율적인 방법임이 밝혀졌습니다. ?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
이 연구는 L.G.에 수여 된 이스라엘 과학 재단 보조금 (ISF-386/18)과 이스라엘 이중 국가 과학 재단 보조금 (BSF-2019008)에 의해 부분적으로 지원되었습니다.
Materials
| Adenine | FORMEDIUM | DOC0230 | |
| ATP | Sigma | A7699 | |
| Biotinylated-BSA | Sigma | A8549 | |
| Casein | Sigma | C7078 | |
| Catalase (C40) | Sigma | C40 | |
| Creatine-Kinase | Sigma | C3755 | |
| Dithiothreitol (DTT) | Sigma | D0632 | |
| EDTA | Sigma | E5134 | |
| EGTA | Sigma | E4378 | |
| Fluorescence filter set for GFP | Chroma | 49002: ET-EGFP (FITC/Cy2) | |
| Fluorescence filter set for Rhodamine | Chroma | 49004: ET-CY3/TRITC | |
| Fluorescence inverted microscope | Zeiss | Axiovert 200M | |
| Galactose | Tivan Biotech | GAL02 | |
| Glucose | Sigma | G8270 | |
| Glucose Oxidase | Sigma | G7141 | |
| Glycerol | Sigma | G5516 | |
| GlycylGlycine | Merck | G0674 | |
| GMPCPP | Jana Bioscience | Nu-405L | |
| GTB | Cytoskeleton | BST01-010 | |
| GTP | Sigma | G8877 | |
| Histidine | Duchefa Biochemie | H0710.0100 | |
| ImageJ-FIJI software | https://imagej.net/plugins/trackmate/ | version 2.1.0/1.53c; Java 1.8.0_172 [64-bit] for Windows 10 | |
| Imidazole | Sigma | I0125 | |
| InstantBlue Coomassie Protein Stain | Abcam | ab119211 | |
| Lens | Zeiss | 100x/1.4 oil DIC objective | |
| Lysine | FORMEDIUM | DOC0161 | |
| Magnesium Chloride | Sigma | M8266 | |
| Methionine | Duchefa Biochemie | M0715.0100 | |
| Neo | Andor Technologies | sCMOS camera | |
| NeutraAvidin | Life | A2666 | |
| Ni-NTA Agarose | Invitrogen | R901-15 | |
| Phospho-Creatine | Sigma | P1937 | |
| Pipes | Sigma | P1851 | |
| Pluronic acid F-127 (poloxamer) | Sigma | P2443 | |
| Potassium Chloride | Sigma | P9541 | |
| Raffinose | Tivan Biotech | RAF01 | |
| Size Exclusion chromatography instument | GE Healthcare | AKTA Pure | |
| Spectrophotometer | ThermoFisher Scientific | NanoDrop | |
| Superose-6 10/300 GL | GE Healthcare | 17-5172-01 | |
| Tris | Roshe | 10708976001 | |
| Triton X-100 | Sigma | T8787 | |
| Tryptophan | Duchefa Biochemie | T0720.0100 | |
| Tubulin protein | Cytoskeleton | T240 | |
| Tubulin, biotinylated | Cytoskeleton | T333P | |
| Tubulin, TRITC Rhodamine | Cytoskeleton | TL530M | |
| Uracil | Sigma | U0750-100G | |
| Yeast nitrogen base | FORMEDIUM | CYN0401S | |
| α-GFP antibody | Santa Cruz Biotechnology | SC8036 | |
| β-mercaptoethanol | Sigma | M3148 |
References
- Singh, S. K., Pandey, H., Al-Bassam, J., Gheber, L. Bidirectional motility of kinesin-5 motor proteins: structural determinants, cumulative functions and physiological roles. Cellular and Molecular Life Sciences. 75 (10), 1757-1771 (2018).
- Hoyt, M. A., He, L., Totis, L., Saunders, W. S. Loss of function of Saccharomyces cerevisiae kinesin-related CIN8 and KIP1 is suppressed by KAR3 motor domain mutations. Genetics. 135 (1), 35-44 (1993).
- Saunders, W. S., Hoyt, M. A. Kinesin-related proteins required for structural integrity of the mitotic spindle. Cell. 70 (3), 451-458 (1992).
- Hoyt, M. A., He, L., Loo, K. K., Saunders, W. S. Two Saccharomyces cerevisiae kinesin-related gene products required for mitotic spindle assembly. Journal of Cell Biology. 118 (1), 109-120 (1992).
- Gerson-Gurwitz, A., et al. Mid-anaphase arrest in S. cerevisiae cells eliminated for the function of Cin8 and dynein. Cellular and Molecular Life Sciences. 66 (2), 301-313 (2009).
- Fridman, V., Gerson-Gurwitz, A., Movshovich, N., Kupiec, M., Gheber, L. Midzone organization restricts interpolar microtubule plus-end dynamics during spindle elongation. EMBO Reports. 10 (4), 387-393 (2009).
- Movshovich, N., et al. Slk19-dependent mid-anaphase pause in kinesin-5-mutated cells. Journal of Cell Science. 121 (15), 2529-2539 (2008).
- Gerson-Gurwitz, A., et al. Directionality of individual kinesin-5 Cin8 motors is modulated by loop 8, ionic strength and microtubule geometry. Embo Journal. 30 (24), 4942-4954 (2011).
- Roostalu, J., et al. Directional switching of the kinesin Cin8 through motor coupling. Science. 332 (6025), 94-99 (2011).
- Shapira, O., Goldstein, A., Al-Bassam, J., Gheber, L. A potential physiological role for bi-directional motility and motor clustering of mitotic kinesin-5 Cin8 in yeast mitosis. Journal of Cell Science. 130 (4), 725-734 (2017).
- Goldstein-Levitin, A., Pandey, H., Allhuzaeel, K., Kass, I., Gheber, L. Intracellular functions and motile properties of bi-directional kinesin-5 Cin8 are regulated by neck linker docking. eLife. 10, 71036 (2021).
- Pandey, H., et al. Drag-induced directionality switching of kinesin-5 Cin8 revealed by cluster-motility analysis. Science Advances. 7 (6), 1687 (2021).
- Pandey, H., Popov, M., Goldstein-Levitin, A., Gheber, L. Mechanisms by which kinesin-5 motors perform their multiple intracellular functions. International Journal of Molecular Sciences. 22 (12), 6420 (2021).
- Fridman, V., et al. Kinesin-5 Kip1 is a bi-directional motor that stabilizes microtubules and tracks their plus-ends in vivo. Journal of Cell Science. 126, 4147-4159 (2013).
- Edamatsu, M. Bidirectional motility of the fission yeast kinesin-5, Cut7. Biochemical and Biophysical Research Communications. 446 (1), 231-234 (2014).
- Popchock, A. R., et al. The mitotic kinesin-14 KlpA contains a context-dependent directionality switch. Nature Communications. 8, 13999 (2017).
- Bodrug, T., et al. The kinesin-5 tail domain directly modulates the mechanochemical cycle of the motor domain for anti-parallel microtubule sliding. eLife. 9 (9), 51131 (2020).
- Gheber, L., Kuo, S. C., Hoyt, M. A. Motile properties of the kinesin-related Cin8p spindle motor extracted from Saccharomyces cerevisiae cells. Journal of Biological Chemistry. 274 (14), 9564-9572 (1999).
- Pandey, H., et al. Flexible microtubule anchoring modulates the bi-directional motility of the kinesin-5 Cin8. Cellular and Molecular Life Sciences. 78 (16), 6051-6068 (2021).
- Shapira, O., Gheber, L. Motile properties of the bi-directional kinesin-5 Cin8 are affected by phosphorylation in its motor domain. Scientific Reports. 6, 25597 (2016).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Britto, M., et al. Schizosaccharomyces pombe kinesin-5 switches direction using a steric blocking mechanism. Proceedings of the National Academy of Sciences of the United States of America. 113 (47), 7483-7489 (2016).
- Kapitein, L. C., et al. Microtubule cross-linking triggers the directional motility of kinesin-5. Journal of Cell Biology. 182 (3), 421-428 (2008).
- Furuta, K., Edamatsu, M., Maeda, Y., Toyoshima, Y. Y. Diffusion and directed movement in vitro motile properties of fission yeast kinesin-14 Pkl1. Journal of Biological Chemistry. 283 (52), 36465-36473 (2008).
- Katrukha, E. A., et al. Probing cytoskeletal modulation of passive and active intracellular dynamics using nanobody-functionalized quantum dots. Nature Communications. 8, 14772 (2017).
- Tinevez, J. Y., et al. TrackMate: An open and extensible platform for single-particle tracking. Methods. 115, 80-90 (2017).
- Jakobs, M. A. H., Dimitracopoulos, A., Franze, K. KymoBulter, a deep learning software for automated kymograph analysis. eLife. 8, 42288 (2019).

