Avaliação Simultânea de Parentesco, Número de Divisão e Fenótipo por Citometria de Fluxo para Células-Tronco Hematopoéticas e Progenitoras
Summary
Apresentamos aqui uma técnica baseada em citometria de fluxo que permite medir simultaneamente o número de divisões celulares, o fenótipo celular de superfície e o parentesco celular. Essas propriedades podem ser testadas estatisticamente usando uma estrutura baseada em permutação.
Abstract
Poucas técnicas podem avaliar fenótipo e destino para uma mesma célula simultaneamente. A maioria dos protocolos atuais utilizados para caracterizar o fenótipo, embora capaz de gerar grandes conjuntos de dados, necessita da destruição da célula de interesse, impossibilitando a avaliação de seu destino funcional. Sistemas diferenciadores biológicos heterogêneos, como a hematopoiese, são, portanto, difíceis de descrever. Com base em corantes de rastreamento de divisão celular, desenvolvemos um protocolo para determinar simultaneamente o parentesco, o número de divisão e o status de diferenciação para muitos progenitores hematopoiéticos únicos. Este protocolo permite avaliar o potencial de diferenciação ex vivo de progenitores hematopoéticos murinos e humanos, isolados de diversas fontes biológicas. Além disso, por ser baseado em citometria de fluxo e um número limitado de reagentes, pode gerar rapidamente uma grande quantidade de dados, em nível unicelular, de maneira relativamente barata. Também fornecemos o pipeline analítico para análise de célula única, combinado com uma estrutura estatística robusta. Como esse protocolo permite a ligação da divisão e diferenciação celular no nível de célula única, ele pode ser usado para avaliar quantitativamente o comprometimento do destino simétrico e assimétrico, o equilíbrio entre auto-renovação e diferenciação e o número de divisões para um determinado destino de compromisso. Em conjunto, este protocolo pode ser utilizado em delineamentos experimentais com o objetivo de desvendar as diferenças biológicas entre progenitores hematopoéticos, a partir de uma perspectiva unicelular.
Introduction
A última década foi marcada pela disseminação mundial de abordagens unicelulares para biologia celular e molecular. Seguindo os passos da genômica de célula única1,2, hoje em dia é possível estudar muitos componentes de uma única célula (por exemplo, DNA, RNA, proteínas), com novas técnicas de célula única -ômicas surgindo a cada ano. Essas técnicas têm lançado luz sobre velhas e novas questões para os campos da imunologia, neurobiologia, oncologia e outros, tanto utilizando células humanas quanto de organismos modelo3. Ao destacar as diferenças entre as células individuais, a ômica unicelular motivou a definição de um novo modelo de hematopoese, centrado na heterogeneidade das células-tronco hematopoéticas e progenitoras (HSPCs) e afastando-se do modelo clássico de populações discretas homogêneas 4,5.
Uma das poucas desvantagens de todas as técnicas -ômicas é a destruição da célula de interesse, impossibilitando a possibilidade de avaliar sua funcionalidade. Por outro lado, outros métodos de célula única, como o ensaio de transplante de célula única e tecnologias de rastreamento de linhagem, fornecem uma leitura da funcionalidade da célula ancestral avaliando o destino de células individuais in vivo 6,7. As tecnologias de rastreamento de linhagens envolvem a marcação da célula de interesse com uma etiqueta genética hereditária7 ou fluorescente8,9, permitindo que o destino de várias células individuais seja seguido ao mesmo tempo. No entanto, a caracterização das células iniciais é tipicamente limitada a um número restrito de parâmetros, como a expressão de algumas proteínas de superfície avaliada por citometria de fluxo10. Além disso, as tecnologias de rastreamento de linhagem de célula única requerem detecção trabalhosa da marca celular, tipicamente via sequenciamento de DNA/RNA ou imagem. Este último ponto, em particular, limita o número de condições que podem ser testadas em um único experimento.
Outra classe de métodos que são usados para estudar a funcionalidade de células únicas são sistemas de cultura de células ex vivo de HSPCs individuais. Fáceis de executar, esses ensaios padrão-ouro envolvem a classificação de células individuais em vasos de cultura celular de 96 poços e, após a cultura, caracterizando o fenótipo de progênie celular, tipicamente por citometria de fluxo ou análise morfológica. Esses ensaios têm sido usados principalmente para caracterizar a diferenciação a longo prazo de HSPCs em células maduras, tipicamente após 2-3 semanas de cultura11,12. Alternativamente, têm sido utilizados para tentar manter e expandir os HSPCs ex vivo 13,14,15,16,17,18, com a promessa de benefício médico para o transplante de células-tronco humanas 19. Por fim, têm sido utilizados para estudar o comprometimento precoce de HSPCs utilizando cultura de curta duração20, sendo o baixo número de células geradas nessa cultura o principal fator limitante. Uma desvantagem desses diferentes tipos de ensaios ex vivo é que eles refletem apenas parcialmente a complexidade in vivo; ainda assim, eles são uma das raras maneiras de estudar a diferenciação humana HSPC.
Uma informação ausente dos métodos unicelulares existentes (célula única-ômica, rastreamento de linhagem e cultura ex vivo) é a detecção precisa de divisões celulares, um parâmetro essencial a ser considerado ao estudar a dinâmica do HSPC21. Uma maneira simples de avaliar o número de divisões por citometria de fluxo é o uso de “corantes proteicos” solúveis, como o éster succinimidílico de diacetato de 5-(e 6)-carboxifluoresceína (CFSE)22. Esses corantes de divisão difundem-se dentro do citoplasma das células coradas, diluem-se pela metade e passam para as duas células-filhas a cada divisão celular, permitindo enumerar até 10 divisões. Combinando vários corantes de divisão, é possível semear vários progenitores individuais no mesmo poço, pois cada corante individual permite a separação dos diferentes descendentes. Este é o princípio por trás do uso de corantes celulares para multiplex, clonal e division-tracking, que foi introduzido pela primeira vez para linfócitos murinos23,24.
Aqui, apresentamos o desenvolvimento do ensaio MultiGen para uso com HSPCs murinos e humanos. Ele permite o teste de muitas células individuais simultaneamente para suas propriedades de diferenciação, divisão e parentesco ex vivo. Este ensaio de alto rendimento, fácil de executar e barato permite medir o fenótipo celular, o número de divisões realizadas, o parentesco celular e a relação clonal com as outras células do poço, tudo ao mesmo tempo. Ele pode ser usado para avaliar quantitativamente o compromisso de destino simétrico e assimétrico, o equilíbrio entre auto-renovação e diferenciação e o número de divisões necessárias para um determinado destino de compromisso. O protocolo requer um classificador de células ativado por fluorescência (FACS) e um citômetro de fluxo com leitor de placas, além do equipamento necessário para a realização da cultura celular. Além do protocolo técnico para execução do ensaio em HSPCs humanos, também fornecemos o quadro detalhado de análise, incluindo os testes estatísticos necessários para avaliar as propriedades celulares relacionadas ao conceito de família celular25. Esse protocolo já foi utilizado com sucesso para descrever o compartimento murino da HSPC26,27.
O protocolo a seguir utiliza como material de partida células CD34+ enriquecidas magneticamente28. Dessa forma, é possível corar e isolar eficientemente os HSPCs humanos de diferentes fontes de sangue (por exemplo, sangue do cordão umbilical, medula óssea, sangue periférico). É importante não descartar a fração CD34– , pois ela será utilizada como parte do protocolo para definir diferentes tipos de controles experimentais. As quantidades e volumes de células mencionados podem ser ampliados ou reduzidos, de acordo com o fluxo de trabalho e as necessidades experimentais. Da mesma forma, o protocolo pode ser adaptado para o estudo de diferentes tipos de progenitores, simplesmente modificando os anticorpos utilizados para as etapas de triagem celular e citometria de fluxo.
Protocol
Representative Results
Discussion
O ensaio MultiGen é um ensaio de alto rendimento, fácil execução e baixo custo, que tem sido fundamental para o estudo de linfócitos23,24,35 e células hematopoéticas murinas 26,27. Aqui, apresentamos um novo desenvolvimento da abordagem que permite decifrar ex vivo a fase inicial do comprometimento humano do HSPC, em nível unicelular, usando cultura de curto prazo (Figura 6). Sistemas de cultura ex vivo de célula única são tipicamente usados para avaliar o destino a longo prazo de HSPCs em células maduras, mas alguns destinos ocorrem mais cedo do que outros36, potencialmente enviesando a análise para menos destinos. Além disso, esses sistemas de cultura geralmente perdem informações sobre divisões durante o compromisso do destino. Os primeiros passos do comprometimento têm se mostrado precoces no início da cultura, às vezes sem divisão26,37, tornando a cultura de curto prazo e a divisão de rastreamento essenciais para estudar o comprometimento precoce do destino. Ao acompanhar simultaneamente o destino, a divisão e o parentesco, este ensaio permite compreender o papel da primeira divisão e a decisão do destino nos HSPCs humanos. Usando o ensaio, é possível inferir após quantas divisões o processo de comprometimento ocorre, o equilíbrio entre auto-renovação e diferenciação para esses progenitores iniciais e como essas propriedades são herdadas ao longo das gerações. Até onde sabemos, este é o único ensaio que permite esses tipos de medidas para HSPCs humanos, com resolução de célula única. Além disso, usando diferentes combinações de corantes de divisão celular, aumentamos o rendimento da análise, tornando este ensaio uma ferramenta valiosa para gerar grandes conjuntos de dados rapidamente. As combinações de corantes permitem acompanhar várias famílias nos mesmos poços, aumentando o número de células disponíveis para análise em cultura de curta duração. O número de combinações poderia ser potencialmente aumentado ainda mais, através da adição de outros corantes (por exemplo, corante amarelo) ou modificando a proporção de CFSE e CTV. No entanto, isso reduz o número de outros parâmetros que podem ser analisados.
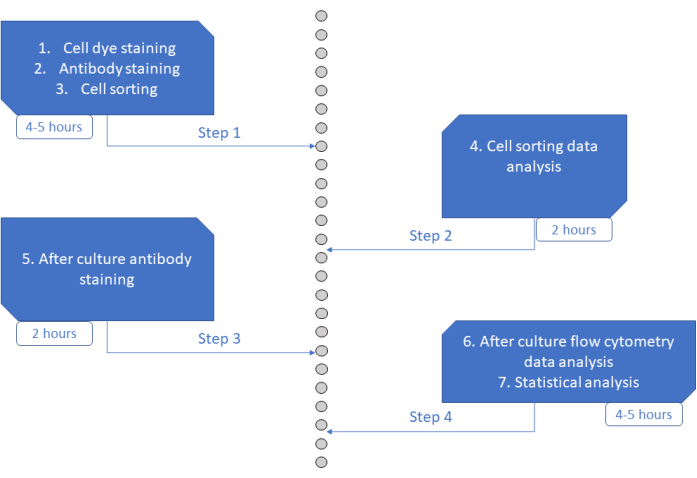
Figura 6: Representação esquemática do protocolo. Clique aqui para ver uma versão maior desta figura.
Para realizar a análise com sucesso, devido ao grande número de poços e ao número reduzido de células a serem analisadas, é necessário executar a análise por citometria de fluxo em um analisador equipado com um leitor de placas. A nova geração de analisadores de bancada é particularmente adaptada a este ensaio, pois a maioria deles tem um volume morto menor para reduzir a porcentagem de perda celular. Isso, por sua vez, garante uma maior eficiência na recuperação da totalidade de cada poço, levando a uma eficiência estimada na faixa de 70%26. Estimar a perda celular durante a aquisição por citometria de fluxo é crucial para a análise de cada família individual. Por exemplo, supondo que não haja morte celular e contando o número de divisões, é possível estimar o número de células por cada família. No entanto, é desejável realizar alguns experimentos confirmatórios, particularmente na estimativa de morte celular nas condições de cultura testadas e medir a taxa de recuperação experimentalmente usando um número definido de células.
Uma das etapas cruciais desse protocolo é a atribuição de pico. Como já mencionado, uma boa distribuição de picos de qualidade é fortemente dependente do isolamento de picos muito estreitos na triagem celular. No entanto, ainda é difícil atribuir o número correto de divisões com base exclusivamente na distribuição. Como a triagem celular e a análise por citometria de fluxo são realizadas em dois equipamentos diferentes, não é possível comparar diretamente a intensidade de cada sinal, de modo que pode ser difícil saber se o primeiro pico observado na extremidade direita do histograma é o pico 0 ou o pico 1. Nesse sentido, poucas soluções são possíveis; Uma maneira é realizar um experimento ortogonal para medir com precisão o número de divisões realizadas por essas células (por exemplo, imagens de células vivas). Outra possibilidade é simplesmente contar o número de células no poço sob um microscópio de campo claro invertido, antes de executar a análise de citometria de fluxo. Isso irá inferir um número médio de divisões (supondo que não haja morte celular). Finalmente, uma solução post-hoc para a atribuição de pico é a detecção de um número incomum de “famílias impossíveis”; Essas famílias são compostas por um número maior do que o possível de células por geração (por exemplo, cinco células na geração 2, ou duas células na geração 1 e uma célula na geração 2). A possibilidade de excluir famílias impossíveis é codificada na etapa de análise estatística e sinaliza a família impossível. Se a ocorrência desses erros for muito alta, é razoável supor que a atribuição de pico precise ser revista.
Nesse protocolo, incluímos alguns exemplos de representação e análise de dados para o ensaio, uma vez que este se tornou uma etapa essencial na geração e interpretação de grandes conjuntos de dados38. O primeiro exemplo é o mapa de calor que mostra a totalidade de todas as células analisadas, organizadas por família. Esta é uma ferramenta eficiente para explorar as propriedades gerais dos dados e possíveis conclusões: as famílias são compostas por múltiplos tipos celulares ou tendem a ser homogêneas em sua composição? As famílias estão espalhadas por várias gerações ou dividem-se, na sua maioria, o mesmo número de vezes? Essa análise exploratória precisa ser complementada com parcelas mais específicas e testes estatísticos. Ele pode ser usado para avaliar quantitativamente o compromisso de destino simétrico e assimétrico, a diferenciação sem divisão, o equilíbrio entre auto-renovação e diferenciação e o número de divisões para um determinado destino de compromisso. É fundamental, durante o planejamento experimental, definir o comprimento da cultura celular de acordo com o tipo de pergunta feita; Por exemplo, para as duas primeiras questões (equilíbrio simétrico/assimétrico e diferenciação sem divisão), o planejamento de etapas muito curtas permite o isolamento de um grande número de famílias que realizaram apenas uma ou nenhuma divisão26. Por outro lado, experimentos mais longos permitem a exploração do número de divisões necessárias para um comprometimento celular específico, pois amostram famílias em diferentes estágios de diferenciação. No entanto, esse método não é projetado para culturas de longo prazo (2-3 semanas), pois a diluição do corante celular não é capaz de rastrear com precisão mais de sete ou oito divisões22. Como consequência, esta ferramenta é principalmente adaptada para estudar o comprometimento precoce de progenitores hematopoiéticos, e não é projetada para tirar conclusões robustas sobre as propriedades de diferenciação a longo prazo dessas células.
O arcabouço estatístico foi desenvolvido especificamente para a análise desse tipo de dado e baseado no conceito depermutações26. Isso foi necessário devido à observação de uma dependência familiar da distribuição do tipo celular e do número de divisões realizadas. Em outras palavras, células que fazem parte da mesma família também são mais propensas a exibir fenótipos semelhantes e dividir o mesmo número de vezes. Embora uma análise aprofundada esteja além do escopo deste trabalho, o conjunto de testes estatísticos fornecido deve ser suficiente para avaliar diferentes condições.
Em conclusão, este protocolo constitui uma ferramenta valiosa para avaliar a dinâmica celular de células-tronco hematopoéticas e progenitoras ex vivo, de forma rápida e de baixo custo. Devido à sua flexibilidade e versatilidade em relação ao ponto de tempo, condições de cultura e tipo de HSPCs analisados, permite testar uma variedade de condições experimentais. Como um ensaio baseado em citometria de fluxo, pode ser implementado na maioria dos laboratórios, e não requer conhecimento prévio extensivo, tornando-se um bom candidato para triagens e experimentos piloto.
Disclosures
The authors have nothing to disclose.
Acknowledgements
Gostaríamos de agradecer aos membros do Institut Curie Flow Facility por sua ajuda na montagem dos experimentos de citometria de fluxo. Queremos também agradecer as contribuições dos demais membros da Equipe Perié, durante múltiplas discussões. Agradecemos à Dra. Julia Marchingo e ao Prof. Phil Hodgkin (Walter End Eliza Hall Institute of Medical Research) por compartilharem seu protocolo de multiplexação de corantes de divisão celular em linfócitos. Agradecemos ao biobanco de sangue de cordão umbilical do hospital Saint Louis por fornecer os recursos biológicos necessários para o desenvolvimento deste protocolo. O estudo foi apoiado por uma bolsa ATIP-Avenir do CNRS e da Fundação Bettencourt-Schueller (para L.P.), subsídios do Labex CelTisPhyBio (ANR-10-LBX-0038) (para L.P. e A.D.), Idex Paris-Science-Lettres Program (ANR-10-IDEX-0001-02 PSL) (para L.P.), o Canceropole INCA Emergence (2021-1-EMERG-54b-ICR-1, para L.P.) e o subsídio ITMO MIIC (21CM044, para L.P.). Além do financiamento do Conselho Europeu de Investigação (ERC) no âmbito do programa de investigação e inovação Horizonte 2020 da União Europeia ERC StG 758170-Microbar (para L.P.), A.D. foi apoiado por uma bolsa da Fondation de France.
Materials
| 1.5 mL polypropylene microcentrifuge tubes | vWR | 87003-294 | |
| 15-mL polypropylene tubes | vWR | 734-0451 | |
| 50-mL polypropylene tubes | vWR | 734-0448 | |
| 96-well U-bottom culture plate | Falcon | 353077 | |
| Anti-human Lin APC | Thermo Fisher | 22-7776-72 | Dilution 1/40 |
| ARIA III | BD | Can be replaced with any FACS sorter able to sort individual cells in 96-wells plate | |
| Carboxyfluorescein succinimidyl ester (CFSE) | Life Technologies | C34570 | |
| Cell Trace Violet (CTV) | Life Technologies | C34571 | |
| Compensation beads | BD | 552843 | |
| Dulbecco’s modified Eagle medium (DMEM) | Life Technologies | 11320033 | |
| Ethylenediaminetetraacetic acid (EDTA) | Thermo Scientific | J62948-36 | Prepare a solution 0.5 M, in sterilised water |
| FC block Fc1.3216 | BD | 564220 | Dilution 1/50 |
| Fetal Bovine Serum (FBS) | Dutscher | S1900-500C | Batch S00CH |
| FlowJo v10.8.1 | BD | ||
| Mouse anti-human CD10 PerCP-5.5, clone HI10a | Biolegend | 312216 | Dilution 1/20 |
| Mouse anti-human CD123 BUV395, clone 7G3 | BD | 564195 | Dilution 1/15 |
| Mouse anti-human CD34 APC-Cy7, clone 581 | Biolegend | 343513 | Dilution 1/40 |
| Mouse anti-human CD38 BV650, clone HB7 | Biolegend | 356620 | Dilution 1/40 |
| Mouse anti-human CD45RA AF700, clone HI100 | BD | 560673 | Dilution 1/20 |
| Mouse anti-human CD45RA PE-Cy7, clone HI100 | BD | 560675 | Dilution 1/20 |
| Mouse anti-human CD90 PE, clone 5E10 | Biolegend | 328110 | Dilution 1/20 |
| Phosphate Buffered Saline (PBS) 1X | Life Technologies | 10010001 | |
| Python | |||
| R | |||
| Sterile 12×75 mm conical polypropylene tubes | Falcon | ||
| ZE5 | Biorad | Can be replaced with any flow cytometry analyzer equipped with a plate reader | |
| Laboratory prepared | |||
| Cell culture media | Depends from the specific experiment. Prepare fresh daily and store at +4 °C until use | ||
| DMEM + 10% FBS | Can be stored in sterile conditions, at +4 °C up to 1 year. To prepare 500 mL, add 50 mL of FBS to 450 mL DMEM | ||
| PBS 1X + EDTA 0.1% | Can be stored in sterile conditions, at room temperature, up to 1 year. To prepare 500 mL, add 3.42 mL of EDTA 0.5 M to 500 mL PBS 1X | ||
| Staining buffer | Can be stored in sterile conditions, at +4 °C up to 1 year. To prepare 500 mL, add 2 mL of EDTA 0.5 M and 1 mL FBS to 500 mL PBS 1X |
References
- Ginhoux, F., Yalin, A., Dutertre, C. A., Amit, I. Single-cell immunology: Past, present, and future. Immunity. 55 (3), 393-404 (2022).
- Ke, M., Elshenawy, B., Sheldon, H., Arora, A., Buffa, F. M. Single cell RNA-sequencing: A powerful yet still challenging technology to study cellular heterogeneity. Bioessays. 44 (11), 2200084 (2022).
- Regev, A., et al. The human cell atlas. Elife. 6, 27041 (2017).
- Laurenti, E., Göttgens, B. From haematopoietic stem cells to complex differentiation landscapes. Nature. 553 (7689), 418-426 (2018).
- Haas, S., Trumpp, A., Milsom, M. D. Causes and consequences of hematopoietic stem cell heterogeneity. Cell Stem Cell. 22 (5), 627-638 (2018).
- Loughran, S. J., Haas, S., Wilkinson, A. C., Klein, A. M., Brand, M. Lineage commitment of hematopoietic stem cells and progenitors: insights from recent single cell and lineage tracing technologies. Experimental Hematology. 88, 1-6 (2020).
- Perié, L., Duffy, K. R. Retracing the in vivo haematopoietic tree using single-cell methods. FEBS Letters. 590 (22), 4068-4083 (2016).
- Yu, V. W. C., et al. Epigenetic memory underlies cell-autonomous heterogeneous behavior of hematopoietic stem cells. Cell. 167 (5), 1310-1322 (2016).
- Ganuza, M., et al. Lifelong haematopoiesis is established by hundreds of precursors throughout mammalian ontogeny. Nature Cell Biology. 19 (10), 1153-1163 (2017).
- Naik, S. H., Schumacher, T. N., Perié, L. Cellular barcoding: A technical appraisal. Experimental Hematology. 42 (8), 598-608 (2014).
- Quek, L., et al. Genetically distinct leukemic stem cells in human CD34 − acute myeloid leukemia are arrested at a hemopoietic precursor-like stage. The Journal of Experimental Medicine. 213 (8), 1513-1535 (2016).
- Karamitros, D., et al. Single-cell analysis reveals the continuum of human lympho-myeloid progenitor cells. Nature Immunology. 19 (1), 85-97 (2018).
- Boitano, A. E., et al. Aryl hydrocarbon receptor antagonists promote the expansion of human hematopoietic stem cells. Science. 329 (5997), 1345-1348 (2010).
- Delaney, C., et al. Notch-mediated expansion of human cord blood progenitor cells capable of rapid myeloid reconstitution. Nature Medicine. 16 (2), 232-236 (2010).
- Fares, I., et al. Cord blood expansion. Pyrimidoindole derivatives are agonists of human hematopoietic stem cell self-renewal. Science. 345 (6203), 1509-1512 (2014).
- Guo, B., Huang, X., Lee, M. R., Lee, S. A., Broxmeyer, H. E. Antagonism of PPAR-γ 3 signaling expands human hematopoietic stem and progenitor cells by enhancing glycolysis. Nature Medicine. 24 (3), 360-367 (2018).
- Vannini, N., et al. The NAD-booster nicotinamide riboside potently stimulates hematopoiesis through increased mitochondrial clearance. Cell Stem Cell. 24 (3), 405-418 (2019).
- Gupta, R., et al. Nov/CCN3 enhances cord blood engraftment by rapidly recruiting latent human stem cell activity. Cell Stem Cell. 26 (4), 527-541 (2020).
- Horwitz, M. E., et al. Omidubicel vs standard myeloablative umbilical cord blood transplantation: results of a phase 3 randomized study. Blood. 138 (16), 1429-1440 (2021).
- Weinreb, C., Rodriguez-Fraticelli, A., Camargo, F. D., Klein, A. M. Lineage tracing on transcriptional landscapes links state to fate during differentiation. Science. 367 (6479), 3381 (2020).
- Loeffler, D., Schroeder, T. Understanding cell fate control by continuous single-cell quantification. Blood. 133 (13), 1406-1414 (2019).
- Tario, J. D., et al. Optimized staining and proliferation modeling methods for cell division monitoring using cell tracking dyes. Journal of Visualized Experiments. (70), e4287 (2012).
- Marchingo, J. M., et al. T-cell stimuli independently sum to regulate an inherited clonal division fate. Nature Communications. 7, 13540 (2016).
- Horton, M. B., et al. Multiplexed division tracking dyes for proliferation-based clonal lineage tracing. Journal of Immunology. 201 (3), 1097-1103 (2018).
- Lehmann, E. L., Romano, J. P., Casella, G. . Testing statistical hypotheses. , 784 (2005).
- Tak, T., et al. HSPCs display within-family homogeneity in differentiation and proliferation despite population heterogeneity. Elife. 10, 360624 (2021).
- Sommerkamp, P., et al. Mouse multipotent progenitor 5 cells are located at the interphase between hematopoietic stem and progenitor cells. Blood. 137 (23), 3218-3224 (2021).
- Kato, K., Radbruch, A. Isolation and characterization of CD34+ hematopoietic stem cells from human peripheral blood by high-gradient magnetic cell sorting. Cytometry. 14 (4), 384-392 (1993).
- Miltenyi, S., Müller, W., Weichel, W., Radbruch, A. High gradient magnetic cell separation with MACS. Cytometry. 11 (2), 231-238 (1990).
- Doulatov, S., et al. Revised map of the human progenitor hierarchy shows the origin of macrophages and dendritic cells in early lymphoid development. Nature Immunology. 11 (7), 585-593 (2010).
- Goardon, N., et al. Coexistence of LMPP-like and GMP-like leukemia stem cells in acute myeloid leukemia. Cancer Cell. 19 (1), 138-152 (2011).
- Laurenti, E., et al. CDK6 levels regulate quiescence exit in human hematopoietic stem cells. Cell Stem Cell. 16 (3), 302-313 (2015).
- Aiuti, A., et al. Lentiviral hematopoietic stem cell gene therapy in patients with Wiskott-Aldrich syndrome. Science. 341 (6148), 1233151 (2013).
- Davison, A. C., Hinkley, D. V. . Bootstrap Methods and their Application. , (1997).
- Horton, M. B., et al. Lineage tracing reveals B cell antibody class switching is stochastic, cell-autonomous, and tuneable. Immunity. 55 (10), 1843-1855 (2022).
- Notta, F., et al. Distinct routes of lineage development reshape the human blood hierarchy across ontogeny. Science. 351 (6269), 2116 (2016).
- Grinenko, T., et al. Hematopoietic stem cells can differentiate into restricted myeloid progenitors before cell division in mice. Nature Communications. 9 (1), 1898 (2018).
- Saeys, Y., Van Gassen, S., Lambrecht, B. N. Computational flow cytometry: Helping to make sense of high-dimensional immunology data. Nature Reviews Immunology. 16 (7), 449-462 (2016).

