- 00:01Concepts
- 03:13gDNA Isolation and Quality Check
- 03:43Isolation of gDNA and gDNA Quality Check
- 05:52Amplification and Purification of 16S rRNA Gene by PCR
- 07:20Analysis of the DNA Sequences
- 09:02Sequence Assembly and Database Search
Sequenciamento de rRNA 16S: uma técnica baseada em PCR para identificar espécies bacterianas
English
Share
Overview
Fonte: Ewa Bukowska-Faniband1, Tilde Andersson1, Rolf Lood1
1 Departamento de Ciências Clínicas Lund, Divisão de Medicina de Infecção, Centro Biomédico, Universidade de Lund, 221 00 Lund, Suécia
O Planeta Terra é um habitat para milhões de espécies bacterianas, cada uma das quais tem características específicas. A identificação de espécies bacterianas é amplamente utilizada na ecologia microbiana para determinar a biodiversidade de amostras ambientais e microbiologia médica para diagnosticar pacientes infectados. As bactérias podem ser classificadas usando métodos convencionais de microbiologia, como microscopia, crescimento em mídia específica, testes bioquímicos e sorológicos e ensaios de sensibilidade a antibióticos. Nas últimas décadas, os métodos de microbiologia molecular revolucionaram a identificação bacteriana. Um método popular é o sequenciamento genético RNA ribossômico 16S (rRNA). Este método não só é mais rápido e mais preciso do que os métodos convencionais, mas também permite a identificação de cepas difíceis de crescer em condições laboratoriais. Além disso, a diferenciação das cepas no nível molecular permite a discriminação entre bactérias fenotipicamente idênticas (1-4).
16S rRNA une-se a um complexo de 19 proteínas para formar uma subunidade 30S do ribossomo bacteriano (5). É codificado pelo gene 16S rRNA, que está presente e altamente conservado em todas as bactérias devido à sua função essencial no conjunto ribossomo; no entanto, também contém regiões variáveis que podem servir como impressões digitais para determinadas espécies. Essas características tornaram o gene 16S rRNA um fragmento genético ideal a ser usado na identificação, comparação e classificação filogenética de bactérias (6).
O sequenciamento genético rRNA 16S é baseado na reação em cadeia de polimerase (PCR) (7-8) seguida pelo sequenciamento de DNA (9). PCR é um método de biologia molecular usado para amplificar fragmentos específicos de DNA através de uma série de ciclos que incluem:
i) Denaturação de um modelo de DNA duplo encalhado
ii) Ressaração de primers (oligonucleotídeos curtos) que são complementares ao modelo
iii) Extensão dos primers pela enzima dna polimerase, que sintetiza uma nova cadeia de DNA
Uma visão geral esquemática do método é mostrada na Figura 1.
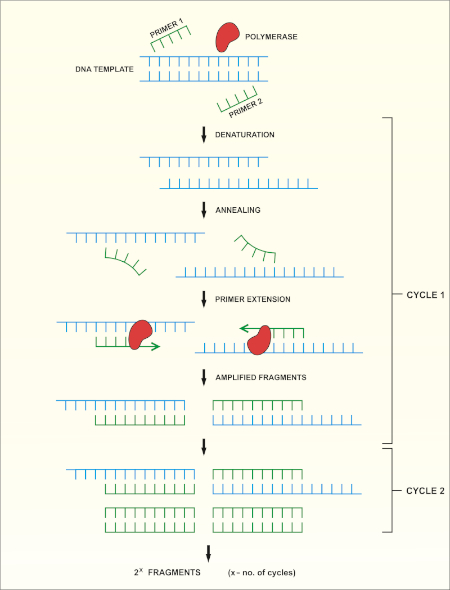
Figura 1: Visão geral esquemática da reação do PCR. Clique aqui para ver uma versão maior desta figura.
Existem vários fatores que são importantes para uma reação de sucesso do PCR, um dos quais é a qualidade do modelo de DNA. O isolamento do DNA cromossômico de bactérias pode ser realizado usando protocolos padrão ou kits comerciais. Deve-se tomar cuidado especial para obter DNA livre de contaminantes que podem inibir a reação da PCR.
Regiões conservadas do gene rRNA 16S permitem o desenho de pares de primer universais (um para a frente e um inverso) que podem se ligar e amplificar a região alvo em qualquer espécie bacteriana. A região alvo pode variar de tamanho. Enquanto alguns pares de primer podem amplificar a maior parte do gene 16S rRNA, outros amplificam apenas partes dele. Exemplos de primers comumente usados são mostrados na Tabela 1 e seus locais de vinculação são retratados na Figura 2.
| Nome do primer | Sequência (5’→3′) | Para a frente/ para trás | Referência |
| 8F b) | AGAGTTTGATCCTGGCTCAG | encaminhar | -1 |
| 27F | AGAGTTTGATCMTGGCTCAG | encaminhar | -10 |
| 515F | GTGCCAGCCCCCGGGGTAA | encaminhar | -11 |
| 911R | GCCCCCGTCAATTCMTTTGA | inverter | -12 |
| 1391R | GACGGGCGGTGTGTRCA | inverter | -11 |
| 1492R | GGTTACCTTGTTACGACTT | inverter | -11 |
Tabela 1: Exemplos de oligonucleotídeos padrão utilizados na amplificação de genes de rRNA 16S a).
a) Os comprimentos esperados do produto PCR gerados usando as diferentes combinações de primer podem ser estimados calculando-se a distância entre os locais de ligação para o primer para a frente e para o primer (ver Figura 2), por exemplo, o tamanho do produto PCR usando o par de primer 8F-1492R é de ~1500 bp, e para o par de primer 27F-911R ~900 bp.
b) também conhecido como fD1

Figura 2: Figura representativa da sequência de rRNA 16S e dos sites de vinculação de primer. As regiões conservadas são coloridas em cinza e regiões variáveis são preenchidas com linhas diagonais. Para permitir a maior resolução, o primer 8F e 1492R (nome baseado na localização na sequência de rRNA) são usados para amplificar toda a sequência, permitindo o sequenciamento de várias regiões variáveis do gene. Clique aqui para ver uma versão maior desta figura.
As condições de ciclismo para PCR (ou seja, a temperatura e o tempo necessários para que o DNA seja desnaturado, enrosco com primers e sintetizado) dependem do tipo de polimerase que é usado e das propriedades dos primers. Recomenda-se seguir as diretrizes do fabricante para uma determinada polimerase.
Após a conclusão do programa PCR, os produtos são analisados por eletroforese de gel agarose. Um PCR bem sucedido rende uma única faixa de tamanho esperado. O produto deve ser purificado antes do sequenciamento para remover primers residuais, desoxiribonucleotídeos, polimerase e tampão que estavam presentes na reação pcr. Os fragmentos de DNA purificados são geralmente enviados para sequenciamento para serviços de sequenciamento comercial; no entanto, algumas instituições realizam sequenciamento de DNA em suas próprias instalações principais.
A sequência de DNA é gerada automaticamente a partir de um cromatgrama de DNA por um computador e deve ser cuidadosamente verificada para a qualidade, pois a edição manual às vezes é necessária. Após esta etapa, a sequência genética é comparada com sequências depositadas no banco de dados de rRNA 16S. As regiões de semelhança são identificadas, e as sequências mais semelhantes são entregues.
Procedure
Applications and Summary
Identifying bacterial species is important for different researchers, as well as for those in healthcare. 16S rRNA sequencing was initially used by researchers to determine phylogenetic relationships between bacteria. In time, it has been implemented in metagenomic studies to determine biodiversity of environmental samples and in clinical laboratories as a method to identify potential pathogens. It enables a quick and accurate identification of bacteria present in clinical samples, facilitating earlier diagnosis and faster treatment of patients.
References
- Weisburg, W.G., Barns, S.M., Pelletier, D.A. and Lane D.J. 16S ribosomal DNA amplification for phylogenetic study. J Bacteriol. 173 (2): 697-703. (1991)
- Drancourt, M., Bollet, C., Carlioz, A., Martelin, R., Gayral, J.P., Raoult D. 16S ribosomal DNA sequence analysis of a large collection of environmental and clinical unidentifiable bacterial isolates. J Clin Microbiol. 38 (10):3623-3630. (2000)
- Woo, P.C., Lau, S.K., Teng, J.L., Tse, H., Yuen, K.Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clin Microbiol Infect. 14 (10):908-934. (2008)
- Tang, Y.W., Ellis, N.M., Hopkins, M.K., Smith, D.H., Dodge, D.E., Persing, D.H. Comparison of phenotypic and genotypic techniques for identification of unusual aerobic pathogenic gram-negative bacilli. J Clin Microbiol. 36 (12):3674-3679. (1998)
- Tsiboli, P., Herfurth, E., Choli, T. Purification and characterization of the 30S ribosomal proteins from the bacterium Thermus thermophilus. Eur J Biochem. 226 (1):169-177. (1994)
- Woese, C.R. Bacterial evolution. Microbiol Rev. 51 (2):221-271. (1987)
- Bartlett, J.M., Stirling, D. A short history of the polymerase chain reaction. Methods Mol Biol. 226:3-6. (2003)
- Wilson, K.H., Blitchington, R.B., Greene, R.C. Amplification of bacterial 16S ribosomal DNA with polymerase chain reaction. J Clin Microbiol. 28 (9):1942-1946. (1990)
- Shendure, J., Balasubramanian, S., Church, G.M., Gilbert, W., Rogers, J., Schloss, J.A., Waterston, R.H. (2017) DNA sequencing at 40: past, present and future. Nature. 550:345-353.
- Lane, D.J. 16S/23S rRNA sequencing. (1991) In Nucleic acid techniques in bacterial systematics. (Goodfellow, M. and Stackebrandt, E., eds.) p.115-175. Wiley and Sons, Chichester, United Kingdom.
- Turner, S., Pryer, K.M., Miao, V.P., Palmer, J.D. (1999) Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. J Eukaryot Microbiol. 46:327-338.
- Fredricks, D.N., Relman, D.A. (1998) Improved amplification of microbial DNA from blood cultures by removal of the PCR inhibitor sodium polyanetholesulfonate. J Clin Microbiol. 36:2810-2816.
- Wilson, K. Preparation of genomic DNA from bacteria. (2001) Curr Protoc Mol Biol. Chapter 2:Unit 2.4.
- Wright, M. H., Adelskov, J., Greene, A.C. (2017) Bacterial DNA extraction using individual enzymes and phenol/chloroform separation. J Microbiol Biol Educ. 18:18.2.48.
- Huang, X., Madan, A. (1999). CAP3: A DNA sequence assembly program. Genome Res. 9:868-877.
Transcript
Earth is home to millions of bacterial species, each with unique characteristics. Identifying these species is critical in evaluating environmental samples. Doctors also need to distinguish different bacterial species to diagnose infected patients.
To identify bacteria, a variety of techniques can be employed, including microscopic observation of morphology or growth on a specific media to observe colony morphology. Genetic analysis, another technique for identifying bacteria has grown in popularity in recent years, due in part to 16S ribosomal RNA gene sequencing.
The bacterial ribosome is a protein RNA complex consisting of two subunits. The 30S subunit, the smaller of these two subunits, contains 16S rRNA, which is encoded by the 16S rRNA gene contained within the genomic DNA. Specific regions of 16S rRNA are highly conserved, due to their essential function in ribosome assembly. While other regions, less critical to function, may vary among bacterial species. The variable regions in 16S rRNA, can serve as unique molecular fingerprints for bacterial species, allowing us to distinguish phenotypically identical strains.
After obtaining a quality sample of gDNA, PCR of the 16S rRNA encoding gene can begin. PCR is a commonly used molecular biology method, consisting of cycles of denaturation of the double-stranded DNA template, annealing of universal primer pairs, which amplify highly conserved regions of the gene, and the extension of primers by DNA polymerase. While some primers amplify most of the 16S rRNA encoding gene, others only amplify fragments of it. After PCR, the products can be analyzed via agarose gel electrophoresis. If amplification was successful, the gel should contain a single band of an expected size, depending upon the primer pair used, up to 1500bp, the approximate length of the 16S rRNA gene.
After purification and sequencing, the obtained sequences can then be entered into the BLAST database, where they can be compared with reference 16S rRNA sequences. As this database returns matches based on the highest similarity, this allows confirmation of the identity of the bacteria of interest. In this video, you will observe 16S rRNA gene sequencing, including PCR, DNA sequence analysis and editing, sequence assembly and database searching.
When handling microorganisms, it is essential to follow good microbiological practice, including using aseptic technique and wearing appropriate personal protective equipment. After performing an appropriate risk assessment for the microorganism or environmental sample of interest, obtain a test culture. In this example, a pure culture of Bacillus subtilis is used.
To begin, grow your microorganism on a suitable medium in the appropriate conditions. In this example, Bacillus subtilis 168 is grown in LB broth overnight in a shaking incubator set to 200 rpm at 37 degrees Celsius. Next, use a commercially available kit to isolate genomic DNA or gDNA from 1.5 milliliters of the B. subtilis overnight culture.
To check the quality of the isolated DNA, first mix five microliters of the isolated gDNA with one microliter of DNA gel loading dye. Then, load the sample onto a 0.8% agarose gel, containing DNA staining reagent, such as SYBR safe or EtBr. After this, load a one kilobase molecular mass standard onto the gel, and run the electrophoresis until the front dye is approximately 0.5 centimeters from the bottom of the gel. Once the gel electrophoresis is complete, visualize the gel on a blue light transilluminator. The gDNA should appear as a thick band, above 10 kilobase in size and have minimal smearing.
After this, to create serial dilutions of the gDNA, label three microcentrifuge tubes as 10X, 100X, and 1000X. Then, use a pipette to dispense 90 microliters of sterile distilled water into each of the tubes. Next, add 10 microliters of the gDNA solution to the 10X tube. Pipette the whole volume up and down to ensure the solution is mixed thoroughly. Then, remove 10 microliters of the solution from the 10X tube and transfer this to the 100X tube. Mix the solution as previously described. Finally, transfer 10 microliters of the solution in the 100X tube, to the 1000X tube.
To begin the PCR protocol, thaw the necessary reagents on ice. Then, prepare the PCR master mix. Since the DNA polymerase is active at room temperature, the reaction set up must occur on ice. Aliquot 49 microliters of the master mix into each of the PCR tubes. Then, add one microliter of template to each of the experimental tubes and one microliter of sterile water to the negative control tube, pipetting up and down to mix. After this, set the PCR machine according to the program described in the table. Place the tubes into the thermocycler and start the program.
Once the program is complete, examine the quality of your product via agarose gel electrophoresis, as previously demonstrated. A successful reaction using the described protocol should yield a single band of approximately 1.5 kilobase. In this example, the sample containing 100X diluted gDNA yielded the highest quality product. Next, purify the best PCR product, in this case, the 100X gDNA, with a commercially available kit. Now the PCR product can be sent for sequencing.
In this example, the PCR product is sequenced using forward and reverse primers. Thus, two data sets, each containing a DNA sequence and a DNA chromatogram, are generated: one for the forward primer and the other for the reverse primer. First, examine the chromatograms generated from each primer. An ideal chromatogram should have evenly spaced peaks with little to no background signals.
If the chromatograms display double peaks, multiple DNA templates may have been present in the PCR products and the sequence should be discarded. If the chromatograms contained peaks of different colors in the same location, the sequencing software likely miscalled nucleotides. This error can be manually identified and corrected in the text file. The presence of broad peaks in the chromatogram indicates a loss of resolution, which causes miscounting of the nucleotides in the associated regions. This error is difficult to correct and mismatches in any of the subsequent steps should be treated as unreliable. Poor chromatogram reading quality, indicated by the presence of multiple peaks, usually occurs at the five prime and three prime ends of the sequence. Some sequencing programs remove these low quality sections automatically. If your sequence was not truncated automatically, identify the low quality fragments and remove their respective bases from the text file.
Use a DNA assembly program to assemble the two primer sequences into one continuous sequence. Remember, sequences obtained using forward and reverse primers should partially overlap. In the DNA assembly program, insert the two sequences in FASTA format into the appropriate box. Then, click the submit button and wait for the program to return the results.
To view the assembled sequence, click on Contigs in the results tab. Then, to view the details of the alignment, select assembly details. Navigate to the website for the basic local alignment search tool, or BLAST, and select the nucleotide BLAST tool to compare your sequence to the database. Enter your sequence into the query sequence text box and select the appropriate database in the scroll down menu. Finally, click the BLAST button on the bottom of the page, and wait for the tool to return the most similar sequences from the database.
In this example, the top hit is B. subtilis strain 168, showing 100% identity with the sequence in the BLAST database. If the top hit does not show 100% identity to your expected species or strain, click on the sequence which most closely matches your query to see the details of the alignment. Aligned nucleotides will be joined by short vertical lines and mismatched nucleotides will have gaps between them. Focusing on the identified mismatched regions, revise the sequence and repeat the BLAST search if desired.
