Introduction
Fosfatidylethanolamine methyltransferase (PEMT) enzymen katalyseren de covalente aanhechting van één of meer methylgroepen met S -adenosylmethionine (SAM) als methylgroepdonor op PE, monomethyl-dimethyl-PE of PE naar monomethyl-PE, dimethyl-PE en / of geeft fosfatidylcholine (PC). Deze enzymen zijn vrijwel overal in dierlijke cellen en schimmels. Ze kunnen ook in sommige planten 1 en ongeveer 10% van de bacteriën, met name die interageren met eukaryoten 2.
PEMTs de biologie van de cel betrokken zijn niet alleen bijdragen aan de productie van PC, die de belangrijkste lipideklasse in dierlijke cellen, maar ook door te voldoen aan andere belangrijke cellulaire functies. In zoogdieren worden PEMTs voornamelijk tot expressie in de lever, waar zij nodig zijn voor normale afscheiding van zeer lage dichtheid lipoproteïne en ze dragen ook bij aan dieet-geïnduceerde obesitas 3, atherosclerose 4 en insuline weerstandkomstig 5. Daarnaast zijn zoogdieren PEMT eveneens uitgedrukt in adipocyten, hoewel lagere niveaus, en deelnemen vetafzetting 6, 7. PEMT rol in kanker ontwikkeling 8, 9 apoptose en celgroei 10 zijn ook aangetoond. In bacteriën zijn PEMT enzymen getoond belangrijk voor normale celgroei 2, virulentie 2 en symbiose met de gastplant 11 zijn.
Het doel en de reden van het huidige protocol is het PEMT activiteit van volledige celextracten te meten zonder de noodzaak om het enzym te zuiveren. Twee verschillende protocollen ontwikkeld om PEMT te meten. De eerste en meest voorkomende meet de overdracht van getritieerd methylgroep van radioactieve SAM op PE, dat is het onderwerp van dit artikel. Dit protocol werd oorspronkelijk ontwikkeld om PEMT activiteit meet van gist 12 en zoogdiercellen (lever) 13 om een underst winnenanding PC biosynthese in deze cellen en de specificiteit van deze enzymen te bepalen. Later is deze techniek toegepast op andere celtypen zoals bacteriën 2 (met een basische pH-waarde voor de test hoewel 15) en protozoa parasieten 14. Deze techniek kan worden gebruikt met gehele celextracten en gezuiverd enzym, en kan mogelijk worden toegepast op elk celextract systeem. Een niet radioactieve assay is ook ontworpen dat steunt op de enzymatische kwantificering van S -adenosylhomocysteine het transmethylatie product van SAM 16. Laatstgenoemde bepaling kan handiger omdat het geen betrekking radioactiviteit maar het is alleen geschikt voor gezuiverde enzymen.
Subscription Required. Please recommend JoVE to your librarian.
Protocol
1. Cell Extract Voorbereiding
- Groeien de Leishmania cellen in een steriele plastic fles afgesloten met luchtdichte deksel bij 26 ° C in een medium van 1x M199 aangevuld met 20 mM HEPES pH 7,4, 100 U / ml penicilline, 100 ug / ml streptomycine, 5 ug / ml heem , 0,35 g / L NACO 2 H, 0,1 mM adenine en 2 ug / ml biopterine zonder schudden. Oogst de cellen door centrifugeren bij 1500 g gedurende 5 minuten bij 4 ° C wanneer zij een celdichtheid van 1-2 x 10 7 / ml bereikt.
- Verwijder het supernatant met een pipet en was de cellen door resuspenderen de celpellet met een serologische pipet in de helft van het volume van de cultuur van koude fosfaatgebufferde zoutoplossing (137 mM NaCl, 2,7 mM KCI, 10 mM Na 2 HPO 4, 1,8 mM KH 2 PO 4, pH 7,4). Gooi celsupernatanten volgens BL2 veiligheidsrichtlijnen.
- Centrifugeer de cellen opnieuw bij 1500 g gedurende 5 minuten bij 4 ° C. Verwijder het supernatant met een pipet. Overgaan to de volgende stap of snap bevriezen de celpellet in vloeibare stikstof voor langdurige opslag bij -80 ° C (tot drie maanden).
- Bereid 2x lysisbuffer (0,5 M sucrose, 0,1 M TrisHCl, pH 7,5, 2 mM EDTA, en 2x proteaseremmer cocktail) en houd het bij 4 ° C op ijs.
- Resuspendeer de celpellet (vers of bevroren) in gelijk volume 2 x lysisbuffer. Voeg 1x volume van glazen kralen. Vortex krachtig bij 4 ° C gedurende 10 minuten.
- Voeg 2 volumes 1x lysis buffer en meng. Centrifuge cel extracten bij 1500 xg bij 4 ° C gedurende 10 minuten om ongebroken cellen en kernen pellet.
- Breng de supernatant met een pipet in een verse centrifugebuis en koel houden celextracten op ijs totdat de proef.
2. Bepaal de eiwitconcentratie van het celextract gebruiken Protein Estimation Kit Zoals bicinchoninezuur Assay
- Bereid de bicinchoninezuur (BCA) oplossing (1 ml / buis) door mengen van de BCA en koper (II) sulfate in een verhouding van 49: 1 (v / v).
- Bereid de eiwitstandaarden van 0, 10, 20, 30, 40, 50, en 60 ug / ml door verdunning van een 10 mg / ml runderserumalbumine (BSA) voorraadoplossing in 1 ml porties van de BCA-oplossing.
- Voeg 2 pi cellen uittreksels 1 ml BCA oplossing in duplo. Incubeer standaarden en eiwit monsters 10 minuten in een voorverwarmde 60 ° C waterbad.
- Doorvoermonsters om ijs 3 min. Meet de absorptie van de standaarden en de eiwitmonsters met een spectrofotometer bij een golflengte van 562 nm.
- Bereken de eiwitconcentratie van de celextracten met de BSA norm als referentie zoals in protocol van de fabrikant beschreven. Verdun celextracten een eiwitconcentratie van 10 mg / met 1x lysisbuffer ml.
3. Enzymatische Assay in 200 pi per buis
LET OP: Draag de volgende stappen in een chemische kap.
- Test elk monster in tweevoud in een 15 mlconische buis. Bereid 20 pi 1 M TrisHCl pH 7,5 per buis en te houden op het ijs. Bereid 2 ml chloroform / methanol (1: 1 (v / v); stopoplossing) bij KT elke buis.
- Pipetteer 20 ul van 1 M TrisHCl pH 7,5 per 15 ml conische buis op ijs.
- Volg de straling veiligheid richtlijnen vanaf hier op. Voeg het equivalent van 0,06 pM (0,2 uCi) S - [Methyl 3 H] adenosyl-L-methionine en 50 uM koud SAM per buis voor een totaal van 50,06 uM SAM. Voeg x pi koud water waar x = 200 (20 (voor buffer) + 20 (voor mobiele extracten) + volume van koude en radioactieve SAM) per buis.
- Overdracht elke conische buis met een voorverwarmde 30 ° C waterbad. Aan elke buis 20 gl celextracten (equivalent van 200 ug eiwit) om de reactie te starten. Incubeer de gewenste tijd (0-45 min).
- Stop de reactie door toevoeging van 2 ml chloroform / methanol (1: 1, v / v; stopoplossing) aan elke buis. Breng de conische buis tot kamertemperatuur (20-256, C).
4. Lipid Extraction
LET OP: Draag de volgende stappen in een chemische kap.
- Voeg 700 ul van water aan elke buis die het monster enzymatische reactie. Vortex krachtig gedurende 30 sec. Centrifugeer bij 1500 g gedurende 5 min bij RT om het organische scheiden van de waterfase.
- Breng de onderste organische fase in een nieuwe 15 ml conische buis met een pipet. Voeg 1 ml water voor elke "onderste fase" met buis en vortex krachtig gedurende 30 sec. Centrifugeer opnieuw bij 1500 xg gedurende 5 min aan de organische scheiden van de waterfase.
- Breng de onderste organische fase in een scintillatie-buisje met een pipet. Droge monsters onder een stroom N2. Gooi het water fasen met de niet-opgenomen radioactieve SAM en de radioactieve conische buizen per straling richtlijnen.
- Voeg 2 ml / tube scintillatievloeistof. Meet de opgenomen radioactiviteit met een scintillatie counter volgens het protocol van de fabrikant en het gebruik instrument.
- Bereken de enzymatische activiteit in nmol / mg eiwit met de volgende algemene formule:
cpm waarde x 10 3 x [totale (radioactief en koud) concentratie van SAM (mm)] x 5 specifieke activiteit van radioactieve SAM (Ci / mmol) x [concentratie radioactieve SAM (mm)]
OPMERKING: de bovenstaande protocol kan worden aangepast voor een SAM-concentratie-afhankelijke of een eiwit afhankelijk PEMT assay. Voor SAM concentratie afhankelijke enzymatische test wordt de tijd constant gehouden (15 min, die in het lineaire gebied) en verschillende hoeveelheden koude SAM wordt toegevoegd aan de test, terwijl voor eiwit afhankelijke PEMT assay SAM concentratie (we kozen 0,05 mM) en tijd worden constant gehouden (15 min). Ook de pH van de buffer kan zo nodig als sommige PEMT enzymen een verschillende optimale pH waarde veranderen.
Subscription Required. Please recommend JoVE to your librarian.
Representative Results
Figuur 1 toont een tijdsafhankelijke PEMT test, die met Leishmania gehele celextract werd uitgevoerd als een enzym bron met endogene PE als substraat. De hoeveelheid radioactiviteit in de organische fase werd gekwantificeerd door scintillatietelling. De verkregen getallen werden gebruikt om de hoeveelheid getritieerd methylgroepen overgebracht naar PE te berekenen. De PEMT activiteit was lineair gedurende ongeveer 20 min. Vervolgens bereikte een plateau bij ongeveer 30 min, waarna het constant bleef nog 15 min. Zoals verwacht werd PEMT activiteit gedetecteerd wanneer er geen celextracten werden toegevoegd aan de assay (figuur 2). Verder werd deze activiteit afgeschaft in aanwezigheid van 100 uM octadecyltrimethylammonium bromide, dat een remmer van L. grote PE methyltransferases LMJ PEM1 en LMJ PEM2 14. PEMT activiteit was ook eiwitconcentratie dependent, en deze activiteit is lineair evenredig met de hoeveelheid eiwit dat de enzymatische assay (figuur 3). Tenslotte werd een SAM concentratieafhankelijke PEMT assay uitgevoerd, waarbij toenemende concentraties SAM werden getest (figuur 4). PEMT activiteit bereikte een plateau bij SAM concentratie van ongeveer 0,5 mm. Alles bij elkaar, deze vier onderzoeken tonen aan dat PEMT activiteit specifiek en kan worden gemeten van volledige celextracten zonder dat het enzym (en) te zuiveren.
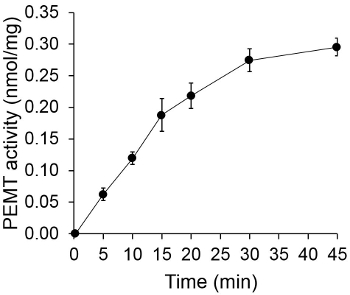
Figuur 1. Tijdsafhankelijke PEMT assay. De enzymatische bepaling werd tweemaal uitgevoerd in duplo met 0,2 mg van hele Leishmania celextracten als functie van de tijd. PEMT activiteit wordt voorgesteld als nmol methylgroepen overgebracht naar PE per mg eiwit en per hr. Voor tijd "O", werden celextracten eerst gemengd met 2 ml stopoplossing van chloroform / methanol (1: 1, betrokken op volume) alvorens toegevoegd aan de testoplossing bevattende SAM. Standaard afwijkingen worden getoond.
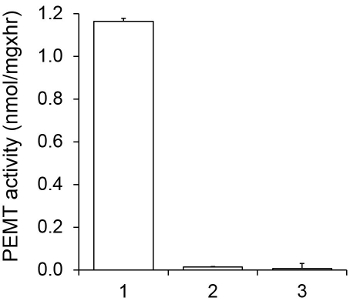
Figuur 2. Specificiteit van PEMT activiteit. PEMT assay tweemaal in tweevoud in de aanwezigheid van 0,05 mM SAM werd uitgevoerd gedurende 15 min. 1, 0,2 mg proteïne-extract; 2, geen mobiele extract; 3, 0,2 mg proteïne en 0,1 mM octadecyltrimethylammonium bromide. Standaard afwijkingen worden getoond.
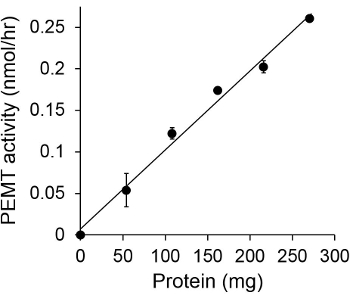
Figuur 3. Eiwit afhankelijk PEMT assay. De enzymatische bepaling werd tweemaal uitgevoerddupliceren zonder (point "O") of aanwezigheid van verschillende hoeveelheden Leishmania eiwitten met 0,05 mM SAM gedurende 15 min. Standaard afwijkingen worden getoond.
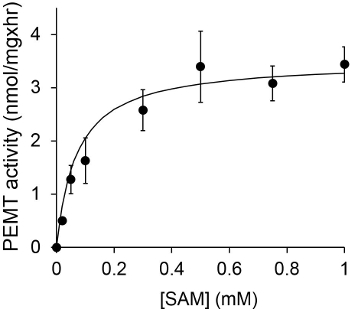
Figuur 4. De PEMT assay werd tweemaal in duplo uitgevoerd met 0,2 mg van hele Leishmania celextracten in aanwezigheid van verschillende concentraties SAM gedurende 15 min. Standaard afwijkingen worden getoond.
Subscription Required. Please recommend JoVE to your librarian.
Discussion
Deze eenvoudige, snelle PEMT test maakt de kwantificering van gemethyleerde vormen van PE als gevolg van de overdracht van radioactieve methylgroepen van SAM op PE middels gehele celextract als eiwitbron. Het is snel, gevoelig en reproduceerbaar, en ook geschikt voor gezuiverde enzymen 17. Monomethyl- of dimethyl-PE kan worden toegevoegd aan de test indien het methyltransferase van belang is specifiek voor deze substraten plaats van PE 12,13,18,19. Als PEMT gezuiverde enzym wordt gebruikt, kan PE worden toegevoegd aan het assay. Een beperking van dit protocol is dat de test niet de reactie producten (monomethyl-PE, dimethyl-PE of PC) te identificeren. Echter, de identiteit van de reactieproducten (monomethyl-PE, dimethyl-PE, PC) verder geanalyseerd door eendimensionale dunnelaagchromatografie zoals beschreven in 20,21. Verder kunnen sommige van de SAM afbraakproducten zoals S -adenosylhomocysteine en 5'-methylthioadenosine kan PEMT b activiteit remmeny feedback inhibitie. Echter, Leishmania bezit een S -adenosylhomocysteine hydrolase 22, die splitst S -adenosylhomocysteine in adenine en S -ribosylhomocysteine en een methylthioadenosine fosforylase, die adenine en methylthioribose-1-fosfaat 23 produceert. Het is echter niet bekend of S -adenosylhomocysteine hydrolase en methylthioadenosine fosforylase activiteiten hoog genoeg zijn om efficiënt te metaboliseren S -adenosylhomocysteine en methylthioadenosine, respectievelijk, zodat geen remming van PEMT activiteit plaatsvindt. In het geval S -adenosylhomocysteine hydrolase en / of 5'-methylthioadenosine metabolische enzymen afwezig in de cel van belang, toevoeging van de respectieve gezuiverde recombinante enzymen kunnen worden toegevoegd aan de test feedback remming door SAM afbraakproducten 24,25 verlichten, 26.
Er zijn vier kritische stappen in dit protocol: i)de proteaseremmer cocktail poeder moet de lysisbuffer wordt toegevoegd juist voor gebruik (stap 1,1); ii) het gehele celextracten worden gebruikt binnen de volgende uren na bereiding (na stap 1,4); iii) in de lipide-extractie stappen (stap 4,2 en 4,4), voorzichtigheid moet worden toegepast op geen van de interfase en waterfase, die de overmaat tritium SAM bevat overdragen, en iv) het radioactieve S aliquot - [Methyl- 3 H] adenosyl-L-methionine en koud reagens na ontvangst als herhaalde cycli van bevriezen en ontdooien degraderen deze in 5'-methylthioadenosine en homoserine lacton gevolgd door hydrolyse tot adenine en S -pentosylmethionine 26,27,28, die kan verantwoordelijk zijn voor het gebrek aan meetbare PEMT activiteit. Bad volledige celextracten kan ook verantwoordelijk zijn voor geen enkele enzymatische activiteit. In dit geval kan de kwaliteit van het celextract worden bepaald door het meten van een bekende enzymatische activiteit. Laatste, gebrek aan PEMT activiteit kan rekenen op de PEMT enZyme specifiek zijn voor monomethyl-PE of PE-dimethyl plaats PE. Toevoeging van monomethyl-PE en / of dimethyl-PE substraten aan de test kan PEMT activiteit te herstellen.
Een alternatief is niet-radioactieve SAM afhankelijke methyltransferase-bepaling ontwikkeld, die is gebaseerd op de enzymatische kwantificering van homocysteïne, de transmethylatie produkten van SAM 29. Dit protocol werd met succes toegepast op PE specifieke methyltransferase activiteit 16 te meten. Helaas, deze test is alleen geschikt voor gezuiverd enzym en niet te gehele celextract, en is de identiteit van de reactieproducten ook niet bekend.
Het onderhavige protocol kan eventueel worden toegepast op elk celtype. Daarnaast kan deze test worden gebruikt om potentiële geneesmiddelen specifiek voor een PEMT van belang in de context van het testen van nieuwe antimicrobiële verbindingen tegen bacteriële infecties of nieuwe geneesmiddelen tegen door dieet geïnduceerde atherosclerose te bestrijden testen, obesitasEn insulineresistentie.
Subscription Required. Please recommend JoVE to your librarian.
Disclosures
Geen belangenconflict verklaard
Materials
| Name | Company | Catalog Number | Comments |
| S-[Methyl-3H]adenosyl-L-methionine (specific activity of 5-15 Ci/mMole) | Perkin Elmer | NET155050UC | Aliquot the reagent and freeze at -20 °C; follow radiation safety guidelines while using this reagent |
| Protease inhibitor cocktail | Roche Life Sciences | 11836170001 | dilute it fresh |
| Glass beads, acid washed, 425-600 mm | Sigma Aldrich | G8772 | |
| Bicinchoninic acid solution | Sigma Aldrich | B9643 | |
| Copper (II) sulfate | Sigma Aldrich | C2284 | |
| Scintillation counter MicroBeta2 with 1-detector | Perkin Elmer | 2450-0010 | |
| Spectrophotometer Biomate 3 | Thermo Scientific | 840208300 | |
| BSA stock solution (10 mg/ml) | New England Biolabs | B9001S | |
| Scintillation liquid | Research Product International Corp | 111198 | |
| S-(5'-Adenosyl)-L-methionine chloride (hydrochloride) | Cayman Chemicals | 13956 | dilute the reagent in 20 mM HCl and freeze aliquots at -80 °C |
References
- Keogh, M. R., Courtney, P. D., Kinney, A. J., Dewey, R. E. Functional characterization of phospholipid N-.methyltransferases from Arabidopsis and soybean. J Biol Chem. 284 (23), 15439-15447 (2009).
- Geiger, O., Lopez-Lara, I. M., Sohlenkamp, C. Phosphatidylcholine biosynthesis and function in bacteria. Biochim Biophys Acta. 1831 (3), 503-513 (2013).
- Gao, X., et al. Decreased lipogenesis in white adipose tissue contributes to the resistance to high fat diet-induced obesity in phosphatidylethanolamine N-.methyltransferase-deficient mice. Biochim Biophys Acta. 1851 (2), 152-162 (2015).
- Zhao, Y., et al. Lack of phosphatidylethanolamine N-.methyltransferase alters plasma VLDL phospholipids and attenuates atherosclerosis in mice. Arterioscler Thromb Vasc Biol. 29 (9), 1349-1355 (2009).
- Vance, D. E. Phospholipid methylation in mammals: from biochemistry to physiological function. Biochim Biophys Acta. 1838 (6), 1477-1487 (2014).
- Nishimaki-Mogami, T., Suzuki, K., Takahashi, A. The role of phosphatidylethanolamine methylation in the secretion of very low density lipoproteins by cultured rat hepatocytes: rapid inhibition of phosphatidylethanolamine methylation by bezafibrate increases the density of apolipoprotein B48-containing lipoproteins. Biochim Biophys Acta. 1304 (1), 21-31 (1996).
- Noga, A. A., Zhao, Y., Vance, D. E. An unexpected requirement for phosphatidylethanolamine N-.methyltransferase in the secretion of very low density lipoproteins. J Biol Chem. 277 (44), 42358-42365 (2002).
- Li, D., et al. Epigenetic repression of phosphatidylethanolamine N-.methyltransferase (PEMT) in BRCA1-mutated breast cancer. Oncotarget. 5 (5), 1315-1325 (2014).
- Cui, Z., Houweling, M., Vance, D. E. Suppression of rat hepatoma cell growth by expression of phosphatidylethanolamine N-.methyltransferase-2. J Biol Chem. 269 (40), 24531-24533 (1994).
- Cui, Z., Shen, Y. J., Vance, D. E. Inverse correlation between expression of phosphatidylethanolamine.N-.methyltransferase-2 and growth rate of perinatal rat livers. Biochim Biophys Acta. 1346 (1), 10-16 (1997).
- Minder, A. C., de Rudder, K. E., Narberhaus, F., Fischer, H. M., Hennecke, H., Geiger, O. Phosphatidylcholine levels in.Bradyrhizobium japonicum. membranes are critical for an efficient symbiosis with the soybean host plant. Mol Microbiol. 39 (5), 1186-1198 (2001).
- Kodaki, T., Yamashita, S. Yeast phosphatidylethanolamine methylation pathway. Cloning and characterization of two distinct methyltransferase genes. J Biol Chem. 262 (32), 15428-15435 (1987).
- Tanaka, Y., Amano, F., Maeda, M., Nishijima, M., Akamatsu, Y. Purification and properties of phosphatidyl-N-.monomethylethanolamine N-.methyltransferase, the enzyme catalyzing the second and the third steps in the phosphatidylethanolamine N-.methyltransferase system, from mouse liver microsomes. Jpn J Med Sci Biol. 43 (3), 59-73 (1990).
- Bibis, S. S., Dahlstrom, K., Zhu, T., Zufferey, R. Characterization of Leishmania major phosphatidylethanolamine methyltransferases LmjPEM1 and LmjPEM2 and their inhibition by choline analogs. Mol Biochem Parasitol. 196 (2), 90-99 (2014).
- deRudder, K. E., Thomas-Oates, J. E., Geiger, O. Rhizobium meliloti. mutants deficient in phospholipid N-.methyltransferase still contain phosphatidylcholine. J Bacteriol. 179 (22), 6921-6928 (1997).
- Aktas, M., Narberhaus, F. In vitro characterization of the enzyme properties of the phospholipid N-.methyltransferase PmtA from Agrobacterium tumefaciens. J Bacteriol. 191 (7), 2033-2041 (2009).
- Ridgway, N. D., Vance, D. E. Phosphatidylethanolamine N-.methyltransferase from rat liver. Methods Enzymol. 209, 366-374 (1992).
- Gaynor, P. M., Carman, G. M. Phosphatidylethanolamine methyltransferase and phospholipid methyltransferase activities from Saccharomyces cerevisiae. Enzymological and kinetic properties. Biochim Biophys Acta. 1045 (2), 156-163 (1990).
- Arondel, V., Benning, C., Somerville, C. R. Isolation and functional expression in Escherichia coli. of a gene encoding phosphatidylethanolamine methyltransferase (EC 2.1.1.17) from Rhodobacter sphaeroides. J Biol Chem. 268 (21), 16002-16008 (1993).
- Wessel, M., Klusener, S., Godeke, J., Fritz, C., Hacker, S., Narberhaus, F. Virulence of Agrobacterium tumefaciens. requires phosphatidylcholine in the bacterial membrane. Mol Microbiol. 62 (3), 906-915 (2006).
- Klusener, S., Aktas, M., Thormann, K. M., Wessel, M., Narberhaus, F. Expression and physiological relevance of Agrobacterium tumefaciens. phosphatidylcholine biosynthesis genes. J Bacteriol. 191 (1), 365-374 (2009).
- Henderson, D. M., et al. Cloning of the gene encoding Leishmania donovani.S.-adenosylhomocysteine hydrolase, a potential target for antiparasitic chemotherapy. Mol Biochem Parasitol. 53 (1-2), 169-183 (1992).
- Koszalka, G. W., Krenitsky, T. A. 5'-Methylthioadenosine (MTA) phosphorylase from promastigote of Leishmania donovani. Purine and Pyrimidine Metabolism in Man V, Adv Exp Med Biol. Nyhan, W. L., Thompson, L. F., Watts, R. W. E. 131, Springer US. 559-563 (1986).
- Biastoff, S., Teuber, M., Zhou, Z. S., Dräger, B. Colorimetric activity measurement of a recombinant putrescine N.-methyltransferase from Datura stramonium. Planta Med. 72 (12), 1136-1141 (2006).
- Hendricks, C. L., Ross, J. R., Pichersky, E., Noel, J. P., Zhou, Z. S. An enzyme-coupled colorimetric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 326 (1), 100-105 (2004).
- Cannon, L. M., Butler, F. N., Wan, W., Zhou, Z. S. A stereospecific colorimetric assay for (S.,S.)-adenosylmethionine quantification based on thiopurine methyltransferase-catalyzed thiol methylation. Anal Biochem. 308 (2), 358-363 (2002).
- Hoffman, J. L. Chromatographic analysis of the chiral and covalent instability of S.-adenosyl-L-methionine. Biochemistry. 25 (15), 4444-4449 (1986).
- Wu, S. E., Huskey, W. P., Borchardt, R. T., Schowen, R. L. Chiral instability at sulfur of S.-adenosylmethionine. Biochemistry. 22 (12), 2828-2832 (1983).
- Dorgan, K. M., et al. An enzyme-coupled continuous spectrophotometric assay for S.-adenosylmethionine-dependent methyltransferases. Anal Biochem. 350 (2), 249-255 (2006).



