적정 수용 체 Ligand 상호 작용의 분리 상수를 결정 하는 방법으로 ELISA
Summary
자세한 프로토콜 ELISA 적정 수행을 설명 합니다. 또한, 새로운 알고리즘 적정 ELISAs를 평가 하 고 결정 플레이트 움직일 수용 체 성 ligand의 바인딩의 분리 상수를 제공 됩니다.
Abstract
분리 상수 바인딩 평형에 두 업체 간의 상호 작용을 설명 하 고 그들의 선호도의 측정 이다. 그것을 비교할 다른 ligands, 예를 들면, 경쟁 저 해제, 단백질 isoforms 및 돌연변이, 바인딩 파트너에 게 그들의 바인딩 강도 대 한 중요 한 매개 변수입니다. 분리 상수 플로팅 바인딩 곡선으로 바인딩된 대 무료 ligand의 농도 의해 결정 됩니다. 반면, 적정 곡선, 바인딩된 ligand의 농도에 비례 하는 신호 추가 ligand의 총 농도 대 한 플롯은 훨씬 쉽게 기록할 수 있습니다. 시선 및 효소 연결 된 immunosorbent 분석 결과 (ELISA)에 의해 신호를 감지할 수 있습니다. 이것은 뱀 독에서 파생 된 rhodocetin α2β1 integrin의 고정된 대상 도메인에 바인딩을 측정 ELISA 적정에 대 한 프로토콜에서 궁 행. 적정 ELISAs 다목적 널리 사용 됩니다. 두 단백질은, 순수 하 고 그들의 농도 알려진 고정된 수용 체와 녹는 ligand 상호 작용 단백질의 어떤 쌍 든 지를 사용할 수 있습니다. 지금까지 어려움은 적정 곡선에서 분리 상수 결정 되었습니다. 이 연구에서는 적정 곡선을 기본 수학 함수 소개 된다. 채도 수확량의 어떤 오류가 그래픽 평가 없이이 알고리즘 아닌-선형 회귀를 통해 수학 평가 의해 직접 원시 데이터 (추가 ligand의 다른 농도에서 농도 신호)의 처리를 허용 한다. 따라서, 여러 적정 곡선을 동시에 기록 하 고 분리 상수 및 바인딩-활성화 수용 체의 농도 그들의 사이에서 특성 매개 변수 집합으로 변형 수 그리고 그들은 통계적으로 평가 될 수 있다. 이 알고리즘와 결합 하면, 적정 ELISAs 분리 상수를 직접 제시의 이점을 얻을. 따라서, 그들은 미래에 보다 효율적으로 사용할 수 있습니다.
Introduction
분리 상수 K는 그것의 ligand (L)에 대 한 수용 체 (R)의 선호도 설명 하는 핵심 매개 변수입니다. 대량 작업의 법칙에 따라, K는 수용 체 ligand 복잡 한 RL R 수용 체와 리간드 l: 분리 평형에 대 한 정의
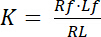 공식 1
공식 1
수용 체와 ligand의 무료/언바운드 상태를 나타내는는 인덱스 f 와. RL, 수용 체 ligand 복합물의 농도 수용 체 바인딩 ligand Lb의 농도. 수용 체 Rt 의 총 농도 무료 수용 체 Rf 와 수용 체 ligand 바인딩 Rb 의 합 = Lb, 분리 상수 또한 쓸 수:
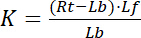 공식 2
공식 2
따라서, 채도 항복 Y, 바인딩된 리간드 Lb 수용 체 Rt의 총 농도의 관계의 일부분으로 정의
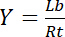 식 3
식 3
무료 리간드 Lf의 농도에 따라 다릅니다.
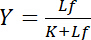 방정식 4
방정식 4
이 쌍곡선 관계 수용 체 ligand 상호 작용의 바인딩 곡선을 설명 하 고의 음모 무료 리간드 Lf의 농도의 기능으로 바인딩된 리간드 Lb 의 농도 보여 줍니다. 바인딩 곡선에서 분리 상수 K 반 최대한 채도 항복 무료 ligand의 농도 파생 될 수 있습니다. 또한, 다른 알고리즘을 바인딩 커브를 선형화 설립 되었습니다, Klotz1,2또는 Scatchard 또는 헤 인 즈 (Bisswanger3검토) 변환에 의해 더블-상호 음모 등. 그러나, 모든 알고리즘 asymptotically 무료 ligand 바인딩 곡선에서의 높은 농도에서 접근 된다, 채도 수율의 최대값 그래픽 사전 평가에서 추정 될 수 있다 며 따라서 문제에서 고통 오류 발생.
또한, 바인딩 곡선의 결정 바인딩 평형 동안 자유롭고 바인딩 ligand의 정량화를 해야합니다. 이 위해 무료 ligand 수용 체 바인딩 ligand에서 분리 하 여 정량 있다. 따라서, ligand와 수용 체는 단백질 수용 체는 반대로 비 단백질 ligand 등 그들의 속성에 다 해야 합니다. 두 바인딩 파트너 단백질 인 경우에, 그들은 그들의 크기, 비용, 또는 다른 분자 기능에서 구별할 수 있다. 그럼에도 불구 하 고, 소규모 바인딩 접근에 있는 ligand 농도의 정량화 어려운 작업입니다. ligand의 방사성 라벨 자주 되었습니다 바운드 ligand의 낮은 농도 검출 하는 데 필요한 특히 수용 체의 상당한 양의 없는 경우 사용할 수 또는 저렴 한. 또한, 수용 체 바인딩 ligand 무시할 수 비 방식에서 중과 격리 후 분리 수 있습니다. 따라서, 평형 젤 여과4,5, 모 세관 전기 이동 법 및 펄스 베이스6, 같은 복잡 한 방법을 수용 체 바인딩 ligand를 계량 하 고 무료 ligand에서 그것을 분리 하는 데 필요한 있습니다.
이러한 바인딩 분석 실험, 달리 적정 실험에서는 바운드 및 무료 ligand의 정량적 분리가 필요 하지 않습니다. 이 위해, 일정 농도에서 수용 체 추가 ligand의 다른 농도와 적정 이다. 수용 체에 바인딩하여 바인딩된 ligand는 무료 ligand에서 그것을 구별 하 고, 예를 들어, 광도, fluorometry, 또는 항 체 검출에 의해 측정 되는 생물 속성이 있습니다. 따라서, S, 채도에 비례 하는 신호 Y를 항복 하 고 따라서 또한 수용 체 바인딩 ligand (Lb)의 농도를 감지 추가 ligand (Lt)의 총 농도의 기능으로. 매개 변수, 신호 S 및 추가 ligand의 총 농도 바운드 및 무료 ligand의 농도 보다 직접적이 고 쉽게 방식으로 측정할. 특히, 효소 연결 된 immunosorbent 분석 결과 (ELISA)에 의해 수용 체 바인딩 ligand 감지 다 잘 결정 접시에 100 µ L 아래에 샘플 볼륨의 감소 뿐만 아니라 여러 ligand 농도의 병렬 측정 허용. ELISA 적정에서 수용 체 물리적으로 동일한 농도에서 결정 플레이트에 흡착 이며 수용 성 리간드로 적정. 수용 체는 움직일 수 플라스틱 표면에 본질적으로 소수 성 흡착에 의해. Langmuir´s 흡착 등온선7에 따라 가능성이 비선형 관계에 수용 체의 코팅 농도와 고정된 수용 체 상호의 표면 농도. 흡착된 수용 체 분자의 전체 숫자 이외에 그들의 활동 상태가 적정 분석에 대 한 또 다른 중요 한 매개 변수입니다. 만 움직일 수 수용 체 ligand 바인딩 활동 유지, 적정 분석 결과 대 한 관련 되 고 결국 직접 확인할 수 없는 적정 분석 결과의 활성 수용 체 Rt 의 총 농도에 기여.
플라스틱 표면에 고정된 수용 체에 포함 되지 않습니다이 사이트는 ligand 같은 다른 단백질을 흡착 하는 경향이 있다. 같은 플라스틱 표면 사이트 리간드의 물리적 흡착 수용 체 바인딩 ligand로 유사한 신호에서 아직 일반적인 방식으로 발생 합니다. 이 일반적인 신호는 단백질으로 코팅 하지 아직 소 혈 청 알 부 민 (BSA)와 차단 될 것입니다 결정 접시의 플라스틱 표면 사이트를 줄이기 위해. 그러나, 일부 수용 체 ligand 적정 분석에 대 한 일반적인 배경 신호를 관찰할 수 있습니다. 그런 다음, 다른 차단 에이전트 0.04% 또는 0.2% 젤라틴의 솔루션 등 트윈 20는 것이 좋습니다.
수용 체에 바인딩을, 후 무료 ligand 두 세척 단계에 의해 제거 됩니다. 바운드 ligand 수용 체, 결정 잘의 플라스틱 표면에 움직일 이며 선택적으로 화학 기정에 의해 강화 된 남아 있습니다. 후속 공유 교차 결합의 바인딩된 ligand와도 함께 고정된 수용 체에 대 한 버퍼 물질 트리 ligand 바인딩에 어떤 변화 없이 HEPES에 대 한 대체 됩니다. HEPES, 트리 스, 달리도 비활성화 하지 않습니다. 도와 공유 교차 결합 그것의 수용 체 바인딩된 ligand 해결 이후 세척 및 인큐베이션 단계 동안 그것의 분리를 방지 한다. 따라서, 수용 체 ligand 상호 작용 화학적으로 고정 하 고 영장 적정 곡선을 세척 및 외피의 후속 단계에 의해 영향을 받지 않습니다. 그러나, 글 고정은 ligand와 같은 방식으로 그들의 상호 작용의 감소 또는 폐지는 수용 체에 화학적으로 수정할 수 있습니다. 또한,는 리간드 내에서 epitopes의 단일 클론 항 체 바인딩된 ligand 척도를 사용 하는 경우에 특히 탐지 항 체 바인딩 선호도 변경할 수 있습니다. 비록 어느 쪽도 정착의 이러한 부작용의 발생이 적정 ELISA에서에서,도로 시험의 감도 적정 실험 전에 모든 수용 체 ligand 상호 작용에 대 한 테스트 되어야 한다. 고정, 후 초과 트리 스 포함 된 버퍼와 3 개의 세척 단계에서 제거 됩니다. 트리 스 nonspecifically 후속 단계에서 항 체를 검출 반응 수 있는 나머지 알데하이드 그룹은.
바인딩된 ligand의 양 광도 ELISA 신호 미를 제공 하는 효소 연결 된 항 체와 계량 이 추가 총 ligand 농도 Lt 대 플롯은 각 잘 하. 쉽게 인수에 불구 하 고 적정 곡선 쌍곡선 함수 바인딩 곡선 달리 아니다. 또한, 그것 되었습니다 분명 적정 곡선에서 분리 상수 K를 계산 하는 방법. 시선 인수 적정 곡선을 선형화 하는 알고리즘을 독립적으로 보고 되었습니다 하지 Stockell8 및9에 있는 Heyn와 Weischet 의해, 하지만 그들은 최대 신호 추정의 불확실성으로 인해 미흡 값은 채도 수익률 추가 ligand의 높은 농도에 접근 한다.
여기, ELISA 적정 및 비 선형 회귀 알고리즘 기술 적정 곡선에서 수용 체 ligand 상호 작용에 대 한 분리 상수 K를 파생 된다. 이 프로토콜은 뱀 독 파생 억제제와 integrin α2β1의 콜라겐 바인딩 A 도메인의 상호 작용에 대 한 궁 행. Integrins는 세포 접착 분자, 주변 기질 또는 기본 지하실 멤브레인10,11셀의 앵커리지 중재. 또한, integrins 모집 추가 신호 분자 및 새로운 휴대 organelles, 세포-매트릭스 상호 작용12,13, 에 adhesomes를 형성 하 여 세포와 세포 외 매트릭스 사이 중요 한 신호 전달 14. 콜라겐, α2β1 integrin의 ligand의 가장 풍부한 단백질 이며 결합 조직15의 중요 한 비 계 구성 요소. Α2β1 integrin과 콜라겐 사이 상호 작용은 integrin α2 소 단위의 A 도메인에 의해 중재 됩니다. Integrin α2A-도메인 포함 divalent 양이온, 콜라겐 바인딩 필요 하 고 그것의 구조를 안정화 합니다. 야생-타입 폼으로 있는 것과 같은 표면 노출 찌 꺼 기 Y216는 글리신에 대 한 대체 했다 α2A 도메인의 돌연변이 세균 식 시스템에서 recombinantly 생산를 수 통해 그들의 올리고-그의-태그는 NiNTA와 절연 TRIS 버퍼 염 분 (TBS, 50 m m TRIS/HCl, pH 7.4, 150 mM NaCl) 포함 2 m MgCl2m16에 대 한 후속 투와 superflow 열. 그들의 농도 bicinchoninic 산 분석 결과 (BCA)로 결정 했다 그리고 그들의 순도 기존의 SDS 페이지 테스트 Coomassie 화려한 블루 R250 스테인드.
Α2β1 integrin과 콜라겐 사이 상호 작용은 뱀 독 구성 요소, 말레이 모사 (Calloselasma rhodostoma)16,17에서 rhodocetin의 바인딩에 의해 차단 됩니다. 이 적정 ELISA에에서 수용 성 리간드로 사용, rhodocetin 원유에서 정제는 앞에서 설명한16독. HEPES 버퍼링 염 분 (HBS; 10 mM HEPES/NaOH, pH 7.4, 150 mM NaCl)에 용 해 하 고-20 ° c.에 냉동 저장 그것의 농도 BCA에 의해 결정 되었다 고 그 순도 SDS 페이지에 의해 입증 되었다. 길 항 제로 rhodocetin 뿐만 아니라 콜라겐은 integrin에 바인딩 차단 α2β1 A-도메인, 하지만 또한 그로 인하여 어떤 세포 또는 혈소판18로 콜라겐에서 신호 방지 integrin의 비활성 형태를 안정 시킨다. 그것은 그것의 수용 체 표적으로 rhodocetin의 분리 상수를 결정 하 고 따라서 그것의 분자 메커니즘 및 제약 잠재적인 예를 들어, 지원을 에이전트로 해명에 생물 의학 중요성의. 이 위해, 그것의 평가 포함 하 여 설명 하는 ELISA 적정을 1:1 상호 작용 산출할와 거의 모든 수용 체 ligand 상호 작용에 적용 됩니다.
Protocol
Representative Results
Discussion
ELISA 적정 수용 체 ligand 상호 작용의 분리를 결정 하는 다양 한 테스트 시스템입니다. 적정으로 ELISA circumvents 자유롭고 바운드 ligands를 효과적으로 분리 하 고 양적, 그들의 농도 분석을 실질적으로 더 많은 연구 필요성 및 간행물 바인딩 곡선을 기록 하는 대신 적정 ELISAs 고용 . 또한, 적정 ELISAs 수행 수용 체 그리고 ligand의 합리적으로 낮은 금액을 필요로 하는. 분리 상수의 정확한 분석에 대 한 수?…
Disclosures
The authors have nothing to disclose.
Acknowledgements
프로토콜 및 알고리즘 도이치 가운데 (DFG 그랜트 SFB1009 A09와 EB177/13-1)에 의해 자금을 프로젝트 내에서 개발 되었다. 저자는 바바라 Schedding 및 기술 지원에 대 한 펠릭스 Schmalbein 박사 Niland 비판적 원고를 읽기 위한 감사 합니다.
Materials
| TRIS | neoFrox | 1125KG001 | |
| HEPES | Sigma-Aldrich | H4034 | |
| NaCl | Applichem | 1,316,591,214 | |
| MgCl2 | Merck | 172571 | |
| integrin a2A, wild-type and mutant, recombinant | isolated in author's lab | ||
| NiNTA superflow column | Qiagen, Germany | 30821 | |
| Coomassie-Brilliant Blue R250 | Serva | 35050 | |
| bicinchoninic acid assay (BCA), protein concentration determination kit | Fisher Scientific | 23225 | |
| bovine serum albumine (BSA), fraction V | Applichem | A1391 | |
| 25 % solution of glutaraldehyde | Merck | 354400 | |
| anti-rabbit immunglobulin-antibodies from goat, conjugated with alkaline phosphatase | Sigma-Aldrich | A9919 | |
| Glycine | Applichem | A1377 | |
| Zn(II)-acetate | Applichem | A4324 | |
| NaOH | Applichem | A1551 | |
| Alkaline phosphatase substrate tablet (5 mg) | Sigma-Aldrich | S0942 | |
| Costar half-area microtiter plate | Thermo Scientific | Corning 3690 | |
| micro reaction tubes | Eppendorf | 30120086 | |
| Microplate ELISA reader | BioTek | Synergy HT |
References
- Klotz, I. M. The application of the law of mass action to binding by proteins; interactions with calcium. Arch Biochem. 9, 109-117 (1946).
- Klotz, I. M. Ligand-receptor complexes: origin and development of the concept. J Biol Chem. 279 (1), 1-12 (2004).
- Bisswanger, H. Ch. 1: Multiple Equilibria, Principles and Derivations. Enzyme Kinetics: Principles and Methods. , 1-26 (2017).
- Shimura, K., Kasai, K. Affinity gel titration: quantitative analysis of the binding equilibrium between immobilized protein and free ligand by a continuous titration procedure. Anal Biochem. 149 (2), 369-378 (1985).
- Gong, M., Nikcevic, I., Wehmeyer, K. R., Limbach, P. A., Heineman, W. R. Protein-aptamer binding studies using microchip affinity capillary electrophoresis. Electrophoresis. 29 (7), 1415-1422 (2008).
- Hanes, M. S., Ratcliff, K., Marqusee, S., Handel, T. M. Protein-protein binding affinities by pulse proteolysis: application to TEM-1/BLIP protein complexes. Protein Sci. 19 (10), 1996-2000 (2010).
- Latour, R. A. The Langmuir isotherm: a commonly applied but misleading approach for the analysis of protein adsorption behavior. J Biomed Mater Res A. 103 (3), 949-958 (2015).
- Stockell, A. The binding of diphosphopyridine nucleotide by yeast glyceraldehyde-3-phosphate dehydrogenase. J Biol Chem. 234 (5), 1286-1292 (1959).
- Heyn, M. P., Weischet, W. O. Circular dichroism and fluorescence studies on the binding of ligands to the α subunit of tryptophan synthase. Biochem. 14 (13), 2962-2968 (1975).
- Campbell, I. D., Humphries, M. J. Integrin structure, activation, and interactions. Cold Spring Harb Perspect Biol. 3 (3), (2011).
- Zeltz, C., Gullberg, D. The integrin-collagen connection–a glue for tissue repair?. J Cell Sci. 129 (4), 653-664 (2016).
- Kanchanawong, P., et al. Nanoscale architecture of integrin-based cell adhesions. Nature. 468 (7323), 580-584 (2010).
- Luo, B. H., Carman, C. V., Springer, T. A. Structural basis of integrin regulation and signaling. Annu Rev Immunol. 25, 619-647 (2007).
- Luo, B. H., Springer, T. A. Integrin structures and conformational signaling. Curr Opin Cell Biol. 18 (5), 579-586 (2006).
- Ricard-Blum, S. The collagen family. Cold Spring Harb Perspect Biol. 3 (1), a004978 (2011).
- Eble, J. A., Tuckwell, D. S. The α2β1 integrin inhibitor rhodocetin binds to the A-domain of the integrin α2 subunit proximal to the collagen-binding site. Biochem J. 376 (Pt 1), 77-85 (2003).
- Eble, J. A., et al. The α2β1 integrin-specific antagonist rhodocetin is a cruciform, heterotetrameric molecule. FASEB J. 23 (9), 2917-2927 (2009).
- Eble, J. A., et al. Dramatic and concerted conformational changes enable rhodocetin to block α2β1 integrin selectively. PLoS Biol. 15 (7), e2001492 (2017).
- Bracht, T., Figueiredo de Rezende, F., Stetefeld, J., Sorokin, L. M., Eble, J. A. Monoclonal antibodies reveal the alteration of the rhodocetin structure upon α2β1 integrin binding. Biochem J. 440 (1), 1-11 (2011).
- Harlow, E., Lane, D. Chapter 5: Immunizations. Antibodies, a laboratory manual. 5, 53-138 (1988).
- Lu, D. Analyzing interactions between SSB and proteins by the use of fluorescence anisotropy. Methods Mol Biol. 922, 155-159 (2012).
- Fielding, L. NMR methods for the determination of protein-ligand dissociation constants. Curr Top Med Chem. 3 (1), 39-53 (2003).
- Leavitt, S., Freire, E. Direct measurement of protein binding energetics by isothermal titration calorimetry. Curr Opin Struct Biol. 11 (5), 560-566 (2001).
- McDonnell, J. M. Surface plasmon resonance: towards an understanding of the mechanisms of biological molecular recognition. Curr Opin Chem Biol. 5 (5), 572-577 (2001).
- Rich, R. L., Myszka, D. G. Advances in surface plasmon resonance biosensor analysis. Curr Opin Biotechnol. 11 (1), 54-61 (2000).
- Pesquero, N. C., et al. Real-time monitoring and kinetic parameter estimation of the affinity interaction of jArtinM and rArtinM with peroxidase glycoprotein by the electrogravimetric technique. Biosens Bioelectron. 26 (1), 36-42 (2010).
- Scheuermann, T. H., Padrick, S. B., Gardner, K. H., Brautigam, C. A. On the acquisition and analysis of microscale thermophoresis data. Anal Biochem. 496, 79-93 (2016).

