تحديد الربط البديل والبولي أدينيل في بيانات RNA-seq
Summary
يعمل الربط البديل (AS) والبولي أدينيل البديل (APA) على توسيع تنوع الأشكال المتماثلة ومنتجاتها. هنا ، نصف بروتوكولات المعلوماتية الحيوية لتحليل مقايسات تسلسل RNA-seq السائبة و 3 ‘للكشف عن وتصور AS و APA المتغيرة عبر الظروف التجريبية.
Abstract
بالإضافة إلى التحليل النموذجي ل RNA-Seq لقياس التعبير الجيني التفاضلي (DGE) عبر الظروف التجريبية / البيولوجية ، يمكن أيضا استخدام بيانات RNA-seq لاستكشاف آليات تنظيمية معقدة أخرى على مستوى exon. يلعب الربط البديل و polyadenylation دورا حاسما في التنوع الوظيفي للجين من خلال توليد أشكال متساوية مختلفة لتنظيم التعبير الجيني على مستوى ما بعد النسخ ، ويمكن أن يؤدي قصر التحليلات على مستوى الجينات بالكامل إلى تفويت هذه الطبقة التنظيمية المهمة. هنا ، نوضح تحليلات مفصلة خطوة بخطوة لتحديد وتصور استخدام موقع exon و polyadenylation التفاضلي عبر الظروف ، باستخدام Bioconductor والحزم والوظائف الأخرى ، بما في ذلك DEXSeq و diffSplice من حزمة Limma و rMATS.
Introduction
تم استخدام RNA-seq على نطاق واسع على مر السنين عادة لتقدير التعبير الجيني التفاضلي واكتشاف الجينات1. بالإضافة إلى ذلك ، يمكن استخدامه أيضا لتقدير الاستخدام المتغير لمستوى إكسون بسبب تعبير الجينات عن أشكال متساوية مختلفة ، وبالتالي المساهمة في فهم أفضل لتنظيم الجينات على مستوى ما بعد النسخ. تولد غالبية الجينات حقيقية النواة أشكالا متساوية مختلفة عن طريق الربط البديل (AS) لزيادة تنوع تعبير mRNA. يمكن تقسيم أحداث AS إلى أنماط مختلفة: تخطي exons كاملة (SE) حيث تتم إزالة exon (“كاسيت”) تماما من النص جنبا إلى جنب مع الإنترونات المرافقة له ؛ اختيار موقع لصق بديل (مانح) 5 ‘(A5SS) واختيار موقع لصق بديل 3 ‘(متقبل) (A3SS) عند وجود موقعين أو أكثر من مواقع لصق على طرفي إكسون ؛ الاحتفاظ بالإنترونات (RI) عند الاحتفاظ ب intron ضمن نسخة mRNA الناضجة والاستبعاد المتبادل لاستخدام exon (MXE) حيث يمكن الاحتفاظ بواحد فقط من الإكسونات المتاحين في وقت 2,3. يلعب polyadenylation البديل (APA) أيضا دورا مهما في تنظيم التعبير الجيني باستخدام مواقع poly البديلة (A) لتوليد أشكال متماثلة متعددة من mRNA من نسخة واحدة4. تقع معظم مواقع polyadenylation (pAs) في المنطقة غير المترجمة 3 ‘(3’ UTRs) ، مما يولد أشكالا متماثلة من mRNA بأطوال UTR متنوعة 3 بوصات. نظرا لأن 3 ‘UTR هو المحور المركزي للتعرف على العناصر التنظيمية ، يمكن أن تؤثر أطوال UTR المختلفة 3 ‘على توطين mRNA واستقراره وترجمته5. هناك فئة من 3 ‘مقايسات تسلسل نهاية محسنة للكشف عن APA التي تختلف في تفاصيل البروتوكول6. تم تصميم خط الأنابيب الموصوف هنا ل PolyA-seq ، ولكن يمكن تكييفه مع البروتوكولات الأخرى كما هو موضح.
في هذه الدراسة ، نقدم مجموعة من طرق تحليل exon التفاضلية 7,8 (الشكل 1) ، والتي يمكن تقسيمها إلى فئتين عريضتين: القائمة على exon (DEXSeq9 ، diffSplice 10) والقائمة على الحدث (تكرار التحليل متعدد المتغيرات لربط النص (rMATS)11). تقارن الطرق القائمة على الإكسون تغير الطي عبر ظروف الإكسونات الفردية ، مقابل مقياس للتغير الكلي في طية الجينات لاستدعاء استخدام إكسون المعبر عنه بشكل تفاضلي ، ومن ذلك تحسب مقياسا على مستوى الجينات لنشاط AS. تستخدم الطرق المستندة إلى الأحداث قراءات تقاطع exon-intron-spanning لاكتشاف وتصنيف أحداث الربط المحددة مثل تخطي exon أو الاحتفاظ بالإنترونات ، وتمييز أنواع AS هذه في الإخراج3. وبالتالي ، توفر هذه الطرق وجهات نظر تكميلية لتحليل كامل لمعيار المحاسبة12,13. اخترنا DEXSeq (استنادا إلى حزمة DESeq214 DGE) و diffSplice (استنادا إلى حزمة Limma10 DGE) للدراسة لأنهما من بين الحزم الأكثر استخداما لتحليل الربط التفاضلي. تم اختيار rMATS كطريقة شائعة للتحليل القائم على الأحداث. طريقة أخرى شائعة قائمة على الأحداث هي MISO (خليط من الأشكال المتساوية)1. بالنسبة إلى APA ، نقوم بتكييف النهج القائم على exon.
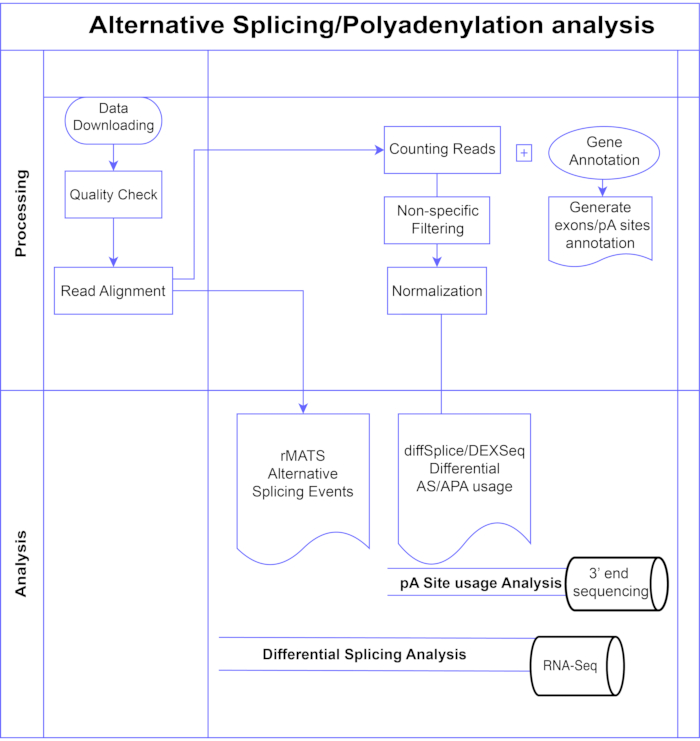
الشكل 1. خط أنابيب التحليل. مخطط انسيابي للخطوات المستخدمة في التحليل. تشمل الخطوات: الحصول على البيانات ، وإجراء فحوصات الجودة ومحاذاة القراءة متبوعة بحساب القراءات باستخدام التعليقات التوضيحية لمواقع exons و introns و pA المعروفة ، والتصفية لإزالة الأعداد المنخفضة والتطبيع. تم تحليل بيانات PolyA-seq لمواقع pA البديلة باستخدام طرق diffSplice / DEXSeq ، وتم تحليل RNA-Seq السائب للربط البديل على مستوى exon باستخدام طرق diffSplice / DEXseq ، وتم تحليل أحداث AS باستخدام rMATS. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
تم الحصول على بيانات RNA-seq المستخدمة في هذا المسح من التعبير الجيني الجامع (GEO) (GSE138691)15. استخدمنا بيانات RNA-seq للفأر من هذه الدراسة مع مجموعتين من الحالات: النوع البري (WT) والضربة القاضية من النوع 1 الشبيه بالعضلات (Mbnl1 KO) مع ثلاث نسخ متماثلة لكل منهما. لإثبات تحليل استخدام موقع polyadenylation التفاضلي ، حصلنا على بيانات الخلايا الليفية الجنينية للفأر (MEFs) PolyA-seq (GEO Accession GSE60487)16. تحتوي البيانات على أربع مجموعات شروط: النوع البري (WT) ، النوع 1 / النوع 2 الشبيه بالعضلات بالضربة القاضية المزدوجة (Mbnl1/2 DKO) ، Mbnl 1/2 DKO مع ضربة قاضية Mbnl3 (KD) و Mbnl1/2 DKO مع التحكم Mbnl3 (Ctrl). تتكون كل مجموعة شرط من نسختين متماثلتين.
| انضمام GEO | رقم تشغيل SRA | اسم العينة | شرط | تكرار | نسيج | التسلسل | طول القراءة | |
| RNA-Seq | جي إس إم 4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 بالضربة القاضية | ممثل 1 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس |
| جي إس إم 4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 بالضربة القاضية | ممثل 2 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 بالضربة القاضية | مندوب 3 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116221 | SRR10261604 | WT_Thymus_1 | نوع البرية | ممثل 1 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116222 | SRR10261605 | WT_Thymus_2 | نوع البرية | ممثل 2 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| جي إس إم 4116223 | SRR10261606 | WT_Thymus_3 | نوع البرية | مندوب 3 | الغدة الصعترية | نهاية مزدوجة | 100 نقطة أساس | |
| 3P-Seq | جي إس إم 1480973 | ريال1553129 | WT_1 | النوع البري (WT) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس |
| جي إس إم 1480974 | ريال1553130 | WT_2 | النوع البري (WT) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 ضربة قاضية مزدوجة (DKO) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 ضربة قاضية مزدوجة (DKO) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480977 | ريال1553133 | DKOsiRNA_1 | Mbnl 1/2 ضربة قاضية مزدوجة مع Mbnl 3 siRNA (دينار كويتي) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480978 | ريال1553134 | DKOsiRNA_2 | Mbnl 1/2 ضربة قاضية مزدوجة مع Mbnl 3 siRNA (دينار كويتي) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 36 نقطة أساس | |
| جي إس إم 1480979 | ريال1553135 | DKONTsiRNA_1 | Mbnl 1/2 ضربة قاضية مزدوجة مع siRNA غير مستهدف (Ctrl) | ممثل 1 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس | |
| جي إس إم 1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 ضربة قاضية مزدوجة مع siRNA غير مستهدف (Ctrl) | ممثل 2 | الخلايا الليفية الجنينية للفأر (MEFs) | نهاية واحدة | 40 نقطة أساس |
الجدول 1. ملخص مجموعات بيانات RNA-Seq و PolyA-seq المستخدمة في التحليل.
Protocol
Representative Results
Discussion
في هذه الدراسة ، قمنا بتقييم النهج القائمة على exon والقائمة على الأحداث للكشف عن AS و APA في بيانات تسلسل RNA-Seq السائبة و 3 ‘التسلسل النهائي. تنتج مناهج AS القائمة على exon كلا من قائمة الإكسونات المعبر عنها بشكل تفاضلي وترتيب على مستوى الجينات مرتبة حسب الأهمية الإحصائية لنشاط الربط التفاضلي على م…
Disclosures
The authors have nothing to disclose.
Acknowledgements
تم دعم هذه الدراسة من قبل زمالة المستقبل لمجلس البحوث الأسترالي (ARC) (FT16010043) ومخطط ANU Futures.
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

