Identificazione di splicing alternativo e poliadenilazione in dati di RNA-seq
Summary
Lo splicing alternativo (AS) e la poliadenilazione alternativa (APA) espandono la diversità delle isoforme di trascrizione e dei loro prodotti. Qui, descriviamo i protocolli bioinformatici per analizzare i saggi di sequenziamento di massa dell’RNA-seq e 3′ per rilevare e visualizzare AS e APA che variano in base alle condizioni sperimentali.
Abstract
Oltre all’analisi tipica dell’RNA-Seq per misurare l’espressione genica differenziale (DGE) in condizioni sperimentali/biologiche, i dati di RNA-seq possono anche essere utilizzati per esplorare altri complessi meccanismi regolatori a livello di esone. Lo splicing alternativo e la poliadenilazione svolgono un ruolo cruciale nella diversità funzionale di un gene generando diverse isoforme per regolare l’espressione genica a livello post-trascrizionale e limitando le analisi all’intero livello genico può mancare questo importante strato regolatore. Qui, dimostriamo analisi dettagliate passo dopo passo per l’identificazione e la visualizzazione dell’utilizzo differenziale del sito di esone e poliadenilazione in tutte le condizioni, utilizzando Bioconductor e altri pacchetti e funzioni, tra cui DEXSeq, diffSplice dal pacchetto Limma e rMATS.
Introduction
L’RNA-seq è stato ampiamente utilizzato nel corso degli anni, tipicamente per stimare l’espressione genica differenziale e la scopertagenica 1. Inoltre, può anche essere utilizzato per stimare l’uso variabile del livello di esone a causa del gene che esprime diverse isoforme, contribuendo così a una migliore comprensione della regolazione genica a livello post-trascrizionale. La maggior parte dei geni eucariotici genera diverse isoforme mediante splicing alternativo (AS) per aumentare la diversità dell’espressione dell’mRNA. Gli eventi AS possono essere suddivisi in diversi modelli: salto di esoni completi (SE) in cui un esone (“cassetta”) viene completamente rimosso dalla trascrizione insieme ai suoi introni laterali; selezione alternativa (donatore) del sito di giunzione 5′ (A5SS) e selezione alternativa del sito di giunzione 3′ (accettore) (A3SS) quando due o più siti di giunzione sono presenti su entrambe le estremità di un esone; ritenzione degli introni (RI) quando un introne viene trattenuto all’interno del trascritto dell’mRNA maturo e mutua esclusione dell’uso dell’esone (MXE) in cui solo uno dei due esoni disponibili può essere trattenuto alla volta 2,3. La poliadenilazione alternativa (APA) svolge anche un ruolo importante nella regolazione dell’espressione genica utilizzando siti di poli alternativi (A) per generare più isoforme di mRNA da una singola trascrizione4. La maggior parte dei siti di poliadenilazione (pAs) si trovano nella regione 3′ non tradotta (3′ UTR), generando isoforme di mRNA con diverse lunghezze UTR 3′. Poiché l’UTR 3′ è l’hub centrale per il riconoscimento degli elementi regolatori, diverse lunghezze UTR 3′ possono influenzare la localizzazione, la stabilità e la traduzione dell’mRNA5. Esistono saggi di sequenziamento finale di classe 3′ ottimizzati per rilevare APA che differiscono nei dettagli del protocollo6. La pipeline qui descritta è progettata per PolyA-seq, ma può essere adattata per altri protocolli come descritto.
In questo studio, presentiamo una pipeline di metodi di analisi differenziale degli esoni7,8 (Figura 1), che possono essere suddivisi in due grandi categorie: basati sull’esone (DEXSeq9, diffSplice 10) e basati sugli eventi (replicate Multivariate Analysis of Transcript Splicing (rMATS)11). I metodi basati sull’esone confrontano il cambiamento di piega tra le condizioni dei singoli esoni, contro una misura del cambiamento complessivo della piega genica per chiamare l’uso differenziale dell’esone, e da ciò calcolare una misura a livello genetico dell’attività AS. I metodi basati su eventi utilizzano letture di giunzione esone-introne per rilevare e classificare eventi di splicing specifici come il salto dell’esone o la ritenzione di introni e distinguere questi tipi di AS nell’output3. Pertanto, questi metodi forniscono punti di vista complementari per un’analisi completa di AS12,13. Abbiamo selezionato DEXSeq (basato sul pacchetto DESeq214 DGE) e diffSplice (basato sul pacchetto Limma10 DGE) per lo studio in quanto sono tra i pacchetti più utilizzati per l’analisi di splicing differenziale. rMATS è stato scelto come metodo popolare per l’analisi basata sugli eventi. Un altro metodo popolare basato su eventi è MISO (Mixture of Isoforms)1. Per l’APA adattiamo l’approccio basato sull’esone.
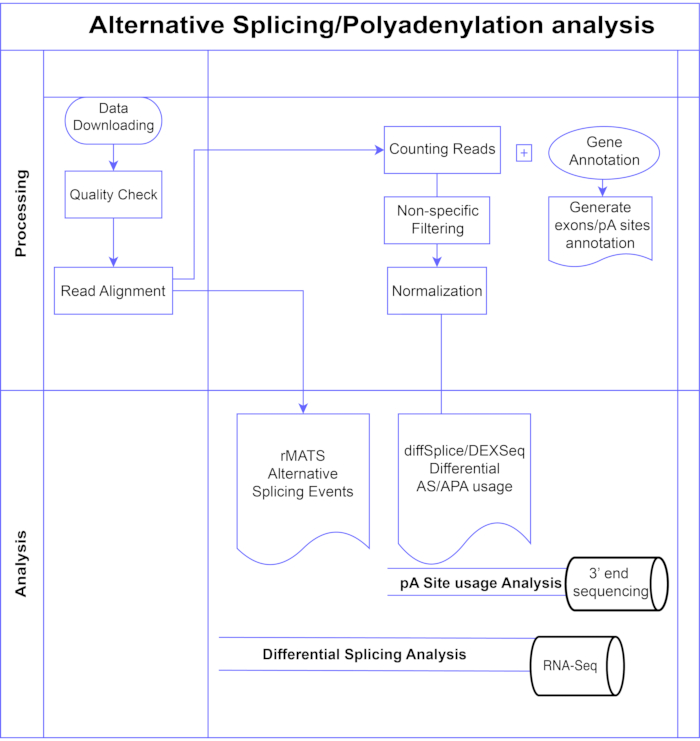
Figura 1. Pipeline di analisi. Diagramma di flusso dei passaggi utilizzati nell’analisi. I passaggi includono: ottenere i dati, eseguire controlli di qualità e allineamento delle letture seguito dal conteggio delle letture utilizzando annotazioni per esoni, introni e siti pA noti, filtraggio per rimuovere conteggi bassi e normalizzazione. I dati di PolyA-seq sono stati analizzati per siti pA alternativi utilizzando metodi diffSplice/DEXSeq, RNA-Seq di massa sono stati analizzati per lo splicing alternativo a livello di esone con metodi diffSplice/DEXseq e gli eventi AS analizzati con rMATS. Fare clic qui per visualizzare una versione ingrandita di questa figura.
I dati RNA-seq utilizzati in questa indagine sono stati acquisiti da Gene Expression Omnibus (GEO) (GSE138691)15. Abbiamo utilizzato i dati RNA-seq di topo di questo studio con due gruppi di condizioni: wild-type (WT) e Muscleblind-like type 1 knockout (Mbnl1 KO) con tre repliche ciascuno. Per dimostrare l’analisi differenziale dell’utilizzo del sito di poliadenilazione, abbiamo ottenuto dati PolyA-seq di fibroblasti embrionali di topo (MEF) (GEO Accession GSE60487)16. I dati hanno quattro gruppi di condizioni: Wild-type (WT), Muscleblind-like type1/type 2 double knockout (Mbnl1/2 DKO), Mbnl 1/2 DKO con Mbnl3 knockdown (KD) e Mbnl1/2 DKO con controllo Mbnl3 (Ctrl). Ogni gruppo di condizioni è costituito da due repliche.
| Adesione GEO | Numero di esecuzione SRA | Nome del campione | Condizione | Replicare | Fazzoletto | Sequenziamento | Lunghezza di lettura | |
| RNA-Seq | GSM4116218 | SRR10261601 | Mbnl1KO_Thymus_1 | Mbnl1 knockout | Rappresentante 1 | Timo | Estremità accoppiata | 100 pb |
| GSM4116219 | SRR10261602 | Mbnl1KO_Thymus_2 | Mbnl1 knockout | Rappresentante 2 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116220 | SRR10261603 | Mbnl1KO_Thymus_3 | Mbnl1 knockout | Rappresentante 3 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116221 | SRR10261604 | WT_Thymus_1 | Tipo selvaggio | Rappresentante 1 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116222 | SRR10261605 | WT_Thymus_2 | Tipo selvaggio | Rappresentante 2 | Timo | Estremità accoppiata | 100 pb | |
| GSM4116223 | SRR10261606 | WT_Thymus_3 | Tipo selvaggio | Rappresentante 3 | Timo | Estremità accoppiata | 100 pb | |
| 3P-Seq | GSM1480973 | SRR1553129 | WT_1 | Tipo selvatico (WT) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb |
| GSM1480974 | SRR1553130 | WT_2 | Tipo selvatico (WT) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480975 | SRR1553131 | DKO_1 | Mbnl 1/2 doppio knockout (DKO) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480976 | SRR1553132 | DKO_2 | Mbnl 1/2 doppio knockout (DKO) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480977 | SRR1553133 | DKOsiRNA_1 | Mbnl 1/2 doppio knockout con Mbnl 3 siRNA (KD) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480978 | SRR1553134 | DKOsiRNA_2 | Mbnl 1/2 doppio knockout con Mbnl 3 siRNA (KD) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 36 pb | |
| GSM1480979 | SRR1553135 | DKONTsiRNA_1 | Mbnl 1/2 doppio knockout con siRNA non mirato (Ctrl) | Rappresentante 1 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb | |
| GSM1480980 | SRR1553136 | DKONTsiRNA_2 | Mbnl 1/2 doppio knockout con siRNA non mirato (Ctrl) | Rappresentante 2 | Fibroblasti embrionali di topo (MEF) | Estremità singola | 40 pb |
Tabella 1. Riepilogo dei set di dati RNA-Seq e PolyA-seq utilizzati per l’analisi.
Protocol
Representative Results
Discussion
In questo studio, abbiamo valutato approcci basati su esoni e basati su eventi per rilevare AS e APA in massa RNA-Seq e 3′ dati di sequenziamento finale. Gli approcci AS basati sugli esoni producono sia un elenco di esoni differenzialmente espressi sia una classificazione a livello genico ordinata in base alla significatività statistica dell’attività complessiva di splicing differenziale a livello genetico (Tabelle 1-2, 4-5). Per il pacchetto diffSplice, l’uso differenziale è determinato adattando mod…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo studio è stato supportato da una Future Fellowship dell’Australian Research Council (ARC) (FT16010043) e da ANU Futures Scheme.
References
- Katz, Y., Wang, E. T., Airoldi, E. M., Burge, C. B. Analysis and design of RNA sequencing experiments for identifying isoform regulation. Nature Methods. 7 (12), 1009-1015 (2010).
- Wang, Y., et al. Mechanism of alternative splicing and its regulation. Biomedical Reports. 3 (2), 152-158 (2015).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in Bioinformatics. 21 (6), 2052-2065 (2020).
- Movassat, M., et al. Coupling between alternative polyadenylation and alternative splicing is limited to terminal introns. RNA Biology. 13 (7), 646-655 (2016).
- Tian, B., Manley, J. L. Alternative polyadenylation of mRNA precursors. Nature Reviews Molecular Cell Biology. 18 (1), 18-30 (2017).
- Herrmann, C. J., et al. PolyASite 2.0: a consolidated atlas of polyadenylation sites from 3′ end sequencing. Nucleic Acids Research. 48 (1), 174-179 (2020).
- Liu, R., Loraine, A. E., Dickerson, J. A. Comparisons of computational methods for differential alternative splicing detection using RNA-seq in plant systems. BMC Bioinformatics. 15 (1), 364 (2014).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biology. 17 (1), 13 (2016).
- Anders, S., Reyes, A., Huber, W. Detecting differential usage of exons from RNA-seq data. Genome Research. 22 (10), 2008-2017 (2012).
- Ritchie, M. E., et al. limma powers differential expression analyses for RNA-sequencing and microarray studies. Nucleic Acids Research. 43 (7), 47 (2014).
- Shen, S., et al. rMATS: Robust and flexible detection of differential alternative splicing from replicate RNA-Seq data. Proceedings of the National Academy of Sciences. 111 (51), 5593-5601 (2014).
- Mehmood, A., et al. Systematic evaluation of differential splicing tools for RNA-seq studies. Briefings in bioinformatics. 21 (6), 2052-2065 (2020).
- Kanitz, A., et al. Comparative assessment of methods for the computational inference of transcript isoform abundance from RNA-seq data. Genome biology. 16 (1), 1-26 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15 (12), 550 (2014).
- Sznajder, L. J., et al. Loss of MBNL1 induces RNA misprocessing in the thymus and peripheral blood. Nature Communications. 11, 1-11 (2020).
- Batra, R., et al. Loss of MBNL leads to disruption of developmentally regulated alternative polyadenylation in RNA-mediated disease. Molecular Cell. 56 (2), 311-322 (2014).
- Leinonen, R., Sugawara, H., Shumway, M., et al. The sequence read archive. Nucleic acids research. 39, 19-21 (2010).
- Tange, O. . GNU parallel-the command-line power tool. 36, 42-47 (2011).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet journal. 17 (1), 10-12 (2011).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- Dobin, A., et al. STAR: ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Robinson, M. D., McCarthy, D. J., Smyth, G. K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics. 26 (1), 139-140 (2010).
- Robinson, M. D., Oshlack, A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biology. 11 (3), 25 (2010).
- Veiga, D. F. T. maser: Mapping Alternative Splicing Events to pRoteins. R package version 1.4.0. , (2019).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (13), 25 (2009).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics. 26 (6), 841-842 (2010).
- Ramírez, F., Dündar, F., Diehl, S., Grüning, B. A., Manke, T. deepTools: a flexible platform for exploring deep-sequencing data. Nucleic acids research. 42 (1), 187-191 (2014).
- Merino, G. A., Conesa, A., Fernández, E. A. A benchmarking of workflows for detecting differential splicing and differential expression at isoform level in human RNA-seq studies. Briefings in bioinformatics. 20 (2), 471-481 (2019).
- Chhangawala, S., Rudy, G., Mason, C. E., Rosenfeld, J. A. The impact of read length on quantification of differentially expressed genes and splice junction detection. Genome biology. 16 (1), 1-10 (2015).
- Conesa, A., et al. A survey of best practices for RNA-seq data analysis. Genome Biol. 17, 13 (2016).
- Trapnell, C., et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks. Nat Protoc. 7 (3), 562-578 (2012).
- Li, B., Dewey, C. N. RSEM: accurate transcript quantification from RNA-Seq data with or without a reference genome. BMC Bioinformatics. 12, 323 (2011).
- Bray, N. L., Pimentel, H., Melsted, P., Pachter, L. Near-optimal probabilistic RNA-seq quantification. Nat Biotechnol. 34 (5), 525-527 (2016).

