Respirometria ad alta risoluzione per valutare la bioenergetica nelle cellule e nei tessuti utilizzando respirometri a camera e a piastre
Summary
La valutazione della fosforilazione ossidativa mediante respirometri ad alta risoluzione è diventata parte integrante dell’analisi funzionale dei mitocondri e del metabolismo energetico cellulare. Qui, presentiamo protocolli per l’analisi del metabolismo energetico cellulare utilizzando respirometri ad alta risoluzione basati su camera e micropiastre e discutiamo i principali vantaggi di ciascun dispositivo.
Abstract
La respirometria ad alta risoluzione (HRR) consente di monitorare la fosforilazione ossidativa in tempo reale per l’analisi dei singoli stati energetici cellulari e la valutazione dei complessi respiratori utilizzando protocolli diversificati di titolazione substrato-disaccoppiatore-inibitore (SUIT). Qui viene dimostrato l’uso di due dispositivi di respirometria ad alta risoluzione e viene presentata una raccolta di base di protocolli applicabili per l’analisi di cellule in coltura, fibre muscolari scheletriche e cardiache e tessuti molli come il cervello e il fegato. I protocolli per cellule e tessuti in coltura sono forniti per un respirometro a camera e cellule coltivate per un respirometro basato su micropiastre, entrambi comprendenti protocolli di respirazione standard. A scopo comparativo, le cellule HEK293 ingegnerizzate con CRISPR carenti di traduzione mitocondriale che causano una carenza multipla del sistema respiratorio vengono utilizzate con entrambi i dispositivi per dimostrare difetti cellulari nella respirazione. Entrambi i respirometri consentono una misurazione completa della respirazione cellulare con i rispettivi meriti tecnici e l’idoneità a seconda della domanda di ricerca e del modello in studio.
Introduction
I mitocondri soddisfano la fornitura chiave di energia e sono un organello compartimentato che contribuisce a processi bioenergetici e metabolici cellulari essenziali come l’anabolismo di nucleotidi, lipidi e amminoacidi, biogenesi del cluster ferro-zolfo e sono implicati nella segnalazione come la morte cellulare controllata 1,2,3 . La bioenergetica mitocondriale attraverso la fosforilazione ossidativa contribuisce a quasi tutti i processi cellulari all’interno della cellula e, di conseguenza, le disfunzioni mitocondriali di origine primaria o secondaria sono associate a un ampio spettro di condizioni di malattia 4,5. La disfunzione mitocondriale comporta non solo alterazioni della struttura o della densità mitocondriale, ma anche della qualità e della regolazione dell’apparato respiratorio6. Questo elemento qualitativo comprende il controllo del substrato, le caratteristiche di accoppiamento, le modifiche post-traduzionali, la dinamica delle cristae e i supercomplessi respiratori 7,8. Pertanto, un’analisi accurata della bioenergetica mitocondriale per approcci sperimentali e diagnostici per valutare il metabolismo energetico della cellula è importante per la salute e la malattia.
La fosforilazione ossidativa mitocondriale (OXPHOS) è una sequenza di reazioni all’interno del sistema respiratorio o del sistema di trasferimento di elettroni (ETS) per la generazione di energia cellulare attraverso l’adenosina trifosfato (ATP)9. La fase multienzimatica per sfruttare l’energia dal flusso di elettroni attraverso i complessi I e II al complesso IV genera un gradiente protonico elettrochimico attraverso la membrana mitocondriale interna, successivamente utilizzato per la fosforilazione dell’adenosina difosfato (ADP) all’ATP tramite il complesso V (F1FO ATP sintasi) (Figura 1A).
In primo luogo, i vettori di due elettroni vengono generati durante il ciclo tricarbossilico (TCA), la glicolisi e l’ossidazione del piruvato: nicotinamide adenina dinucleotide (NADH) e diidroflavina adenina dinucleotide (FADH2). Il NADH viene ossidato nel complesso I (NADH deidrogenasi), durante il quale due elettroni vengono trasferiti al coenzima Q (il chinone viene ridotto a chinolo), mentre i protoni vengono pompati nello spazio intermembrana (IMS). In secondo luogo, il complesso II (Succinato deidrogenasi) ossida FADH2 e alimenta gli elettroni al coenzima Q senza pompare protoni. In terzo luogo, nel complesso III (citocromo c ossidoreduttasi), gli elettroni del coenzima Q vengono trasferiti al citocromo c mentre i protoni vengono pompati nell’IMS. In quarto luogo, il citocromo c trasferisce gli elettroni al complesso IV (citocromo c ossidasi), il complesso finale per pompare protoni e dove l’ossigeno funziona come un accettore di elettroni per assimilare i protoni, formando infine acqua. È questo ossigeno che i mitocondri consumano che può essere misurato da un ossigrafo. Infine, i protoni generati dal complesso I, dal complesso III e dal complesso IV vengono utilizzati per ruotare il complesso V, generando così ATP9.
È importante sottolineare che il trasferimento di elettroni avviene non solo in modo lineare, altrimenti indicato come la catena di trasporto degli elettroni. Invece, gli elettroni possono essere trasferiti al pool di coenzima Q attraverso più vie respiratorie e facilitare il flusso di elettroni convergenti. I substrati nadH e il succinato, ad esempio, possono entrare rispettivamente attraverso il complesso I e il complesso II. Gli elettroni dell’ossidazione degli acidi grassi possono essere donati tramite il complesso flavoproteico che trasferisce elettroni. In effetti, un’analisi completa di OXPHOS richiede un approccio olistico con substrati di carburante appropriati (Figura 1A).
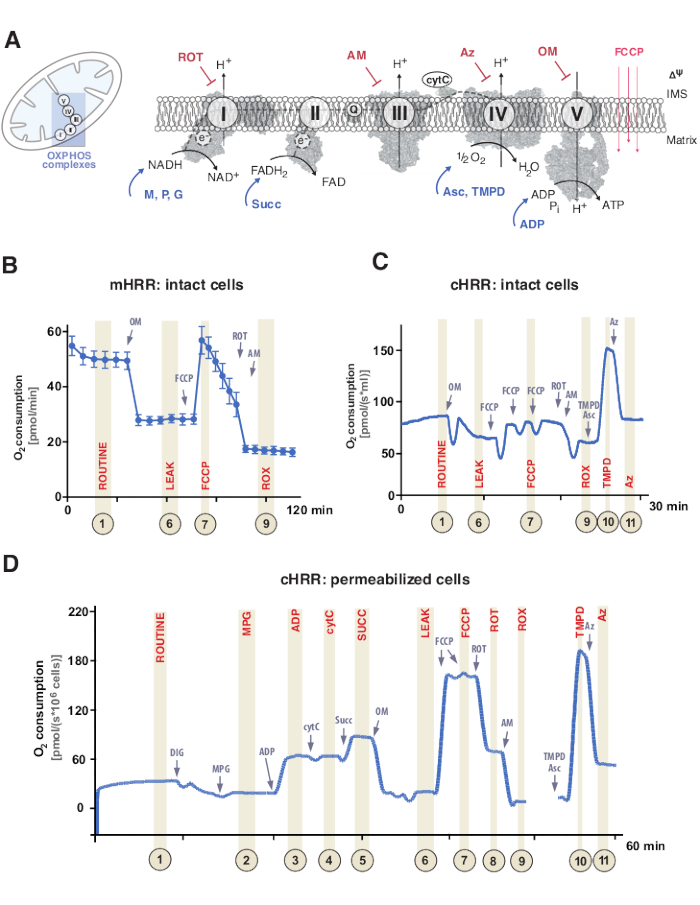
Figura 1: Fosforilazione ossidativa mitocondriale e protocolli specifici di substrato e inibitori. (A) Mitocondrio e schema del sistema di trasferimento di elettroni (CI-CIV) e F1F0 ATP sintasi mitocondriale (CV). Tutte le strutture provengono da PDB. Le figure raffigurano solo substrati e inibitori descritti in questo studio). (B) Traccia campione di flusso di ossigeno in celle HEK293 intatte utilizzando il protocollo standard in un dispositivo mHRR. (C) Traccia campione di flusso di ossigeno in celle HEK293 intatte utilizzando il protocollo standard in un dispositivo cHRR. (D) Traccia campione del flusso di ossigeno nei fibroblasti umani permeabilizzati da un donatore sano con il rispettivo protocollo SUIT. Abbreviazioni: 1 = Respirazione di routine delle cellule intatte; 2 = Stato 2; 3 = Stato 3(I); 4 = Stato 3(I) con cytC; 5 = Stato 3 (I+II); 6 = Perdita(OM); 7 = capacità ETS; 8 = S(ROT); 9 = ROX; 10 = TMPD; 11 = Az. ROT = Rotenone, AM = Antimicina, ATP = Adenosina trifosfato, Az = Azide, OM = Oligomicina, FCCP = cianuro di carbonile p-trifluoro-metossifenil-idrazzone; Asc = Ascorbato, TMPD = N,N,N′,N′-tetrametil-p-fenilendiammina, Succ = Succinato, M = Malato, P = Piruvato, ADP = Adenosina difosfato, NAD = Nicotinamide adenina dinucleotide, IMS = Spazio intermembrana, FAD = Flavin adenina dinucleotide. Fare clic qui per visualizzare una versione più grande di questa figura.
L’analisi della capacità di OXPHOS mitocondriale utilizzando HRR è diventata un metodo biochimico strumentale di valore diagnostico non solo per i difetti mitocondriali primari10,11 ma estendendosi a tutti gli altri regni della biologia come il cancro e l’invecchiamento12. HRR consente la determinazione della respirazione cellulare mediante l’analisi della capacità mitocondriale oxphos, che riflette direttamente il deficit del complesso respiratorio mitocondriale individuale o combinato, e indirettamente è associato a disfunzione cellulare e metabolismo energetico alterato9. Metodologicamente, le misurazioni della respirazione vengono eseguite utilizzando cellule, tessuti o mitocondri isolati 11,13,14, con materiale congelato solo parzialmente adatto 15,16. Il tessuto congelato ha dimostrato di avere un ETS intatto con stabilità supercomplessa mantenuta15. Pertanto, a differenza dei tradizionali intermedi TCA, i rispettivi substrati vengono immessi direttamente nell’ETS. Tuttavia, l’accoppiamento tra la sintesi di ETS e ATP viene perso poiché l’integrità della membrana è compromessa da danni da congelamento (formazione di cristalli di ghiaccio).
Gli esperimenti di respirazione si svolgono normalmente a una temperatura fisiologica di 37 °C per gli endotermi in cellule o tessuti non permeabilizzati o permeabilizzati. Mentre il primo considera il contesto metabolico citosolico, il secondo fornisce il contributo energetico dei singoli complessi OXPHOS e dell’ATPasi attraverso l’aggiunta di specifici substrati (e inibitori). La sequenza e la variazione di substrati e inibitori hanno portato allo sviluppo di una vasta gamma di protocolli SUIT17 e saggi18 per affrontare varie questioni scientifiche sulla funzione di OXPHOS (esaminate al punto12). Il protocollo di base della respirazione cellulare valuta quattro diversi stati: i) respirazione di routine – la respirazione in un rispettivo mezzo respiratorio senza alcuna aggiunta di substrati o inibitori che consumano substrati ma endogeni. Questo stato può rivelare OXPHOS generale o difetti respiratori indotti secondariamente causati, ad esempio, da profili metabolici alterati. Successivamente, l’aggiunta dell’oligomicina inibitore dell’ATPasi rivela la permeabilità della membrana mitocondriale interna ai protoni, definita come ii) perdita respiratoria. La successiva titolazione di un protonoforo come il cianuro di carbonile uncoupler p-trifluoro-metossifenil-idrazzone (FCCP) consente di determinare lo stato in cui la capacità ETS è massima in un circuito protonico aperto-transmembrana, definito come iii) respirazione disaccoppiata. È importante sottolineare che uno stato disaccoppiato può verificarsi anche da interventi sperimentali attraverso un eccessivo danno meccanico alle membrane mitocondriali. Al contrario, lo stato non accoppiato si riferisce al disaccoppiamento respiratorio da parte di un meccanismo intrinseco fisiologicamente controllato. Infine, la completa inibizione dell’ETS mediante aggiunta dell’antimicina inibitore del complesso III e dell’inibitore del complesso I rotenone determina il consumo residuo di ossigeno (ROX) da processi non mitocondriali che consumano ossigeno (Figura 1A-C).
La bioenergetica mitocondriale è costituita da cinque distinti stati respiratori19,20. La respirazione dello stato 1 è senza substrati aggiuntivi o ADP, ad eccezione di ciò che è disponibile endogenamente. Dopo l’aggiunta di ADP, ma ancora, nessun substrato, si ottiene la respirazione dello stato 2. Quando vengono aggiunti substrati, consentendo il trasferimento di elettroni e la sintesi di ATP, viene raggiunta la respirazione dello stato 3. In questo stato, la capacità di OXPHOS può essere definita a concentrazioni saturanti di ADP, fosfato inorganico, ossigeno, NADH- e substrati legati al succinato. La respirazione allo stato 4 o la respirazione LEAK può essere definita come uno stato senza ADP o ATP sintasi chimicamente inibite pur avendo substrati sufficienti. Infine, quando tutto l’ossigeno è esaurito (anossico) in un ambiente a camera chiusa, si osserva la respirazione allo stato 5.
Esistono diversi metodi per valutare gli stati energetici cellulari14 con due dispositivi che dominano l’attuale valutazione in tempo reale di OXPHOS attraverso l’analisi del consumo di ossigeno, misurato come funzione della diminuzione dell’ossigeno nel tempo in un sistema a camera chiusa con diversa applicabilità a seconda del modello sperimentale e della domanda di ricerca: il respirometro ad alta risoluzione Oroboros 2k e l’analizzatore di flusso extracellulare Seahorse XF. Entrambi i dispositivi registrano i tassi di consumo di ossigeno come una diminuzione dei picomoli (pmol) di ossigeno (O2) al secondo come valore assoluto all’interno della camera o del pozzo della micropiastra. Il consumo specifico di ossigeno per massa si ottiene normalizzando il rispettivo consumo di ossigeno in una specifica ricetta tampone per numero di cellule (milioni), peso dei tessuti (mg) o quantità di proteine.
L’O2k (Oroboros Instruments) è un sistema chiuso a due camere dotato di un sensore polarografico di ossigeno (abbreviato in respirometro ad alta risoluzione basato su camera: cHRR). Ogni camera sperimentale contiene 2 ml di liquido che viene mantenuto omogeneo da agitatori magnetici. Il sensore polarografico di ossigeno utilizza un approccio amperometrico per misurare l’ossigeno: contiene un catodo d’oro, un anodo di cloruro d’argento / argento e tra una soluzione KCI che crea una cella elettrochimica su cui viene applicata una tensione (0,8 V). L’ossigeno proveniente dal mezzo di analisi si diffonde attraverso una membrana fluorurata di etilene propilene da 25 μm (permeabile A2) e subisce una riduzione al catodo, producendo perossido di idrogeno. All’anodo, l’argento viene ossidato dal perossido di idrogeno, generando una corrente elettrica. Questa corrente elettrica (ampere) è linearmente correlata alla pressione parziale dell’ossigeno. La pressione parziale dell’ossigeno e il fattore di solubilità dell’ossigeno del mezzo di saggio vengono utilizzati per calcolare la concentrazione di ossigeno. Poiché la pressione parziale dell’ossigeno dipende dalla temperatura sperimentale e le misurazioni polarografiche sono sensibili alla temperatura, le fluttuazioni di temperatura richiedono una regolazione precisa (±0,002 °C) da parte di un blocco riscaldante Peltier. La temperatura può essere controllata in un intervallo di 4 °C e 47 °C.
L’analizzatore di flusso extracellulare Seahorse XF (Agilent) è un sistema basato su piastre con formato micropiastra a 24 o 96 pozzetti in cui tre elettrodi di fluorescenza misurano il consumo di ossigeno nel tempo in ciascun pozzo (abbreviato in respirometro ad alta risoluzione basato su micropiastre: mHRR). Un massimo di quattro porte nella cartuccia del test sono disponibili per l’iniezione automatica durante il test. Un test contiene più cicli, ciascuno con tre fasi: 1) miscelazione, 2) attesa e 3) misurazione. Durante la fase di misurazione, le sonde del sensore vengono abbassate nella micropiastra creando una camera temporaneamente chiusa contenente un volume di 7-10 μL per misurare la luce emessa. Questa luce viene emessa da fluorofori incorporati in polimeri sulla punta delle sonde del sensore, che rilevano O2 in base alla tempra a fosforescenza. L’intensità del segnale di fluorescenza è proporzionale a O2 e influenzata dalla temperatura del sensore e del mezzo di saggio. Pertanto, una stima accurata dell’ossigeno richiede un approccio relativo con un pozzo di fondo senza alcun campione. Il ripristino della concentrazione di ossigeno avviene durante la fase di miscelazione quando il sensore si muove su e giù per mescolare il volume sopra la camera temporanea. Ogni ciclo calcola un tasso di consumo di ossigeno. La temperatura può essere controllata in un intervallo di 16 °C e 42 °C.
HRR è il gold standard per valutare la bioenergetica cellulare nelle malattie primarie e associate ai mitocondri e nel metabolismo cellulare generale. In questo studio, vengono forniti protocolli di base per HRR per valutare la funzione di OXPHOS nelle cellule e nei tessuti.
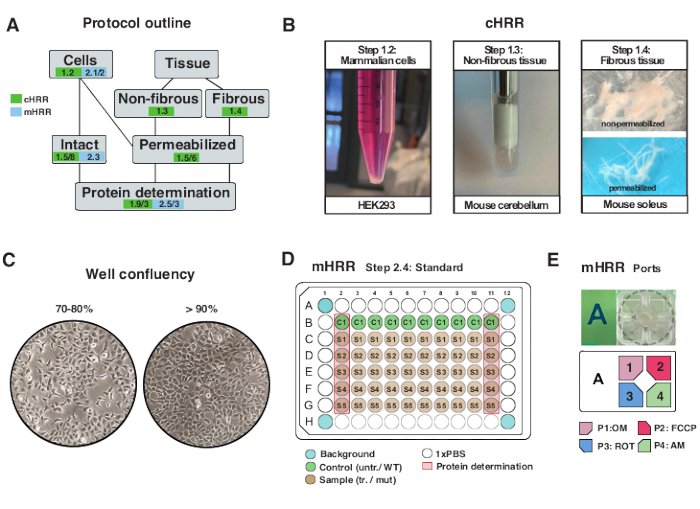
Figura 2: Flusso di lavoro per i preparati cellulari e tissutali per la cHRR e preparazione cellulare per la respirometria mHRR. (A) Schema dei protocolli forniti. (B) Cellule di mammifero (fase 1.2): hek293 pellet pari a 3 x 106 celle (pannello di sinistra). Tessuto non fibroso (fase 1.3): Preparazione del lisato di cervelletto murino in vasaio di teflon da 2 ml (pannello centrale). Permeabilizzazione del muscolo scheletrico indotta da saponina (pannello destro passo 1.4) per la respirometria cHRR. (C) Layout standard di semina delle micropiastre (fase 2.4) e controllo della confluenza per l’analisi delle cellule eucariotiche (HEK293) per la respirometria mHRR. (D, E) Schema di carico della porta di iniezione per la respirometria mHRR (fase 2.4). Fare clic qui per visualizzare una versione più grande di questa figura.
Protocol
Representative Results
Discussion
Tradizionalmente, la bioenergetica mitocondriale è stata studiata con elettrodi di ossigeno di tipo Clark. Una mancanza di risoluzione e throughput, tuttavia, giustificata per il progresso tecnologico. Ad oggi, l’O2k (indicato come cHRR) e il Seahorse XF96 Flux Analyzer (denominato mHRR) sono stati ampiamente adottati nel campo della bioenergetica cellulare. Qui, presentiamo una raccolta comprensibile di protocolli per l’analisi del metabolismo energetico cellulare attraverso la valutazione della respirazione mitocondri…
Disclosures
The authors have nothing to disclose.
Acknowledgements
Questo lavoro è stato sostenuto da finanziamenti dell’Accademia di Finlandia (C.B.J),della Magnus Ehrnroot Foundation (C.B.J) e da una borsa di dottorato della Integrated Life Sciences Graduate School (R.A.).
Materials
| 2 mL Potter-Elvehjem Glass/PTFE Tissue Grinder/Homogenizer | Omni International | 07-358029 | |
| 95% O2, 5% CO2 medical gas mixture | Potter for tissue grinding | ||
| ADP | Sigma | A 4386 | |
| Antimycin A | Sigma | A 8674 | Chemical |
| Ascorbate | Merck | PHR1279-1G | Chemical, dissolve in ethanol |
| BSA (fatty accid free) | Sigma | A 6003 | Chemical |
| CaCO3 | Sigma | C 4830 | Chemical |
| Cytochrome c | Sigma | C 7752 | Chemical |
| Digitonin | Sigma | D 5628 | Chemical |
| Dithiothreitol | Sigma | D 0632 | Chemical, dissolve in DMSO |
| D-Sucrose | Roth | 4621.1 | Chemical |
| Dulbecco’s modified Eagle’s medium (High glucose) | Fisher Scientific | 41965-039 | Chemical |
| Dulbecco’s modified Eagle’s medium (No Glucose) | Fisher Scientific | A14430-01 | |
| EGTA | Sigma | E 4378 | |
| Etomoxir | Sigma | E1905 | Chemical |
| Falcon 15 ml Conical Centrifuge Tubes | Fisher Scientific | AM12500 | Chemical |
| Falcon 50 ml Conical Centrifuge Tubes | Fisher Scientific | AM12501 | |
| FCCP | Sigma | C 2920 | |
| Glucose | Sigma | G7021 | Chemical, dissolve in ethanol |
| Glutamate | Sigma | G 1626 | Chemical |
| GlutaMax (100x) (200 nM L-alanyl-L-glutamine dipeptide) | Fisher Scientific | 35050061 | Chemical |
| HEK293 cells | ATTC | CRL-1573 | |
| Hemocytometer | Fisher Scientific | 0267151B | Instrument for cell counting |
| Hepes | Sigma | H 7523 | Chemical |
| Imidazole | Fluka | 56750 | Chemical |
| KCl | Merck | 1.04936 | Chemical |
| L-carnitine | Sigma | C0283 | Chemical |
| Malate | Sigma | M 1000 | Chemical |
| MES hydrate | Sigma | M8250 | Chemical |
| MgCl2 | Sigma | M 9272 | Chemical |
| Na2ATP | Sigma | A 2383 | Chemical |
| Na2Phosphocreatine | Sigma | P 7936 | Chemical |
| Na-pyruvate (100 mM) (100x) | Fisher Scientific | 11360070 | |
| NEAA (Non-essential amino acids) 100x | Fisher Scientific | 11140035 | |
| Normal FBS (10x) | Fisher Scientific | 10500064 | |
| O2k-Core: Oxygraph-2k | Oroboros Instruments | 10000-02 | High-resolution respirometry instrument |
| O2k-Titration Set | Oroboros Instruments | 20820-03 | Hamilton syringes for chemical injections |
| Oligomycin | Sigma | O 4876 | Chemical, dissolve in ethanol |
| Palmitoylcarnitine | Sigma | P 4509 | Chemical |
| Penicillin-Streptomycin | Fisher Scientific | 15140122 | |
| Pierce BCA Protein Assay Kit | Fisher Scientific | 23227 | |
| Pyruvate | Sigma | P 2256 | Chemical |
| RIPA-Buffer | Fisher Scientific | 89900 | Chemical |
| Rotenone | Sigma | R 8875 | Chemical, dissolve in ethanol |
| Saponin | Sigma | S7900 | Chemical |
Seahorse XF DMEM assay medium pack, pH 7.4 |
Agilent, Santa Clara, CA |
103680-100 | |
| Seahorse XFe96 Extracellular Flux Analyzer | Agilent, Santa Clara, CA |
High-throughput respirometry instrument | |
| Seahorse XFe96 FluxPak | Agilent, Santa Clara, CA |
Includes assay plates, cartridges, loading guides for transferring compounds to the assay cartridge, and calibrant solution. |
|
| Small scissors | Fisher Scientific | 08-951-20 | |
| Sodium azide | Sigma | S2002 | Chemical |
| Succinate | Sigma | S 2378 | Chemical |
| Taurine | Sigma | T 8691 | Chemical |
| TMPD | Sigma | T 3134 | Chemical |
| Trypan Blue solution | Merck | 72-57-1 | Chemical |
| Trypsin 0.25% EDTA | Fisher Scientific | 25200056 | |
| Two thin-edged forceps | Fisher Scientific | 12-000-122 | |
| Uridine stock (500x) | Sigma | U3750 | Chemical |
References
- McBride, H. M., Neuspiel, M., Wasiak, S. Mitochondria: More than just a powerhouse. Current Biology. 16 (14), 551-560 (2006).
- Mehta, M. M., Weinberg, S. E., Chandel, N. S. Mitochondrial control of immunity. Beyond ATP. Nature Reviews Immunology. 17 (10), 608-620 (2017).
- Spinelli, J. B., Haigis, M. C. The multifaceted contributions of mitochondria to cellular metabolism. Nature Cell Biology. 20 (7), 745-754 (2018).
- Gnaiger, E. Capacity of oxidative phosphorylation in human skeletal muscle. New perspectives of mitochondrial physiology. International Journal of Biochemistry and Cell Biology. 41 (10), 1837-1845 (2009).
- Gorman, G. S., et al. Mitochondrial diseases. Nature Reviews Disease Primers. 2, 1-23 (2016).
- Boushel, R., Gnaiger, E., Schjerling, P., Skovbro, M., Kraunsøe, R., Dela, F. Patients with type 2 diabetes have normal mitochondrial function in skeletal muscle. Diabetologia. 50 (4), 790-796 (2007).
- Cogliati, S., et al. Mitochondrial cristae shape determines respiratory chain supercomplexes assembly and respiratory efficiency. Cell. 155 (1), 160-171 (2013).
- Kühlbrandt, W. Structure and function of mitochondrial membrane protein complexes. BMC Biology. 13, 1-11 (2015).
- Gnaiger, E. Mitochondrial pathways and Respiratory control. An introduction to OXPHOS analysis. Bioenergetics communications. 5th ed. , (2020).
- Jackson, C. B., et al. Mutations in SDHD lead to autosomal recessive encephalomyopathy and isolated mitochondrial complex II deficiency. Journal of Medical Genetics. 51 (3), 170-175 (2014).
- Pesta, D., Gnaiger, E. High-resolution respirometry: OXPHOS protocols for human cells and permeabilized fibers from small biopsies of human muscle. Methods in Molecular Biology. 810, 25-58 (2012).
- Horan, M. P., Pichaud, N., Ballard, J. W. O. Review: Quantifying mitochondrial dysfunction in complex diseases of aging. Journals of Gerontology – Series A Biological Sciences and Medical Sciences. 67 (10), 1022-1035 (2012).
- Doerrier, C., Garcia-Souza, L. F., Krumschnabel, G., Wohlfarter, Y., Mészáros, A. T., Gnaiger, E. High-resolution fluorespirometry and oxphos protocols for human cells, permeabilized fibers from small biopsies of muscle, and isolated mitochondria. Methods in Molecular Biology. 1782, 31-70 (2018).
- Zhang, J., et al. Measuring energy metabolism in cultured cells, including human pluripotent stem cells and differentiated cells. Nature Protocols. 7 (6), 1068-1085 (2012).
- García-Roche, M., Casal, A., Carriquiry, M., Radi, R., Quijano, C., Cassina, A. Respiratory analysis of coupled mitochondria in cryopreserved liver biopsies. Redox Biology. 17, 207-212 (2018).
- Acin-Perez, R., et al. A novel approach to measure mitochondrial respiration in frozen biological samples. The EMBO Journal. 39 (13), 1-18 (2020).
- Cell metabolism assay kits. Seahorse assay kits and media Available from: https://www.agilent.com/en/product/cell-analysis/real-time-cell-metabolic-analysis/xf-assay-lits-reagents-cell-assay-media (2021)
- Chance, B., Williams, G. R. A method for the localization of sites for oxidative phosphorylation. Nature. 176 (4475), 250-254 (1955).
- Gnaiger, E., et al. Mitochondrial respiratory states and rates. MitoFit Preprint Arch. , (2019).
- Gnaiger, E. O2k-procedures: SOP O2k quality control 1: Polarographic oxygen sensors and accuracy of calibration Section Page. Oroboros. 03 (18), 1-21 (2020).
- Robinson, B. H., Petrova-Benedict, R., Buncic, J. R., Wallace, D. C. Nonviability of cells with oxidative defects in galactose medium: A screening test for affected patient fibroblasts. Biochemical Medicine and Metabolic Biology. 48 (2), 122-126 (1992).
- King, M. P., Attardi, G. Human cells lacking mtDNA: Repopulation with exogenous mitochondria by complementation. Science. 246 (4929), 500-503 (1989).
- Makrecka-Kuka, M., Krumschnabel, G., Gnaiger, E. High-resolution respirometry for simultaneous measurement of oxygen and hydrogen peroxide fluxes in permeabilized cells, tissue homogenate and isolated mitochondria. Biomolecules. 5 (3), 1319-1338 (2015).
- Fasching, M., Gnaiger, E. O2k quality control 2: Instrumental oxygen background correction and accuracy of oxygen flux. Mitochondrial Physiology Network. 14 (06), 1-14 (2016).
- Gnaiger, E., Lassnig, B., Kuznetsov, A., Rieger, G., Margreiter, R. Excess capacity of cytochrome c oxidase. Journal of Experimental Biology. 1139, 1129-1139 (1998).
- Gnaiger, E., et al. Mitochondria in the Cold. Life in the Cold. , 431-442 (2000).
- Fontana-Ayoub, M., Fasching, E., Gnaiger, Selected media and chemicals for respirometry with mitochondrial preparations. Mitochondrial Physiology Network. 02 (17), 1-9 (2014).
- Gerencser, A. A., et al. Quantitative microplate-based respirometry with correction for oxygen diffusion. Analytical Chemistry. 81 (16), 6868-6878 (2009).
- Krumschnabel, G., Eigentler, A., Fasching, M., Gnaiger, E. Use of safranin for the assessment of mitochondrial membrane potential by high-resolution respirometry and fluorometry. Methods in Enzymology. 542, 163-181 (2014).
- Nászai, A., Terhes, E., Kaszaki, J., Boros, M., Juhász, L. Ca(2+)N it be measured? Detection of extramitochondrial calcium movement with high-resolution fluorespirometry. Scientific Reports. 9 (1), 1-13 (2019).
- Pajak, B., et al. 2-Deoxy-d-Glucose and its analogs: From diagnostic to therapeutic agents. International Journal of Molecular Sciences. 21 (1), 234 (2019).
- Mercier-Letondal, P., Marton, C., Godet, Y., Galaine, J. Validation of a method evaluating T cell metabolic potential in compliance with ICH Q2 (R1). Journal of Translational Medicine. 19 (1), 1-15 (2021).
- Sauerbeck, A., et al. Analysis of regional brain mitochondrial bioenergetics and susceptibility to mitochondrial inhibition utilizing a microplate based system. Journal of Neuroscience Methods. 198 (1), 36-43 (2011).
- Jackman, M. R., Willis, W. T. Characteristics of mitochondria isolated from type I and type IIb skeletal muscle. American Journal of Physiology – Cell Physiology. 270 (2), 673-678 (1996).
- Ponsot, E., et al. Mitochondrial tissue specificity of substrates utilization in rat cardiac and skeletal muscles. Journal of Cellular Physiology. 203 (3), 479-486 (2005).
- Schönfeld, P., Reiser, G. Why does brain metabolism not favor burning of fatty acids to provide energy-Reflections on disadvantages of the use of free fatty acids as fuel for brain. Journal of Cerebral Blood Flow and Metabolism. 33 (10), 1493-1499 (2013).
- Calderon-Dominguez, M., Mir, J. F., Fucho, R., Weber, M., Serra, D., Herrero, L. Fatty acid metabolism and the basis of brown adipose tissue function. Adipocyte. 5 (2), 98-118 (2016).
- Divakaruni, A. S., Rogers, G. W., Murphy, A. N. Measuring mitochondrial function in permeabilized cells using the seahorse XF analyzer or a clark-type oxygen electrode. Current Protocols in Toxicology. 2014, 1-16 (2014).
- Iuso, A., Repp, B., Biagosch, C., Terrile, C., Prokisch, H. Assessing mitochondrial bioenergetics in isolated mitochondria from various mouse tissues using Seahorse XF96 analyzer. Methods in Molecular Biology. 1567, 217-230 (2017).
- Rogers, G. W., et al. High throughput microplate respiratory measurements using minimal quantities of isolated mitochondria. PLoS ONE. 6 (7), 21746 (2011).
- Jordá, A., Zaragozá, R., Portolés, M., Báguena-Cervellera, R., Renau-Piqueras, J. Long-term high-protein diet induces biochemical and ultrastructural changes in rat liver mitochondria. Archives of Biochemistry and Biophysics. 265 (2), 241-248 (1988).
- Jackson, C. B., Gallati, S., Schaller, A. QPCR-based mitochondrial DNA quantification: Influence of template DNA fragmentation on accuracy. Biochemical and Biophysical Research Communications. 423 (3), 441-447 (2012).
- Hirsch, H. M. Tissue autoxidation inhibitors: II. The presence of inhibitor in intact cells; Assay of liver and hepatoma effect on radio-oxidations. 癌症研究. 16 (11), 1076-1082 (1956).
- Picard, M., et al. Mitochondrial structure and function are disrupted by standard Isolation methods. PLoS ONE. 6 (3), 18317 (2011).
- Tanumihardja, E., Slaats, R. H., Van Der Meer, A. D., Passier, R., Olthuis, W., Van Den Berg, A. Measuring both pH and O2 with a single On-Chip sensor in cultures of human pluripotent stem cell-derived cardiomyocytes to track induced changes in cellular metabolism. ACS Sensors. 6 (1), 267-274 (2021).
- Harms, F., Stolker, R. J., Mik, E. Cutaneous respirometry as novel technique to monitor mitochondrial function: A feasibility study in healthy volunteers. PLoS ONE. 11 (7), 159544 (2016).
- Levitsky, Y., et al. Micro-respirometry of whole cells and isolated mitochondria. RSC Advances. 9 (57), 33257-33267 (2019).

