Estudos de Interações Acompanhante-Cochaperone usando ensaio homogêneo baseado em contas
Summary
Este protocolo apresenta uma técnica para sondar interações proteína-proteína usando contas doadoras ligadas à glutationa com co-acompanhantes e contas aceitadoras de TPR fundidas gst-fused e contas aceitadoras juntamente com um peptídeo derivado de Hsp90. Usamos essa técnica para rastrear pequenas moléculas para interromper as interações de interação Hsp90-FKBP51 ou Hsp90-FKBP52 e identificamos inibidores potentes e seletivos de interação Hsp90-FKBP51.
Abstract
O direcionamento da proteína de choque térmico 90 (Hsp90)-cochaperona interações fornece a possibilidade de regular especificamente os processos intracelulares dependentes de Hsp90. O pentapeptídeo meevd conservado no C-terminus de Hsp90 é responsável pela interação com o motivo de repetição de tetratricopeptida (TPR) de co-acompanhantes. FK506-binding protein (FKBP) 51 e FKBP52 são dois co-acompanhantes similares TPR-motivo envolvidos em doenças dependentes de hormônios esteroides com diferentes funções. Portanto, identificar moléculas especificamente bloqueando interações entre Hsp90 e FKBP51 ou FKBP52 fornece um potencial terapêutico promissor para várias doenças humanas. Aqui, descrevemos o protocolo para um ensaio homogêneo de proximidade luminescente amplificado para sondar interações entre Hsp90 e seus co-acompanhantes parceiros FKBP51 e FKBP52. Primeiro, purificamos as proteínas contendo motivos TPR FKBP51 e FKBP52 na forma de glutationa S-transferase (GST) marcada. Usando as contas doadoras ligadas à glutationa com proteínas TPR-motivos fundidas gst e as contas aceitadoras juntamente com um peptídeo terminal de 10-mer C de Hsp90, sondamos interações proteína-proteína em um ambiente homogêneo. Usamos este ensaio para testar pequenas moléculas para interromper as interações de interação Hsp90-FKBP51 ou Hsp90-FKBP52 e identificamos inibidores potentes e seletivos de interação Hsp90-FKBP51.
Introduction
Acompanhantes moleculares contribuem para a homeostase proteica, incluindo dobramento de proteínas, transporte e degradação. Eles regulam diversos processos celulares e estão ligados a inúmeras doenças como câncer e doenças neurodegenerativas1. A proteína de choque térmico 90 (Hsp90) é uma das acompanhantes mais importantes cuja função depende de alterações conformais impulsionadas pela hidrólise ATP e vinculantes com proteínas do cliente mediadas por seus acompanhantes2. Apesar de um potencial óbvio do Hsp90 como alvo terapêutico, o ajuste fino de sua função representa um grande desafio. Existem vários inibidores de Hsp90 voltados para a região de ligação ATP n-terminal, que foram avaliados em ensaios clínicos, mas nenhum deles foi aprovado para comercialização3. Devido à falta de um bolso de ligação de ligantes bem definido4,visando a região do terminal C de Hsp90 teve sucesso limitado4. Recentemente, a interrupção das interações de Hsp90-cochaperona por pequenas moléculas tem sido investigada como uma estratégia alternativa5. Direcionar as interações de cochaperona Hsp90 não provocaria resposta geral ao estresse celular e proporcionaria a possibilidade de regular especificamente vários processos intracelulares. O pentapeptida meevd conservado no C-terminus de Hsp90 é responsável pela interação com o motivo de repetição de tetratricopeptida (TPR) dos co-acompanhantes6. Das 736 proteínas contendo motivos TPR anotadas no banco de dados de proteínas humanas, ~20 proteínas diferentes interagem com o Hsp90 através deste peptídeo7. Moléculas competindo por ligação de peptídeos MEEVD interromperiam as interações entre Hsp90 e co-acompanhantes contendo um domínio TPR. O local de ligação de peptídeos tem estrutura terciária semelhante, mas a homologia geral entre diferentes domínios de motivos TPR é relativamente baixa7, proporcionando uma oportunidade de identificar moléculas especificamente capazes de bloquear interações entre Hsp90 e co-acompanhantes particulares do TPR-motivo. Entre esses co-acompanhantes de TPR-motivo, fk506-binding protein (FKBP) 51 e FKBP52 são reguladores de sinalização do receptor hormonal esteroide (SHR) e envolvidos em várias doenças dependentes de hormônios esteroides, incluindo câncer, doenças relacionadas ao estresse, doenças metabólicas e doença de Alzheimer8. Embora as ações FKBP51 e FKBP52 > similaridade de sequência de 80%, suas funções diferem: FKBP52 é um regulador positivo da atividade SHR, enquanto FKBP51 é um regulador negativo na maioria dos casos8. Portanto, identificar moléculas, bloqueando especificamente as interações entre Hsp90 e FKBP51 ou FKBP52, fornece um potencial terapêutico promissor para doenças relacionadas.
Ummplified Luminescent Proximity Homogenous Assay (AlphaScreen) foi desenvolvido pela primeira vez em 1994 por Ullman EF et al.9. Agora é amplamente utilizado para detectar diferentes tipos de interações biológicas, como peptídeo10, proteína11,DNA12, RNA13e açúcar14. Nesta técnica, existem dois tipos de contas (diâmetro 200 nm), uma é a conta doadora e a outra é a conta aceitadora. As biomoléculas são imobilizadas sobre essas contas; suas interações biológicas trazem contas doadoras e aceitadoras para a proximidade. A 680 nm, um fotoensibilizador na conta doadora ilumina e converte oxigênio em oxigênio único. Como o oxigênio único tem uma vida curta, ele só pode difundir até 200 nm. Se o cordão aceitor estiver próximo, seu derivado de tiaxieno reage com o oxigênio único gerando quemiluminescência a 370 nm. Esta energia ativa ainda mais fluoroforos no mesmo projeto de conta aceitador para emitir luz em 520-620 nm15. Se as interações biológicas forem interrompidas, a conta aceitadora e o doador não podem chegar à proximidade, resultando na decadência de oxigênio única e no sinal de baixa produção.
Aqui descrevemos um protocolo usando esta técnica para triagem de pequenas moléculas inibindo interações entre co-acompanhantes de Hsp90 e TPR, especialmente FKBP51 e FKBP52. Os 10 peptídeos longos de aminoácidos correspondentes ao Hsp90 extremo C-terminus estão ligados a contas aceitadoras. Co-acompanhantes de TPR com marca GST purificados interagem com contas doadoras ligadas à glutona. Quando a interação entre peptídeos derivados de Hsp90 e co-acompanhantes de TPR-motivos reúne as contas, um sinal amplificado é produzido(Figura 1A). Se as pequenas moléculas rastreadas puderem inibir as interações entre os co-acompanhantes de Hsp90 e TPR-motivo, esse sinal amplificado será diminuído(Figura 1B). Seu IC50 pode ser calculado por medição quantitativa. Este protocolo pode ser estendido a qualquer acompanhante – TPR-motivo co-acompanhante interações de interesse e é de grande importância no desenvolvimento de novas moléculas, bloqueando especificamente a interação entre Hsp90 e FKBP51 ou FKBP52.
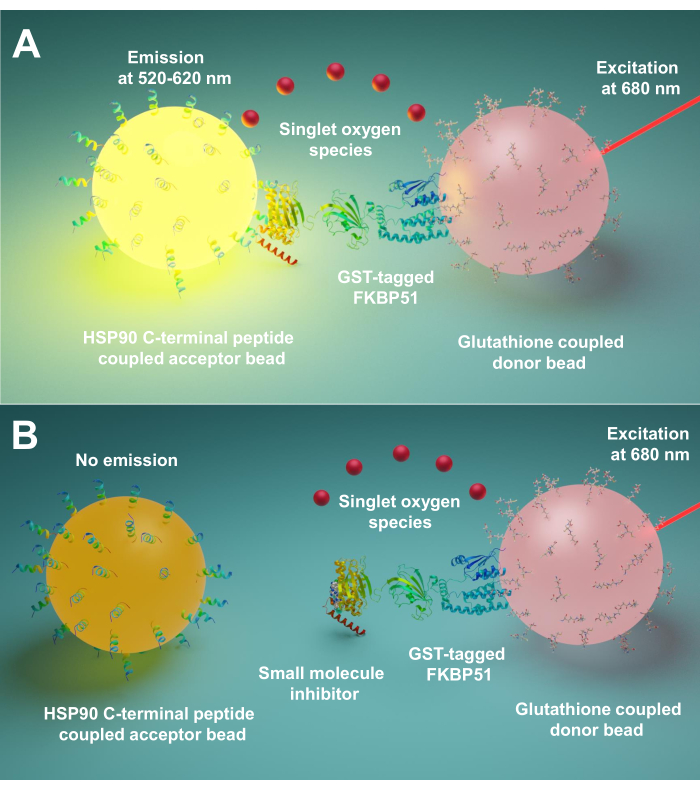
Figura 1: O princípio básico deste ensaio. (A) GST-FKBP51 purificado interage com contas doadoras ligadas à glutationa. Os 10 peptídeos longos de aminoácidos correspondentes ao extremo C-terminus de Hsp90 estão ligados a contas aceitadoras. A interação entre peptídeos derivados de Hsp90 e domínio TPR do FKBP51 aproxima o doador e o acceptor beads. A 680 nm, um fotoensibilizador na conta doadora ilumina e converte oxigênio em oxigênio único. A derivada de tiaxieno no projeto de garantia reage com o oxigênio singlet e gera chemiluminescence a 370 nm. Esta energia ativa ainda mais fluoroforos no mesmo projeto de conta aceitador para emitir luz a 520-620 nm. (B) Quando pequenas moléculas inibem as interações entre Hsp90 e FKBP51, o doador e as contas aceitadoras não podem alcançar a proximidade. Em seguida, o oxigênio singlet com decaimentos de curta duração, e nenhum sinal detectável é produzido. Clique aqui para ver uma versão maior desta figura.
Protocol
Representative Results
Discussion
Aqui descrevemos um protocolo usando o ensaio para triagem de pequenas moléculas inibindo interações entre co-acompanhantes de Hsp90 e TPR-motivo, especialmente FKBP51 e FKBP52. Sua pontuação Z alta (>0,8) demonstra a robustez e confiabilidade para um formato de alto rendimento. Os resultados podem ser obtidos dentro de uma hora, e pequenas quantidades de contas, proteínas e compostos são necessárias. Além disso, este protocolo poderia facilmente ser estendido a qualquer interação de interesse de Hsp90/Hsp70 -…
Offenlegungen
The authors have nothing to disclose.
Acknowledgements
Este estudo foi apoiado por subsídios do Conselho Sueco de Pesquisa (2018-02843), Fundação Brain (Fo 2019-0140), Fundação para Doenças Geriátricas do Instituto Karolinska, Fundação Gunvor e Josef Anérs, Fundação Magnus Bergvalls, Fundação Gun e Bertil Stohnes, Fundação Tore Nilssons para pesquisa médica, Fundação Margaret Afha Ugglas e Fundação
Materials
| 384-well plates | Perkin Elmer | 6008350 | Assay volume 25 ml |
| Amicon 10.000 MWCO centrifugation unit | Millipore | UFC901008 | Concentrate protein |
| Ampicillin | Sigma | A0166 | Antibiotics |
| Bacteria shaker Unimax 1010 | Heidolph | Culture bacteria | |
| cDNA clones for human FKBP51 | Source BioScience | clone id: 5723416 | pCMV-SPORT6 vector |
| cDNA clones for human FKBP52 | Source BioScience | clone id: 7474554 | pCMV-SPORT6 vector |
| Chemically Competent E. coli | Invitrogen | C602003 | One Shot BL21 Star (DE3) |
| Data analysis software | GraphPad Prism | 9.0.0 | Analysis data and make figures |
| Data analysis software | Excel | Analysis data | |
| DMSO | Supelco | 1.02952.1000 | Dilute compounds |
| DPBS | Gibco | 14190-144 | Prepare solution |
| EDTA | Calbiochem | 344504 | Prevent proteolysis during sonication |
| Glutathione | Sigma | G-4251 | Elute GST-tagged proteins |
| Glutathione donor beads | Perkin Elmer | 6765300 | Donor bead |
| GST-trap column | Cytiva (GE Healthcare) | 17528201 | Purify GST-tagged proteins |
| Isopropyl-β-D-thiogalactoside | Thermo Fisher Scientific | R0392 | Induce protein expression |
| LB Broth (Miller) | Sigma | L3522 | Microbial growth medium |
| PCR instrument | BIO-RAD | S1000 Thermal Cycler | Amplification/PCR |
| PD-10 column | Cytiva (GE Healthcare) | 17085101 | Solution exchange |
| pGEX-6P-1 vector | Cytiva (GE Healthcare) | 28954648 | Plasmid |
| pGEX-6P-2 vector | Cytiva (GE Healthcare) | 28954650 | Plasmid |
| Plate reader | Perkin Elmer | EnSpire 2300 Multilabel Reader | Read alpha plate |
| Plate reader software | Perkin Elmer | EnSpire Manager | Plate reader software |
| Plate reader software protocol | Perkin Elmer | Alpha 384-well Low volume | Use this protocol to read plate |
| PMSF | Sigma | P7626 | Prevent proteolysis during sonication |
| protease inhibitor cocktail | Sigma | S8830 | Prevent proteolysis during sonication |
| Sodium azide | Sigma | S2002 | As a preservative |
| Sodium cyanoborohydride (NaBH3CN) | Sigma | 156159 | Activates matrix for coupling |
| Ten amino acid peptide NH2-EDASRMEEVD-COOH corresponding to amino acids 714-724 of human Hsp90 beta isoform | Peptide 2.0 inc | Synthesize Hsp90 C-terminal peptide | |
| Test-Tube Rotator | LABINCO | Make end-over-end agitation | |
| Tris-HCl | Sigma | 10708976001 | Block unreacted sites of acceptor beads |
| Tween-20 | Sigma | P1379 | Prevent beads aggregation |
| Ultra centrifuge Avanti J-20 XP | Beckman Coulter | Centrifuge to get bacteria cell pellets | |
| Ultrasonic cell disruptor | Microson | Sonicate cells to release protein | |
| Unconjugated acceptor beads | Perkin Elmer | 6762003 | Acceptor beads |
| XCell SureLock Mini-Cell and XCell II Blot Module | Invitrogen | EI0002 | SDS-PAGE |
Referenzen
- Muchowski, P. J., Wacker, J. L. Modulation of neurodegeneration by molecular chaperones. Nature Reviews Neuroscience. 6 (1), 11-22 (2005).
- Eckl, J. M., Richter, K. Functions of the Hsp90 chaperone system: lifting client proteins to new heights. International Journal of Biochemistry and Molecular Biology. 4 (4), 157-165 (2013).
- Yuno, A. Clinical evaluation and biomarker profiling of Hsp90 inhibitors. Methods in Molecular Biology. 1709, 426-441 (2018).
- Dutta Gupta, S., Bommaka, M. K., Banerjee, A. Inhibiting protein-protein interactions of Hsp90 as a novel approach for targeting cancer. European Journal of Medicinal Chemistry. 178, 48-63 (2019).
- Pavlov, P. F., Hutter-Paier, B., Havas, D., Windisch, M., Winblad, B. Development of GMP-1 a molecular chaperone network modulator protecting mitochondrial function and its assessment in fly and mice models of Alzheimer’s disease. Journal of Cellular and Molecular Medicine. 22 (7), 3464-3474 (2018).
- Young, J. C., Obermann, W. M., Hartl, F. U. Specific binding of tetratricopeptide repeat proteins to the C-terminal 12-kDa domain of hsp90. Journal of Biological Chemistry. 273 (29), 18007-18010 (1998).
- Scheufler, C., et al. Structure of TPR domain-peptide complexes: critical elements in the assembly of the Hsp70-Hsp90 multichaperone machine. Cell. 101 (2), 199-210 (2000).
- Storer, C. L., Dickey, C. A., Galigniana, M. D., Rein, T., Cox, M. B. FKBP51 and FKBP52 in signaling and disease. Trends in Endocrinology & Metabolism. 22 (12), 481-490 (2011).
- Ullman, E. F., et al. Luminescent oxygen channeling immunoassay: measurement of particle binding kinetics by chemiluminescence. Proceedings of the National Academy of Sciences of the United States of America. 91 (12), 5426-5430 (1994).
- Wigle, T. J., et al. Screening for inhibitors of low-affinity epigenetic peptide-protein interactions: an AlphaScreen-based assay for antagonists of methyl-lysine binding proteins. Journal of Biomolecular Screening. 15 (1), 62-71 (2010).
- Guenat, S., et al. Homogeneous and nonradioactive high-throughput screening platform for the characterization of kinase inhibitors in cell lysates. Journal of Biomolecular Screening. 11 (8), 1015-1026 (2006).
- Sabatucci, A., et al. A new methodological approach for in vitro determination of the role of DNA methylation on transcription factor binding using AlphaScreen(R) analysis: Focus on CREB1 binding at hBDNF promoter IV. Journal of Neuroscience Methods. 341, 108720 (2020).
- Mills, N. L., Shelat, A. A., Guy, R. K. Assay Optimization and Screening of RNA-Protein Interactions by AlphaScreen. Journal of Biomolecular Screening. 12 (7), 946-955 (2007).
- Huang, X., et al. A competitive alphascreen assay for detection of hyaluronan. Glycobiology. 28 (3), 137-147 (2018).
- Principles of alphascreen amplified luinescent proximmity homogenous assay. PerkinElmer Life Sciences Available from: https://www.perkinelmer.com/lab-solutions/resources/docs/APP_AlphaScreen_Principles.pdf (2021)
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple statistical parameter for use in evaluation and validation of high throughput screening assays. Journal of Biomolecular Screening. 4 (2), 67-73 (1999).

