Polymerasekettingreactie en dot-blot-hybridisatie voor leptospira-detectie in watermonsters
Summary
In deze studie werd een dot-blot-applicatie ontworpen om Leptospira te detecteren van de drie belangrijkste clades in watermonsters. Deze methode maakt de identificatie mogelijk van minimale DNA-hoeveelheden die specifiek het doelwit zijn van een digoxigenine-gelabelde sonde, die gemakkelijk kan worden gedetecteerd door een anti-digoxigenine-antilichaam. Deze aanpak is een waardevol en bevredigend instrument voor screeningsdoeleinden.
Abstract
De dot-blot is een eenvoudige, snelle, gevoelige en veelzijdige techniek die de identificatie mogelijk maakt van minimale hoeveelheden DNA die specifiek het doelwit zijn van sondehybridisatie in aanwezigheid van drager-DNA. Het is gebaseerd op de overdracht van een bekende hoeveelheid DNA op een inerte vaste drager, zoals een nylon membraan, met behulp van het dot-blot-apparaat en zonder elektroforetische scheiding. Nylonmembranen hebben het voordeel van een hoog nucleïnezuurbindend vermogen (400 μg/cm2), een hoge sterkte en zijn positief of neutraal geladen. De gebruikte sonde is een zeer specifiek ssDNA-fragment van 18 tot 20 basen dat lang is gelabeld met digoxigenine (DIG). De sonde zal zich conjugeren met het Leptospira-DNA . Zodra de sonde is gehybridiseerd met het doel-DNA, wordt deze gedetecteerd door een anti-digoxigenine-antilichaam, waardoor deze gemakkelijk kan worden gedetecteerd door de emissies die worden onthuld in een röntgenfilm. De stippen met een emissie komen overeen met de DNA-fragmenten die van belang zijn. Deze methode maakt gebruik van de niet-isotopische etikettering van de sonde, die een zeer lange halfwaardetijd kan hebben. Het nadeel van dit standaard immunolabel is een lagere gevoeligheid dan isotopensondes. Desalniettemin wordt het beperkt door polymerasekettingreactie (PCR) en dot-blot-assays te koppelen. Deze aanpak maakt de verrijking van de doelsequentie en de detectie ervan mogelijk. Bovendien kan het worden gebruikt als een kwantitatieve toepassing in vergelijking met een seriële verdunning van een bekende standaard. Een dot-blot-toepassing om Leptospira te detecteren van de drie belangrijkste clades in watermonsters wordt hier gepresenteerd. Deze methodologie kan worden toegepast op grote hoeveelheden water nadat ze zijn geconcentreerd door centrifugatie om bewijs te leveren van de aanwezigheid van Leptospiral DNA. Dit is een waardevol en bevredigend hulpmiddel voor algemene screeningsdoeleinden en kan worden gebruikt voor andere niet-kweekbare bacteriën die in water aanwezig kunnen zijn, waardoor het begrip van het ecosysteem wordt verbeterd.
Introduction
Leptospirose bij de mens is voornamelijk afkomstig van milieubronnen 1,2. De aanwezigheid van Leptospira in meren, rivieren en beken is een indicator van de overdracht van leptospirose onder dieren in het wild en huisdieren en productiedieren die uiteindelijk in contact kunnen komen met deze watermassa’s 1,3,4. Bovendien is Leptospira geïdentificeerd in niet-natuurlijke bronnen, waaronder rioolwater, stilstaand en kraanwater 5,6.
Leptospira is een wereldwijd verspreide bacterie 7,8, en de rol van het milieu bij het behoud en de overdracht ervan is algemeen erkend. Leptospira kan overleven in drinkwater met een variabele pH en mineralen9, en in natuurlijke waterlichamen1. Het kan ook lange tijd overleven in gedestilleerd water10, en onder een constante pH (7,8) kan het tot 152 dagen overleven11. Bovendien kan Leptospira interageren in bacteriële consortia om barre omstandigheden te overleven12,13. Het kan deel uitmaken van biofilms in zoet water met Azospirillum en Sphingomonas en is zelfs in staat om te groeien en temperaturen van meer dan 49 °C14,15 te doorstaan. Het kan zich ook vermenigvuldigen in drassige grond en tot 379 dagen levensvatbaar blijven16, waardoor het vermogen om de ziekte te veroorzaken tot een jaar lang behouden blijft17,18. Er is echter weinig bekend over de ecologie in waterlichamen en hoe deze daarin is verdeeld.
Sinds de ontdekking was de studie van het geslacht Leptospira gebaseerd op serologische tests. Pas in de huidige eeuw kwamen moleculaire technieken steeds vaker voor in de studie van deze spirochaete. De dot-blot is nauwelijks gebruikt voor zijn identificatie met behulp van (1) een isotopische sonde op basis van het 16S rRNA en op een inter-simple sequence repeat (ISSR)19,20, (2) als een op nanogoud gebaseerde immunoassay voor menselijke leptospirose toegepast op urine21, of (3) als een op antilichamen gebaseerde test voor runderurinemonsters22. De techniek raakte in onbruik omdat deze oorspronkelijk gebaseerd was op isotopische sondes. Het is echter een bekende techniek die, in combinatie met PCR, verbeterde resultaten oplevert en als veilig wordt beschouwd vanwege het gebruik van niet-isotopische sondes. PCR speelt een cruciale rol bij de verrijking van het Leptospira-DNA door een specifiek DNA-fragment te amplificeren dat in sporenhoeveelheden in een monster kan worden aangetroffen. Tijdens elke PCR-cyclus wordt de hoeveelheid van het beoogde DNA-fragment in de reactie verdubbeld. Aan het einde van de reactie is het amplicon vermenigvuldigd met een factor van meer dan een miljoen23. Het door PCR geamplificeerde product, vaak niet zichtbaar bij agarose-elektroforese, wordt zichtbaar door specifieke hybridisatie met een DIG-gelabelde sonde in de dot-blot 24,25,26.
De dot-blot-techniek is eenvoudig, robuust en geschikt voor tal van monsters, waardoor het toegankelijk is voor laboratoria met beperkte middelen. Het is gebruikt in een verscheidenheid aan bacteriestudies, waaronder (1) orale bacteriën27, (2) andere soorten monsters zoals voedsel en ontlasting28, en (3) de identificatie van niet-kweekbare bacteriën29, vaak in overeenstemming met andere moleculaire technieken. Een van de voordelen van de dot-blot-techniek zijn: (1) Het membraan heeft een hoge bindingscapaciteit, in staat om meer dan 200 μg/cm2 nucleïnezuren en tot 400 μg/cm2 te binden; (2) Dot-blot-resultaten kunnen visueel worden geïnterpreteerd zonder dat er speciale apparatuur nodig is, en (3) ze kunnen gemakkelijk jarenlang bij kamertemperatuur (RT) worden bewaard.
Het geslacht Leptospira is ingedeeld in pathogene, intermediaire en saprofytische clades30,31. Het onderscheid tussen deze clades kan worden bereikt op basis van specifieke genen zoals lipL41, lipL32 en het 16S rRNA. LipL32 is aanwezig in de pathogene clades en vertoont een hoge gevoeligheid in verschillende serologische en moleculaire instrumenten, terwijl het afwezig is in saprofytensoorten21. Het huishoudgen lipL41 staat bekend om zijn stabiele expressie en wordt gebruikt in moleculaire technieken32, terwijl het 16S rRNA-gen wordt gebruikt voor hun classificatie.
Deze methodologie kan worden toegepast op grote hoeveelheden water nadat ze zijn geconcentreerd door centrifugeren. Het maakt het mogelijk om verschillende punten en diepten in een waterlichaam te beoordelen om de aanwezigheid van leptospiraalvormig DNA en de clade waartoe het behoort te detecteren. Deze tool is waardevol voor zowel ecologische als algemene screeningsdoeleinden en kan ook worden gebruikt om andere niet-kweekbare bacteriën te detecteren die in water aanwezig kunnen zijn.
Bovendien zijn PCR- en dot-blot-assays technisch en economisch betaalbaar voor een breed scala aan laboratoria, zelfs voor laboratoria die niet over geavanceerde of dure apparatuur beschikken. Deze studie heeft tot doel de op digoxigenine gebaseerde dot-blot toe te passen voor de identificatie van de drie Leptospira-clades in watermonsters die zijn verzameld uit natuurlijke waterlichamen.
Bacteriestammen
Twaalf Leptospira serovars (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi en Wolffi) werden in deze studie opgenomen. Deze serovars maken deel uit van de collectie van de afdeling Microbiologie en Immunologie, Faculteit Diergeneeskunde en Zoötechniek, Nationale Autonome Universiteit van Mexico, en ze worden momenteel gebruikt in de microagglutinatietest (MAT).
Alle Leptospira-serovars werden gekweekt in EMJH en hun DNA werd geëxtraheerd met behulp van een commerciële DNA-extractiekit (zie Materiaaltabel). Een genomische DNA-mix van de twaalf serovars werd gebruikt als positieve controle voor de pathogene clade van Leptospira . Als positieve controle van de Leptospira-intermediaire clade werd ook genoom-DNA van Leptospira fainei serovar Hurstbridge-stam BUT6 opgenomen, en als positieve controle voor de Leptospira-saprofytenclade werd ook genomisch DNA van Leptospira biflexa serovar Patoc-stam Patoc I opgenomen.
Negatieve controles bestonden uit een leeg plasmide, DNA van niet-verwante bacteriën (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii en Escherichia coli), en water van PCR-kwaliteit, dat diende als niet-sjablooncontrole.
Monsters van water
Twaalf proefmonsters werden verzameld met behulp van een gestratificeerde-lukrake bemonsteringsmethode van het Cuemanco Biological and Aquaculture Research Center (CIBAC) (19° 16′ 54″ N 99° 6′ 11″ W). Deze monsters werden verkregen op drie diepten: oppervlakkig, 10 en 30 cm (figuur 1A, B). De procedures voor het verzamelen van water hadden geen invloed op bedreigde of beschermde diersoorten. Elk monster werd verzameld in een steriele microcentrifugebuis van 15 ml. Om het monster te verzamelen, werd elke buis voorzichtig ondergedompeld in het water, op de geselecteerde diepte gevuld en vervolgens verzegeld. De monsters werden op 22 °C gehouden en onmiddellijk naar het laboratorium vervoerd voor verwerking.
Elk monster werd geconcentreerd door centrifugatie in steriele microcentrifugebuisjes van 1,5 ml bij 8000 x g gedurende 20 minuten bij kamertemperatuur. Deze stap werd herhaald totdat alle monsters waren geconcentreerd in één buis, die vervolgens werd gebruikt voor DNA-extractie (Figuur 1C).
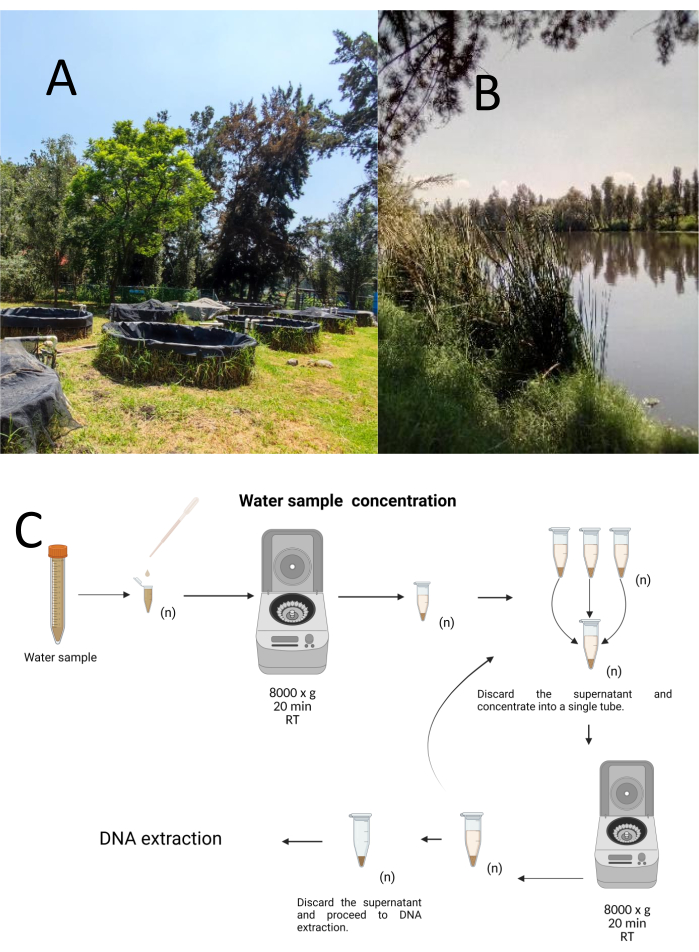
Figuur 1: Concentratie van watermonsters door centrifugatie. (A) Waterbemonsteringsvijvers en (B) Natuurlijke beken. (C) Verwerking van watermonsters op basis van centrifugatie in herhaalde stappen, zo vaak als nodig is (n). Klik hier om een grotere versie van deze figuur te bekijken.
DNA-extractie
Totaal DNA werd geïsoleerd met behulp van een in de handel verkrijgbare genomische DNA-kit volgens de instructies van de fabrikant (zie Materiaaltabel). DNA-extracties werden geëlueerd in 20 μL elutiebuffer en de DNA-concentratie werd bepaald door een UV-spectrofotometer bij 260-280 nm en bewaard bij 4 °C tot gebruik.
PCR-amplificatie
De PCR-doelen waren de 16S rRNA-, lipL41- en lipL32-genen, die DNA van het geslacht Leptospira identificeren en het onderscheid tussen de drie clades mogelijk maken: pathogeen, saprofytische en intermediairen. Zowel de primers als de sondeontwerpen waren gebaseerd op de eerdere werken van Ahmed et al., Azali et al., Bourhy et al., Weiss et al., en Branger et al.33,34,35,36,37. De volgorde van elke sonde, primer en geamplificeerd fragment wordt beschreven in tabel 1 en hun uitlijning met referentiesequenties wordt gegeven in aanvullend bestand 1, aanvullend bestand 2, aanvullend bestand 3, aanvullend bestand 4 en aanvullend bestand 5. De PCR-reagentia en thermocyclusvoorwaarden worden beschreven in het protocolgedeelte.
Amplificatieproducten werden gevisualiseerd door elektroforetische scheiding op een 1% agarosegel in TAE (40 mM Tris-base, 20 mM azijnzuur en 1 mM EDTA; pH 8,3), bij 60 V gedurende 45 minuten met ethidiumbromidedetectie, zoals weergegeven in aanvullende figuur 1. Genoom-DNA verkregen van elke serovar werd gebruikt met concentraties variërend van 6 x 106 tot 1 x 104 genomische equivalente kopieën (GEq) in elke PCR-reactie, gebaseerd op de genoomgrootte van L. interrogans (4, 691, 184 bp)38 voor pathogene Leptospira, de genoomgrootte van L. biflexa (3, 956, 088 bp)39 voor saprofytische Leptospira, en de genoomgrootte van L. fainei serovar Hurstbridge-stam BUT6 (4, 267, 324 bp) met toetredingsnummer AKWZ00000000.2.
De gevoeligheid van de sondes werd beoordeeld met DNA van elke pathogene serovar, L. biflexa serovar Patoc stam Patoc I en L. fainei serovar Hurstbridge-stam BUT6 in elk experiment. Om de specificiteit van de PCR- en dot-blot-hybridisatietest te beoordelen, werd DNA van niet-verwante bacteriën opgenomen.
Tabel 1: PCR-primers en -sondes om producten te amplificeren voor het identificeren van de pathogene, saprofyt- en intermediaire clades van Leptospira. Klik hier om deze tabel te downloaden.
Dot-blot hybridisatie-test
De techniek wordt dot-blot genoemd omdat de gaten waarin het DNA-monster wordt geplaatst een puntvorm hebben, en wanneer ze worden opgezogen om op hun plaats te worden gefixeerd door vacuümzuiging, krijgen ze deze vorm. Deze techniek is ontwikkeld door Kafatos et al.40. De techniek maakt de semi-kwantificering van Leptospira in elk PCR-positief monster mogelijk. Het protocol bestaat uit een denaturatie met NaOH 0,4 M bij kamertemperatuur, monsters met Leptospira-DNA van 30 ng tot 0,05 ng, overeenkomend met 6 x 106 tot 1 x 104 leptospiren, worden gedept op een nylon membraan met een dot-blot-apparaat met 96 putjes. Na immobilisatie wordt het DNA aan het membraan gebonden door blootstelling aan 120 mJ UV-licht. Elke DNA-sonde wordt geconjugeerd met digoxigenine-11 dUTP door een terminale transferasekatalysestap aan het 3′-uiteinde (Digoxigenine is een plantaardige steroïde verkregen uit Digitalis purpurea, gebruikt als verslaggever41). Na de strikte hybridisatie van de gelabelde DNA-sonde (50 pmol) bij de specifieke temperatuur op het doel-DNA, worden de DNA-hybriden gevisualiseerd door de chemiluminescentiereactie met het anti-digoxigenine alkalische fosfatase-antilichaam dat covalent is geconjugeerd met zijn substraat CSPD. De luminescentie wordt vastgelegd door blootstelling aan een röntgenfilm (figuur 2).
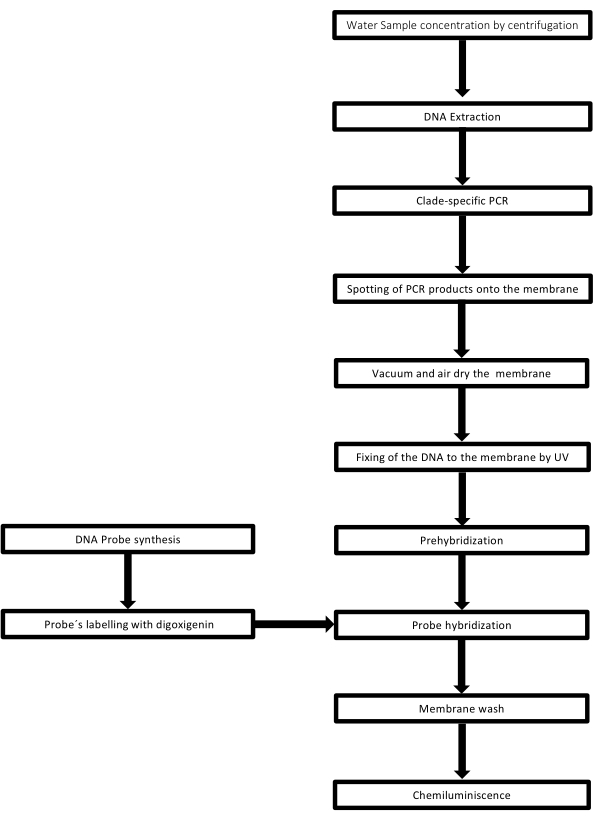
Figuur 2: Stappen van de procedure voor de PCR-dot-blot-test. Klik hier om een grotere versie van deze figuur te bekijken.
Protocol
Representative Results
Discussion
De kritieke stappen van de dot-blot-techniek omvatten (1) DNA-immobilisatie, (2) blokkering van de vrije bindingsplaatsen op het membraan met niet-homoloog DNA, (3) de complementariteit tussen de sonde en het doelfragment onder gloeiomstandigheden, (4) verwijdering van de niet-gehybridiseerde sonde en (5) de detectie van het reportermolecuul41.
De PCR-Dot-blot heeft bepaalde beperkingen, zoals het feit dat de techniek geen informatie geeft over de grootte van het gehybr…
Divulgations
The authors have nothing to disclose.
Acknowledgements
We zijn dank verschuldigd aan de Leptospira-collectie van de afdeling Microbiologie en Immunologie, Faculteit Diergeneeskunde en Zoötechniek, Nationale Autonome Universiteit van Mexico. We zijn dankbaar voor de genereuze donatie van de referentie Leptospira-stammen ; Leptospira fainei serovar Hurstbridge stam BUT6 en Leptospira biflexa serovar Patoc stam Patoc I aan Dr. Alejandro de la Peña Moctezuma. Wij danken Dr. José Antonio Ocampo Cervantes, de CIBAC-coördinator, en het personeel voor hun logistieke steun. EDT maakte deel uit van het Terminal Project-programma voor niet-gegradueerde studenten van de Metropolitan Autonomous University-Campus Cuajimalpa. We erkennen de Biorender.com software voor het maken van figuren 1 en 3 tot en met 9.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
References
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

