Réaction en chaîne par polymérase et hybridation par transfert de points pour la détection de leptospires dans des échantillons d’eau
Summary
Dans cette étude, une application dot-blot a été conçue pour détecter Leptospira dans les trois clades principaux dans des échantillons d’eau. Cette méthode permet d’identifier des quantités minimales d’ADN spécifiquement ciblées par une sonde marquée à la digoxigénine, facilement détectées par un anticorps anti-digoxigénine. Cette approche est un outil précieux et satisfaisant à des fins de dépistage.
Abstract
Le dot-blot est une technique simple, rapide, sensible et polyvalente qui permet d’identifier des quantités minimales d’ADN spécifiquement ciblées par hybridation par sonde en présence d’ADN porteur. Il est basé sur le transfert d’une quantité connue d’ADN sur un support solide inerte, tel qu’une membrane en nylon, à l’aide de l’appareil dot-blot et sans séparation électrophorétique. Les membranes en nylon ont l’avantage d’avoir une capacité de liaison élevée des acides nucléiques (400 μg/cm2), une résistance élevée et une charge positive ou neutre. La sonde utilisée est un fragment d’ADNsb très spécifique de 18 à 20 bases de long marqué à la digoxigénine (DIG). La sonde se conjuguera avec l’ADN de Leptospira . Une fois que la sonde s’est hybridée avec l’ADN cible, elle est détectée par un anticorps anti-digoxigénine, ce qui permet sa détection facile grâce à ses émissions révélées dans un film radiographique. Les points avec une émission correspondront aux fragments d’ADN d’intérêt. Cette méthode utilise le marquage non isotopique de la sonde, qui peut avoir une demi-vie très longue. L’inconvénient de cet immuno-marqueur standard est une sensibilité plus faible que celle des sondes isotopiques. Néanmoins, elle est atténuée par le couplage de la réaction en chaîne par polymérase (PCR) et des tests dot-blot. Cette approche permet l’enrichissement de la séquence cible et sa détection. De plus, il peut être utilisé comme une application quantitative par rapport à une dilution en série d’un étalon bien connu. Une application dot-blot pour détecter Leptospira dans les trois clades principaux dans des échantillons d’eau est présentée ici. Cette méthodologie peut être appliquée à de grandes quantités d’eau une fois qu’elles ont été concentrées par centrifugation pour fournir des preuves de la présence d’ADN leptospiralé. Il s’agit d’un outil précieux et satisfaisant à des fins de dépistage général, et il peut être utilisé pour d’autres bactéries non cultivables qui peuvent être présentes dans l’eau, améliorant ainsi la compréhension de l’écosystème.
Introduction
La leptospirose chez l’homme provient principalement de sources environnementales 1,2. La présence de Leptospira dans les lacs, les rivières et les ruisseaux est un indicateur de la transmission de la leptospirose chez les animaux sauvages et les animaux domestiques et de production qui peuvent éventuellement entrer en contact avec ces plans d’eau 1,3,4. De plus, Leptospira a été identifié dans des sources non naturelles, notamment dans les eaux usées, l’eau stagnante et l’eau du robinet 5,6.
Leptospira est une bactérie distribuée dans le monde entier 7,8, et le rôle de l’environnement dans sa préservation et sa transmission a été bien reconnu. Leptospira peut survivre dans l’eau potable sous un pH variable et des minéraux9, et dans des plans d’eau naturels1. Il peut également survivre pendant de longues périodes dans l’eau distillée10, et sous un pH constant (7,8), il peut survivre jusqu’à 152 jours11. De plus, Leptospira peut interagir dans des consortiums bactériens pour survivre à des conditions difficiles12,13. Il peut faire partie de biofilms en eau douce avec Azospirillum et Sphingomonas et est même capable de croître et de supporter des températures supérieures à 49 °C14,15. Il peut également se multiplier dans un sol gorgé d’eau et rester viable jusqu’à 379 jours16, préservant sa capacité à provoquer la maladie jusqu’à un an17,18. Cependant, on sait peu de choses sur l’écologie des masses d’eau et sur la façon dont elle y est distribuée.
Depuis sa découverte, l’étude du genre Leptospira s’est basée sur des tests sérologiques. Ce n’est qu’au cours du siècle actuel que les techniques moléculaires sont devenues plus répandues dans l’étude de ce spirochète. Le dot-blot a été à peine utilisé pour son identification à l’aide (1) d’une sonde isotopique basée sur l’ARNr 16S et sur une répétition de séquence inter-simple (ISSR)19,20, (2) en tant qu’immunodosage basé sur le nanogold pour la leptospirose humaine appliquée à l’urine21, ou (3) en tant que test basé sur des anticorps pour des échantillons d’urine bovine22. La technique est tombée en désuétude car elle était à l’origine basée sur des sondes isotopiques. Cependant, il s’agit d’une technique bien connue qui, associée à la PCR, donne de meilleurs résultats et est considérée comme sûre en raison de l’utilisation de sondes non isotopiques. La PCR joue un rôle crucial dans l’enrichissement de l’ADN de Leptospira en amplifiant un fragment d’ADN spécifique qui peut être trouvé à l’état de traces dans un échantillon. Au cours de chaque cycle de PCR, la quantité du fragment d’ADN ciblé est doublée dans la réaction. À la fin de la réaction, l’amplicon a été multiplié par un facteur de plus d’un million23. Le produit amplifié par PCR, souvent non visible dans l’électrophorèse de l’agarose, devient visible par hybridation spécifique avec une sonde marquée DIG dans le dot-blot 24,25,26.
La technique dot-blot est simple, robuste et adaptée à de nombreux échantillons, ce qui la rend accessible aux laboratoires disposant de ressources limitées. Il a été utilisé dans une variété d’études sur les bactéries, y compris (1) les bactéries buccales27, (2) d’autres types d’échantillons tels que les aliments et les matières fécales28, et (3) l’identification de bactéries incultivables29, souvent en accord avec d’autres techniques moléculaires. Parmi les avantages offerts par la technique dot-blot figurent : (1) La membrane a une capacité de liaison élevée, capable de lier plus de 200 μg/cm2 d’acides nucléiques et jusqu’à 400 μg/cm2 ; (2) Les résultats Dot-blot peuvent être interprétés visuellement sans nécessiter d’équipement spécial, et (3) ils peuvent être stockés pendant des années à température ambiante (RT).
Le genre Leptospira a été classé en clades pathogènes, intermédiaires et saprophytes30,31. La distinction entre ces clades peut être obtenue sur la base de gènes spécifiques tels que lipL41, lipL32 et l’ARNr 16S. LipL32 est présent dans les clades pathogènes et présente une grande sensibilité dans divers outils sérologiques et moléculaires, alors qu’il est absent chez les espèces de saprophytes21. Le gène ménager lipL41 est connu pour son expression stable et utilisé dans les techniques moléculaires32, tandis que le gène de l’ARNr 16S est utilisé pour leur classification.
Cette méthodologie peut être appliquée à de grands volumes d’eau une fois qu’ils ont été concentrés par centrifugation. Il permet d’évaluer différents points et profondeurs au sein d’un plan d’eau afin de détecter la présence d’ADN leptospiralé et du clade auquel il appartient. Cet outil est précieux à des fins de dépistage écologique et général et peut également être utilisé pour détecter d’autres bactéries non cultivables qui peuvent être présentes dans l’eau.
De plus, les tests PCR et dot-blot sont techniquement et économiquement abordables pour un large éventail de laboratoires, même ceux qui ne disposent pas d’équipements sophistiqués ou coûteux. Cette étude vise à appliquer le dot-blot à base de digoxigénine pour l’identification des trois clades de Leptospira dans des échantillons d’eau prélevés dans des plans d’eau naturels.
Souches bactériennes
Douze sérovars de Leptospira (Autumnalis, Bataviae, Bratislava, Canicola, Celledoni, Grippothyphosa, Hardjoprajitno, Icterohaemorrhagiae, Pomona, Pyrogenes, Tarassovi et Wolffi) ont été inclus dans cette étude. Ces sérotypes font partie de la collection du Département de microbiologie et d’immunologie de la Faculté de médecine vétérinaire et de zootechnie de l’Université nationale autonome du Mexique, et ils sont actuellement utilisés dans le test de microagglutination (MAT).
Tous les sérotypes de Leptospira ont été cultivés dans l’EMJH, et leur ADN a été extrait à l’aide d’une trousse d’extraction d’ADN commerciale (voir le tableau des matériaux). Un mélange d’ADN génomique des douze sérovars a été utilisé comme contrôle positif pour le clade pathogène de Leptospira . En tant que contrôle positif du clade intermédiaire de Leptospira , l’ADN génomique de la souche BUT6 du sérotype Hurstbridge de Leptospira fainei a été inclus, et en tant que contrôle positif du clade des saprophytes de Leptospira , l’ADN génomique de la sérotype de Leptospira biflexa de la souche Patoc I a également été inclus.
Les témoins négatifs étaient constitués d’un plasmide vide, d’ADN provenant de bactéries non apparentées (Ureaplasma urealyticum, Staphylococcus aureus, Brucella abortus, Salmonella typhimurium, Shigella boydii, Klebsiella pneumoniae, Acinetobacter baumannii et Escherichia coli) et d’eau de qualité PCR, qui a servi de témoin non matriciel.
Échantillons d’eau
Douze échantillons choisis ont été prélevés à l’aide d’une méthode d’échantillonnage stratifié-aléatoire du Centre de recherche biologique et aquacole de Cuemanco (CIBAC) (19° 16′ 54 » N 99° 6′ 11 » O). Ces échantillons ont été obtenus à trois profondeurs : superficielle, 10 et 30 cm (figures 1A, B). Les procédures de collecte de l’eau n’ont eu aucun impact sur les espèces menacées ou protégées. Chaque échantillon a été prélevé dans un tube à microcentrifuger stérile de 15 ml. Pour recueillir l’échantillon, chaque tube a été doucement immergé dans l’eau, rempli à la profondeur sélectionnée, puis scellé. Les échantillons ont été maintenus à 22 °C et rapidement transportés au laboratoire pour être traités.
Chaque échantillon a été concentré par centrifugation dans des tubes de microcentrifugation stériles de 1,5 mL à 8000 x g pendant 20 min à température ambiante. Cette étape a été répétée jusqu’à ce que tous les échantillons soient concentrés dans un seul tube, qui a ensuite été utilisé pour l’extraction de l’ADN (figure 1C).
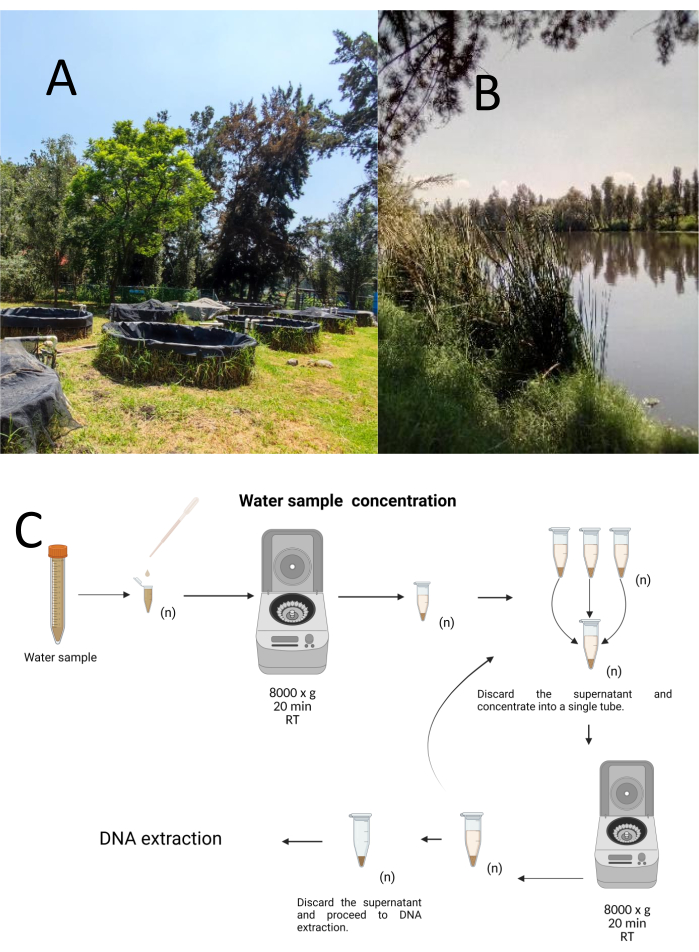
Figure 1 : Concentration des échantillons d’eau par centrifugation. (A) Bassins d’échantillonnage d’eau, et (B) Cours d’eau naturels. (C) Traitement d’échantillons d’eau par centrifugation en plusieurs étapes autant de fois que nécessaire (n). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Extraction de l’ADN
L’ADN total a été isolé à l’aide d’un kit d’ADN génomique commercial conformément aux instructions du fabricant (voir le tableau des matériaux). Les extractions d’ADN ont été éluées dans 20 μL de tampon d’élution, et la concentration d’ADN a été déterminée par un spectrophotomètre UV à 260-280 nm, et stockée à 4 °C jusqu’à l’utilisation.
Amplification par PCR
Les cibles de la PCR étaient les 16 gènes de l’ARNrS, lipL41 et lipL32, qui identifient l’ADN du genre Leptospira et permettent de distinguer les trois clades : pathogène, saprophyte et intermédiaire. Les conceptions des amorces et des sondes étaient basées sur les travaux antérieurs d’Ahmed et al., Azali et al., Bourhy et al., Weiss et al., et Branger et al.33,34,35,36,37. La séquence de chaque sonde, amorce et fragment amplifié est décrite dans le Tableau 1, et leur alignement avec les séquences de référence est fourni dans le Fichier supplémentaire 1, le Fichier supplémentaire 2, le Fichier supplémentaire 3, le Fichier supplémentaire 4 et le Fichier supplémentaire 5. Les réactifs de PCR et les conditions de thermocyclage sont décrits dans la section protocole.
Les produits d’amplification ont été visualisés par séparation électrophorétique sur un gel d’agarose à 1 % dans un TAE (40 mM de base Tris, 20 mM d’acide acétique et 1 mM d’EDTA ; pH 8,3), à 60 V pendant 45 min avec détection au bromure d’éthidium, comme le montre la figure supplémentaire 1. L’ADN génomique obtenu à partir de chaque sérotype a été utilisé avec des concentrations allant de 6 x 106 à 1 x 104 copies équivalentes génomiques (GEq) dans chaque réaction PCR, sur la base de la taille du génome de L. interrogans (4 691, 184 pb)38 pour Leptospira pathogène, de la taille du génome de L. biflexa (3 956, 088 pb)39 pour Leptospira saprophyte, et la taille du génome de la souche BUT6 du sérovar de L. fainei Hurstbridge (4 267, 324 pb) avec le numéro d’accession AKWZ00000000,2.
La sensibilité des sondes a été évaluée à l’aide de l’ADN de chaque sérotype pathogène, de la souche sérovar Patoc I de L. biflexa et de la souche BUT6 de Hurstbridge sérovar de L. fainei . Pour évaluer la spécificité de la PCR et de l’essai d’hybridation par transfert de points, on a inclus de l’ADN de bactéries non apparentées.
Tableau 1 : Amorces et sondes PCR pour amplifier les produits d’identification des clades pathogènes, saprophytes et intermédiaires de Leptospira. Veuillez cliquer ici pour télécharger ce tableau.
Essai d’hybridation dot-blot
La technique est appelée dot-blot car les trous dans lesquels l’échantillon d’ADN est placé ont une forme de point, et lorsqu’ils sont aspirés pour être fixés en place par aspiration sous vide, ils acquièrent cette forme. Cette technique a été mise au point par Kafatos et al.40. La technique permet la semi-quantification de Leptospira dans chaque échantillon positif à la PCR. Le protocole consiste en une dénaturation avec du NaOH 0,4 M à température ambiante, des échantillons avec de l’ADN de Leptospira de 30 ng à 0,05 ng, correspondant à 6 x 106 à 1 x 104 leptospires, sont tamponnés sur une membrane en nylon avec un appareil de transfert de points de 96 puits. Après l’immobilisation, l’ADN est lié à la membrane par l’exposition à une lumière UV de 120 mJ. Chaque sonde d’ADN est conjuguée avec la digoxigénine-11 dUTP par une étape de catalyse terminale de la transférase à l’extrémité 3′ (la digoxigénine est un stéroïde végétal obtenu à partir de Digitalis purpurea, utilisé comme rapporteur41). Suite à l’hybridation rigoureuse de la sonde d’ADN marquée (50 pmol) à la température spécifique sur l’ADN cible, les hybrides d’ADN sont visualisés par la réaction de chimiluminescence avec l’anticorps anti-digoxigénine phosphatase alcaline conjugué de manière covalente avec son substrat CSPD. La luminescence est capturée par l’exposition à un film radiographique (figure 2).
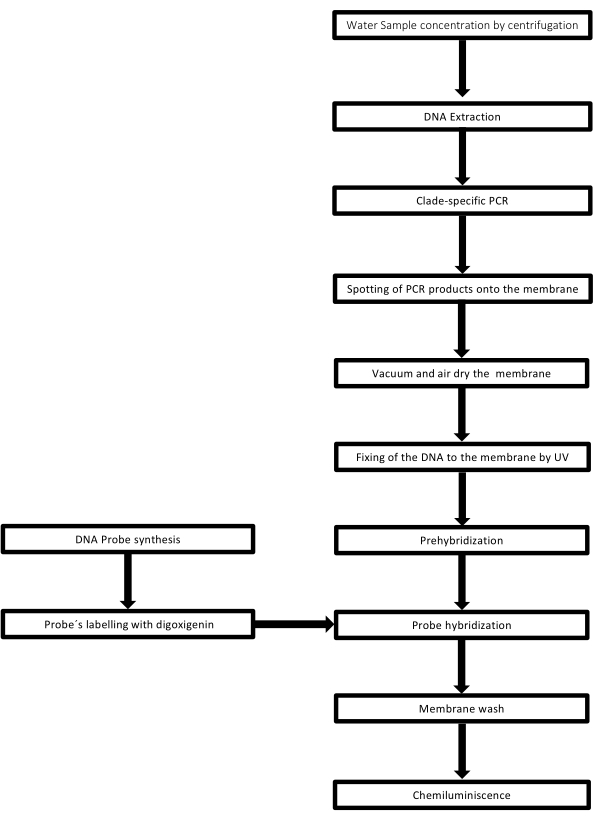
Figure 2 : Étapes de la procédure pour le test PCR-dot-blot. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocol
Representative Results
Discussion
Les étapes critiques de la technique dot-blot comprennent (1) l’immobilisation de l’ADN, (2) le blocage des sites de liaison libres sur la membrane avec de l’ADN non homologue, (3) la complémentarité entre la sonde et le fragment cible dans des conditions de recuit, (4) l’élimination de la sonde non hybridée et (5) la détection de la molécule rapporteure41.
La PCR-Dot-blot présente certaines limites, telles que la technique ne fournit pas d’information…
Divulgations
The authors have nothing to disclose.
Acknowledgements
Nous sommes redevables à la collection Leptospira du Département de Microbiologie et d’Immunologie, Faculté de Médecine Vétérinaire et de Zootechnie, Université Nationale Autonome du Mexique. Nous sommes reconnaissants pour le généreux don des souches de référence Leptospira ; Leptospira fainei sérovar Hurstbridge souche BUT6 et Leptospira biflexa sérovar Patoc souche Patoc I au Dr Alejandro de la Peña Moctezuma. Nous remercions le Dr José Antonio Ocampo Cervantes, le coordinateur du CIBAC, et le personnel pour leur soutien logistique. EDT s’inscrivait dans le cadre du programme Terminal Project pour les étudiants de premier cycle de l’Université autonome métropolitaine-Campus Cuajimalpa. Nous remercions le logiciel Biorender.com pour la création des figures 1 et 3 à 9.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
References
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

