تفاعل البلمرة المتسلسل وتهجين اللطخة النقطية للكشف عن Leptospira في عينات المياه
Summary
في هذه الدراسة ، تم تصميم تطبيق نقطي للكشف عن Leptospira من الفصائل الرئيسية الثلاثة في عينات المياه. تسمح هذه الطريقة بتحديد الحد الأدنى من كميات الحمض النووي المستهدفة على وجه التحديد بواسطة مسبار يحمل علامة ديجوكسيجينين ، ويمكن اكتشافه بسهولة بواسطة جسم مضاد للديجوكسيجينين. هذا النهج هو أداة قيمة ومرضية لأغراض الفرز.
Abstract
اللطخة النقطية هي تقنية بسيطة وسريعة وحساسة ومتعددة الاستخدامات تتيح تحديد كميات قليلة من الحمض النووي المستهدف على وجه التحديد عن طريق تهجين المسبار في وجود الحمض النووي الناقل. يعتمد على نقل كمية معروفة من الحمض النووي إلى دعامة صلبة خاملة ، مثل غشاء النايلون ، باستخدام جهاز البقعة النقطية وبدون فصل كهربائي. تتميز أغشية النايلون بقدرة ربط الحمض النووي العالية (400 ميكروغرام / سم2) ، والقوة العالية ، وهي مشحونة بشكل إيجابي أو محايد. المسبار المستخدم عبارة عن جزء من ssDNA محدد للغاية من 18 إلى 20 قاعدة موسومة بالديجوكسيجينين (DIG). سوف يترافق المسبار مع الحمض النووي Leptospira . بمجرد تهجين المسبار مع الحمض النووي المستهدف ، يتم اكتشافه بواسطة جسم مضاد مضاد للديجوكسيجينين ، مما يسمح باكتشافه بسهولة من خلال انبعاثاته التي تم الكشف عنها في فيلم الأشعة السينية. سوف تتوافق النقاط ذات الانبعاث مع شظايا الحمض النووي ذات الأهمية. تستخدم هذه الطريقة وضع العلامات غير النظيرية للمسبار ، والتي قد يكون لها عمر نصف طويل جدا. عيب هذا الملصق المناعي القياسي هو حساسية أقل من تحقيقات النظائر. ومع ذلك ، يتم تخفيفه عن طريق اقتران تفاعل البوليميراز المتسلسل (PCR) ومقايسات البقع النقطية. يتيح هذا النهج إثراء التسلسل المستهدف واكتشافه. بالإضافة إلى ذلك ، يمكن استخدامه كتطبيق كمي عند مقارنته بالتخفيف التسلسلي لمعيار معروف. يتم تقديم تطبيق لطخة نقطية للكشف عن Leptospira من الكتل الرئيسية الثلاثة في عينات المياه هنا. يمكن تطبيق هذه المنهجية على كميات كبيرة من المياه بمجرد تركيزها عن طريق الطرد المركزي لتقديم دليل على وجود الحمض النووي Leptospiral. هذه أداة قيمة ومرضية لأغراض الفحص العام ، ويمكن استخدامها للبكتيريا الأخرى غير القابلة للزراعة التي قد تكون موجودة في الماء ، مما يعزز فهم النظام البيئي.
Introduction
داء البريميات في البشر ينشأ أساسا من مصادر بيئية1،2. يعد وجود Leptospira في البحيرات والأنهار والجداول مؤشرا على انتقال داء البريميات بين الحياة البرية الأليفة والمنتجة التي قد تتلامس في النهاية مع هذه المسطحات المائية1،3،4. علاوة على ذلك ، تم تحديد Leptospira في مصادر غير طبيعية ، بما في ذلك مياه الصرف الصحي والراكدة ومياهالصنبور 5,6.
Leptospira هي بكتيريا موزعة في جميع أنحاء العالم 7,8 ، وقد تم التعرف على دور البيئة في الحفاظ عليها ونقلها بشكل جيد. يمكن أن تعيش Leptospira في مياه الشرب تحت درجة الحموضة والمعادنالمتغيرة 9 ، وفي المسطحات المائيةالطبيعية 1. يمكنه أيضا البقاء على قيد الحياة لفترات طويلة في الماء المقطر10 ، وتحت درجة حموضة ثابتة (7.8) ، قد يعيش حتى 152 يوما11. علاوة على ذلك ، قد تتفاعل Leptospira في اتحادات بكتيرية للبقاء على قيد الحياة في الظروف القاسية12,13. قد يكون جزءا من الأغشية الحيوية في المياه العذبة مع Azospirillum و Sphingomonas ، بل إنه قادر على النمو وتحمل درجات حرارة تتجاوز 49 درجة مئوية14,15. يمكن أن تتكاثر أيضا في التربة المشبعة بالمياه وتظل قابلة للحياة لمدة تصل إلى 379 يوما16 ، مما يحافظ على قدرتها على التسبب في المرض لمدة تصل إلى عام17,18. ومع ذلك ، لا يعرف سوى القليل عن البيئة داخل المسطحات المائية وكيف يتم توزيعها داخلها.
منذ اكتشافها ، استندت دراسة جنس Leptospira إلى الاختبارات المصلية. لم يكن حتى القرن الحالي أن التقنيات الجزيئية أصبحت أكثر انتشارا في دراسة هذا spirochaete. نادرا ما تم استخدام اللطخة النقطية لتحديدها باستخدام (1) مسبار نظيري يعتمد على 16S rRNA وعلى تكرار تسلسل بسيط (ISSR) 19،20 ، (2) كاختبار مناعي قائم على الذهب النانوي لداء البريميات البشري المطبق على البول21 ، أو (3) كمقايسة قائمة على الأجسام المضادة لعينات بول الأبقار22. سقطت هذه التقنية في حالة إهمال لأنها كانت تستند في الأصل إلى تحقيقات نظيرية. ومع ذلك ، فهي تقنية معروفة جيدا ، إلى جانب تفاعل البوليميراز المتسلسل ، تسفر عن نتائج محسنة ، وتعتبر آمنة بسبب استخدام المجسات غير النظيرية. يلعب تفاعل البوليميراز المتسلسل دورا حاسما في إثراء الحمض النووي Leptospira عن طريق تضخيم جزء معين من الحمض النووي يمكن العثور عليه بكميات ضئيلة في العينة. خلال كل دورة PCR ، تتضاعف كمية جزء الحمض النووي المستهدف في التفاعل. في نهاية التفاعل ، تم ضرب amplicon بعامل أكثر منمليون 23. يصبح المنتج الذي يتم تضخيمه بواسطة تفاعل البوليميراز المتسلسل ، والذي غالبا ما يكون غير مرئي في رحلان الأغاروز الكهربائي ، مرئيا من خلال تهجين محدد باستخدام مسبار يحمل علامة DIG في البقعةالنقطية 24،25،26.
تقنية dot-blot بسيطة وقوية ومناسبة للعديد من العينات ، مما يجعلها في متناول المختبرات ذات الموارد المحدودة. تم استخدامه في مجموعة متنوعة من دراسات البكتيريا ، بما في ذلك (1) البكتيريا الفموية27 ، (2) أنواع العينات الأخرى مثل الطعام والبراز28 ، و (3) تحديد البكتيريا غير القابلة للزراعة29 ، غالبا بالاتفاق مع التقنيات الجزيئية الأخرى. من بين المزايا التي توفرها تقنية dot-blot ما يلي: (1) يتمتع الغشاء بقدرة ربط عالية ، قادرة على ربط أكثر من 200 ميكروغرام / سم2 من الأحماض النووية وما يصل إلى 400 ميكروغرام / سم2 ؛ (2) يمكن تفسير نتائج البقع النقطية بصريا دون الحاجة إلى معدات خاصة ، و (3) يمكن تخزينها بسهولة لسنوات في درجة حرارة الغرفة (RT).
تم تصنيف جنس Leptospira إلى مجموعات مسببة للأمراض ومتوسطة ورمية30,31. يمكن تحقيق التمييز بين هذه الفصائل بناء على جينات محددة مثل lipL41 و lipL32 و 16S rRNA. LipL32 موجود في الكتل المسببة للأمراض ويظهر حساسية عالية في مختلف الأدوات المصلية والجزيئية ، في حين أنه غائب في الأنواع الرمية21. يعرف جين التدبير المنزلي lipL41 بتعبيره المستقر ويستخدم في التقنيات الجزيئية32 ، بينما يستخدم جين 16S rRNA لتصنيفها.
يمكن تطبيق هذه المنهجية على كميات كبيرة من المياه بمجرد تركيزها بواسطة الطرد المركزي. يسمح بتقييم النقاط والأعماق المختلفة داخل الجسم المائي للكشف عن وجود الحمض النووي البريمي والمجموعة التي ينتمي إليها. هذه الأداة ذات قيمة لكل من أغراض الفحص البيئي والعام ويمكن استخدامها أيضا للكشف عن البكتيريا الأخرى غير القابلة للزراعة التي قد تكون موجودة في الماء.
بالإضافة إلى ذلك ، فإن فحوصات تفاعل البوليميراز المتسلسل واللطخة النقطية ميسورة التكلفة تقنيا واقتصاديا لمجموعة واسعة من المختبرات ، حتى تلك التي تفتقر إلى معدات متطورة أو باهظة الثمن. تهدف هذه الدراسة إلى تطبيق اللطخة النقطية القائمة على الديغوكسيجينين لتحديد مجموعات Leptospira الثلاثة في عينات المياه التي تم جمعها من المسطحات المائية الطبيعية.
السلالات البكتيرية
تم تضمين اثني عشر Leptospira serovars (Autumnalis ، Bataviae ، Bratislava ، Canicola ، Celledoni ، Grippothyphosa ، Hardjoprajitno ، Icterohaemorrhagiae ، Pomona ، Pyrogenes ، Tarassovi ، و Wolffi) في هذه الدراسة. هذه المصل هي جزء من المجموعة في قسم علم الأحياء الدقيقة والمناعة ، كلية الطب البيطري وعلم ، الجامعة الوطنية المستقلة في المكسيك ، وتستخدم حاليا في اختبار التراص الدقيق (MAT).
تم استزراع جميع Leptospira serovars في EMJH ، وتم استخراج الحمض النووي الخاص بهم باستخدام مجموعة استخراج الحمض النووي التجارية (انظر جدول المواد). تم استخدام مزيج الحمض النووي الجينومي من المصل الاثني عشر كعنصر تحكم إيجابي للزمرة المسببة للأمراض Leptospira . كعنصر تحكم إيجابي في مجموعة Leptospira الوسيطة ، تم تضمين الحمض النووي الجينومي من سلالة Leptospira fainei serovar Hurstbridge BUT6 ، وكعنصر تحكم إيجابي في مجموعة Leptospira saprophyte ، تم أيضا تضمين الحمض النووي الجينومي لسلالة Leptospira biflexa serovar Patoc Patoc I.
تألفت الضوابط السلبية من بلازميد فارغ ، وحمض نووي من بكتيريا غير ذات صلة (Ureaplasma urealyticum ، والمكورات العنقودية الذهبية ، والبروسيلا المجهضة ، والسالمونيلا التيفية ، والشيغيلة البوجيدية ، والكلبسيلة الرئوية ، والراكدة البوماني ، والإشريكية القولونية) ، والمياه من درجة تفاعل البوليميراز المتسلسل ، والتي كانت بمثابة تحكم غير قالب.
عينات المياه
تم جمع اثنتي عشرة عينة تجريبية باستخدام طريقة أخذ العينات الطبقية العشوائية من مركز بحوث Cuemanco البيولوجي وتربية الأحياء المائية (CIBAC) (19 ° 16′ 54″ شمالا 99 ° 6′ 11″ غربا). تم الحصول على هذه العينات على ثلاثة أعماق: سطحية ، 10 ، و 30 سم (الشكل 1 أ ، ب). لم تؤثر إجراءات جمع المياه على أي أنواع مهددة بالانقراض أو محمية. تم جمع كل عينة في أنبوب طرد مركزي معقم سعة 15 مل. لجمع العينة ، تم غمر كل أنبوب برفق في الماء ، وملئه بالعمق المحدد ، ثم إغلاقه. تم الاحتفاظ بالعينات عند 22 درجة مئوية ونقلها على الفور إلى المختبر لمعالجتها.
تم تركيز كل عينة عن طريق الطرد المركزي في أنابيب طرد مركزي معقمة سعة 1.5 مل عند 8000 × جم لمدة 20 دقيقة في درجة حرارة الغرفة. تكررت هذه الخطوة حتى تم تركيز جميع العينات في أنبوب واحد ، والذي تم استخدامه بعد ذلك لاستخراج الحمض النووي (الشكل 1C).
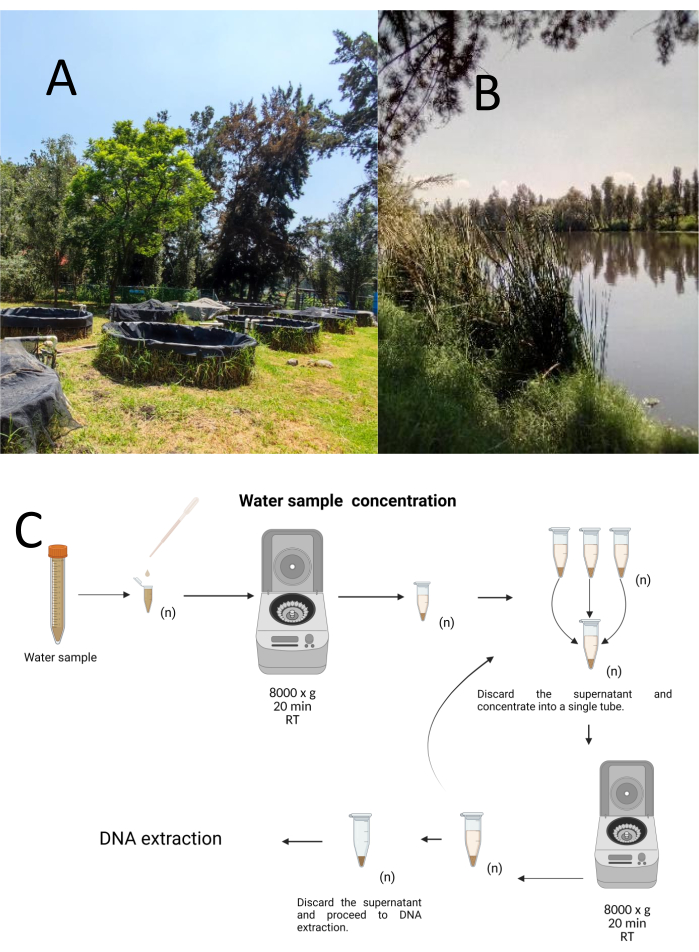
الشكل 1: تركيز عينات المياه بالطرد المركزي. أ: أحواض أخذ عينات المياه، ب: الجداول الطبيعية. (ج) معالجة عينات المياه القائمة على الطرد المركزي في خطوات متكررة عدة مرات حسب الحاجة (ن). يرجى النقر هنا لعرض نسخة أكبر من هذا الرقم.
استخراج الحمض النووي
تم عزل إجمالي الحمض النووي باستخدام مجموعة الحمض النووي الجينومي التجارية وفقا لتعليمات الشركة المصنعة (انظر جدول المواد). تم استخلاص استخلاص الحمض النووي في 20 ميكرولتر من محلول الشطف ، وتم تحديد تركيز الحمض النووي بواسطة مقياس الطيف الضوئي للأشعة فوق البنفسجية عند 260-280 نانومتر ، وتخزينه عند 4 درجات مئوية حتى الاستخدام.
تضخيم PCR
كانت أهداف تفاعل البوليميراز المتسلسل هي جينات 16S rRNA و lipL41 و lipL32 ، والتي تحدد الحمض النووي من جنس Leptospira وتسمح بالتمييز بين المجموعات الثلاث: المسببة للأمراض ، والرمية ، والمتوسطة. استندت كل من البادئات وتصميمات المسبار إلى الأعمال السابقة لأحمد وآخرون ، وأزالي وآخرون ، وبورهي وآخرون ، وفايس وآخرون ، وبرانجر وآخرون 33،34،35،36،37. يتم وصف تسلسل كل مسبار وتمهيد وجزء مضخم في الجدول 1 ، ويتم توفير مواءمتها مع التسلسلات المرجعية في الملف التكميلي 1 والملف التكميلي 2 والملف التكميلي 3 والملف التكميلي 4 والملف التكميلي 5. يتم وصف كواشف تفاعل البوليميراز المتسلسل وظروف التدوير الحراري في قسم البروتوكول.
تم تصور منتجات التضخيم عن طريق الفصل الكهربائي على هلام أغاروز 1٪ في TAE (قاعدة 40 mM Tris ، و 20 mM حمض الخليك ، و 1 mM EDTA ؛ درجة الحموضة 8.3) ، عند 60 فولت لمدة 45 دقيقة مع الكشف عن بروميد الإيثيديوم ، كما هو موضح في الشكل التكميلي 1. تم استخدام الحمض النووي الجينومي الذي تم الحصول عليه من كل مصل بتركيزات تتراوح من 6 × 106 إلى 1 × 104 نسخ مكافئة جينومية (GEq) في كل تفاعل تفاعل البوليميراز المتسلسل ، بناء على حجم جينوم L. interrogans (4 ، 691 ، 184 bp)38 ل Leptospira الممرضة ، حجم الجينوم ل L. biflexa (3 ، 956 ، 088 bp)39 ل Leptospira الرمية ، وحجم الجينوم لسلالة L. fainei serovar Hurstbridge BUT6 (4،267،324 bp) مع رقم الانضمام AKWZ00000000.2.
تم تقييم حساسية المجسات باستخدام الحمض النووي من كل مصل ممرض ، سلالة L. biflexa serovar Patoc Patoc I ، وسلالة L. fainei serovar Hurstbridge BUT6 في كل تجربة. لتقييم خصوصية اختبار PCR وتهجين البقع النقطية ، تم تضمين الحمض النووي من البكتيريا غير ذات الصلة.
الجدول 1: بادئات ومجسات تفاعل البوليميراز المتسلسل لتضخيم المنتجات لتحديد الكتل المسببة للأمراض ، والخلايا الرمية ، والوسيطة من Leptospira. الرجاء الضغط هنا لتنزيل هذا الجدول.
مقايسة تهجين البقعة النقطية
تسمى هذه التقنية اللطخة النقطية لأن الثقوب التي توضع فيها عينة الحمض النووي لها شكل نقطة ، وعندما يتم امتصاصها لتثبيتها في مكانها عن طريق الشفط الفراغي ، فإنها تكتسب هذا الشكل. تم تطوير هذه التقنية بواسطة Kafatos et al.40. تسمح هذه التقنية بالقياس شبه الكمي لليبتوسبيرا في كل عينة إيجابية لتفاعل البوليميراز المتسلسل. يتكون البروتوكول من تمسخ مع NaOH 0.4 M في درجة حرارة الغرفة ، والعينات التي تحتوي على Leptospira DNA من 30 نانوغرام إلى 0.05 نانوغرام ، المقابلة ل 6 × 106 إلى 1 × 104 leptospires ، يتم مسحها على غشاء نايلون بجهاز نقطية 96 بئرا. بعد التثبيت ، يرتبط الحمض النووي بالغشاء عن طريق التعرض لضوء الأشعة فوق البنفسجية 120 مللي جول. يتم اقتران كل مسبار DNA مع digoxigenin-11 dUTP بواسطة خطوة تحفيز ترانسفيراز طرفية في نهاية 3 ‘(Digoxigenin هو ستيرويد نباتي تم الحصول عليه من Digitalis purpurea ، يستخدم كمراسل41). بعد التهجين الصارم لمسبار الحمض النووي المسمى (50 pmol) عند درجة حرارة محددة على الحمض النووي المستهدف ، يتم تصور هجين الحمض النووي من خلال تفاعل التلألؤ الكيميائي مع الجسم المضاد للفوسفاتيز القلوي المضاد للديجوكسيجينين المترافق تساهميا مع الركيزة CSPD. يتم التقاط التلألؤ عن طريق التعرض لفيلم الأشعة السينية (الشكل 2).
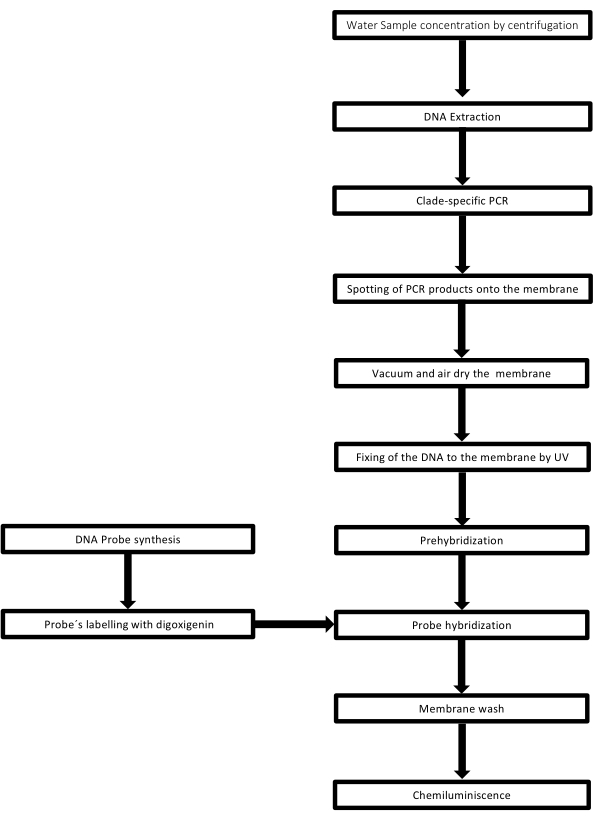
الشكل 2: خطوات إجراء فحص PCR-dot-blot. الرجاء الضغط هنا لعرض نسخة أكبر من هذا الشكل.
Protocol
Representative Results
Discussion
تشمل الخطوات الحاسمة لتقنية اللطخة النقطية (1) تجميد الحمض النووي ، (2) حجب مواقع الارتباط الحرة على الغشاء بالحمض النووي غير المتماثل ، (3) التكامل بين المسبار والجزء المستهدف في ظل ظروف التلدين ، (4) إزالة المسبار غير المهجن ، و (5) الكشف عن جزيء المراسل41.
يحتوي PCR-Dot…
Divulgations
The authors have nothing to disclose.
Acknowledgements
نحن مدينون لمجموعة Leptospira التابعة لقسم علم الأحياء الدقيقة والمناعة ، كلية الطب البيطري وعلم ، الجامعة الوطنية المستقلة في المكسيك. نحن ممتنون للتبرع السخي من سلالات Leptospira المرجعية. سلالة Leptospira fainei serovar Hurstbridge BUT6 و Leptospira biflexa serovar Patoc سلالة باتوك الأول للدكتور أليخاندرو دي لا بينيا موكتيزوما. نشكر الدكتور خوسيه أنطونيو أوكامبو سرفانتس ، منسق CIBAC ، والموظفين على دعمهم اللوجستي. كانت EDT في إطار برنامج مشروع المحطة الطرفية للطلاب الجامعيين في جامعة متروبوليتان المستقلة – الحرم الجامعي كواجيمالبا. ونقر بالبرنامج Biorender.com لإنشاء الأشكال 1 ومن 3 إلى 9.
Materials
| REAGENTS | |||
| Purelink DNA extraction kit | Invitrogen | K182002 | |
| Gotaq Flexi DNA Polimerase (End-Point PCR Taq polymerase kit) | Promega | M3001 | |
| Whatman filter paper, grade 1, | Merk | WHA1001325 | |
| Nylon Membranes, positively charged Roll 30cm x 3 m | Roche | 11417240001 | |
| Anti-Digoxigenin-AP, Fab fragments Sheep Polyclonal Primary-antibody | Roche | 11093274910 | |
| Medium Base EMJH | Difco | S1368JAA | |
| Leptospira Enrichment EMJH | Difco | BD 279510 | |
| Blocking Reagent | Roche | 11096176001 | |
| CSPD ready to use Disodium 3-(4-methoxyspiro {1,2-dioxetane-3,2′-(5′-chloro) tricyclo [3.3.1.13,7] decan}8-4-yl) phenyl phosphate | Merk | 11755633001 | |
| Deoxyribonucleic acid from herring sperm | Sigma Aldrich | D3159 | |
| Developer Carestream | Carestream Health Inc | GBX5158621 | |
| Digoxigenin-11-ddUTP | Roche | 11363905910 | |
| EDTA, Disodium Salt (Dihydrate) | Promega | H5032 | |
| Ficoll 400 | Sigma Aldrich | F8016 | |
| Fixer Carestream | Carestream Health Inc | GBX 5158605 | |
| Lauryl sulfate Sodium Salt (Sodium dodecyl sulfate; SDS) C12H2504SNa | Sigma Aldrich | L5750 | |
| N- Lauroylsarcosine sodium salt CH3(CH2)10CON(CH3) CH2COONa | Sigma Aldrich | L-9150 | It is an anionic surfactant |
| Polivinylpyrrolidone (PVP-40) | Sigma Aldrich | PVP40 | |
| Polyethylene glycol Sorbitan monolaurate (Tween 20) | Sigma Aldrich | 9005-64-5 | |
| Sodium Chloride (NaCl) | Sigma Aldrich | 7647-14-5 | |
| Sodium dodecyl sulfate (SDS) | Sigma Aldrich | 151-21-3 | |
| Sodium hydroxide (NaOH) | Sigma Aldrich | 1310-73-2 | |
| Sodium phosphate dibasic (NaH2PO4) | Sigma-Aldrich | 7558-79-4 | |
| Terminal transferase, recombinant | Roche | 3289869103 | |
| Tris hydrochloride (Tris HCl) | Sigma-Aldrich | 1185-53-1 | |
| SSPE 20X | Sigma-Aldrich | S2015-1L | It can be Home-made following Supplementary File 6 |
| Primers | Sigma-Aldrich | On demand | Follow table 1 |
| Probes | Sigma-Aldrich | On demand | Follow table 1 |
| Equipment | |||
| Nanodrop™ One Spectrophotometer | Thermo-Scientific | ND-ONE-W | |
| Refrigerated microcentrifuge Sigma 1-14K, suitable for centrifugation of 1.5 ml microcentrifuge tubes at 14,000 rpm | Sigma-Aldrich | 1-14K | |
| Disinfected adjustable pipettes, range 2-20 µl, 20-200 µl | Gilson | SKU:F167360 | |
| Disposable 1.5 ml microcentrifuge tubes (autoclaved) | Axygen | MCT-150-SP | |
| Disposable 600 µl microcentrifuge tubes (autoclaved) | Axygen | 3208 | |
| Disposable Pipette tips 1-10 µl | Axygen | T-300 | |
| Disposable Pipette tips 1-200 µl | Axygen | TR-222-Y | |
| Dot-Blot apparatus Bio-Dot | BIORAD | 1706545 | |
| Portable Hergom Suction | Hergom | 7E-A | |
| Scientific Light Box (Visible-light PH90-115V) | Hoefer | PH90-115V | |
| UV Crosslinker | Hoefer | UVC-500 | |
| Thermo Hybaid PCR Express Thermocycler | Hybaid | HBPX110 | |
| Radiographic cassette with IP Plate14 X 17 | Fuji |
References
- Bierque, E., Thibeaux, R., Girault, D., Soupé-Gilbert, M. E., Goarant, C. A systematic review of Leptospira in water and soil environments. PLOS One. 15 (1), e0227055 (2020).
- Haake, D. A., Levett, P. N. Leptospirosis in humans. Current Topics in Microbiology and Immunology. 387, 65-97 (2015).
- Tripathy, D. N., Hanson, L. E. Leptospires from water sources at Dixon Springs Agricultural Center. Journal of Wildlife Diseases. 9 (3), 209-212 (1973).
- Smith, D. J., Self, H. R. Observations on the survival of Leptospira australis A in soil and water. The Journal of Hygiene. 53 (4), 436-444 (1955).
- Karpagam, K. B., Ganesh, B. Leptospirosis: a neglected tropical zoonotic infection of public health importance-an updated review. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 39 (5), 835-846 (2020).
- Casanovas-Massana, A., et al. Spatial and temporal dynamics of pathogenic Leptospira in surface waters from the urban slum environment. Water Research. 130, 176-184 (2018).
- Costa, F., et al. Global morbidity and mortality of Leptospirosis: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003898 (2015).
- Mwachui, M. A., Crump, L., Hartskeerl, R., Zinsstag, J., Hattendorf, J. Environmental and behavioural determinants of Leptospirosis transmission: A systematic review. PLOS Neglected Tropical Diseases. 9 (9), e0003843 (2015).
- Andre-Fontaine, G., Aviat, F., Thorin, C. Waterborne Leptospirosis: Survival and preservation of the virulence of pathogenic Leptospira spp. in fresh water. Current Microbiology. 71 (1), 136-142 (2015).
- Trueba, G., Zapata, S., Madrid, K., Cullen, P., Haake, D. Cell aggregation: A mechanism of pathogenic Leptospira to survive in freshwater. International Microbiology: the Official Journal of the Spanish Society for Microbiology. 7 (1), 35-40 (2004).
- Smith, C. E., Turner, L. H. The effect of pH on the survival of leptospires in water. Bulletin of the World Health Organization. 24 (1), 35-43 (1961).
- Barragan, V. A., et al. Interactions of Leptospira with environmental bacteria from surface water. Current Microbiology. 62 (6), 1802-1806 (2011).
- Abdoelrachman, R. Comparative investigations into the influence of the presence of bacteria on the life of pathogenic and apathogenic leptospirae. Antonie van Leeuwenhoek. 13 (1), 21-32 (1947).
- Singh, R., et al. Microbial diversity of biofilms in dental unit water systems. Applied and Environmental Microbiology. 69 (6), 3412-3420 (2003).
- Kumar, K. V., Lall, C., Raj, R. V., Vedhagiri, K., Vijayachari, P. Coexistence and survival of pathogenic leptospires by formation of biofilm with Azospirillum. FEMS Microbiology Ecology. 91 (6), 051 (2015).
- Yanagihara, Y., et al. Leptospira Is an environmental bacterium that grows in waterlogged soil. Microbiology Spectrum. 10 (2), 0215721 (2022).
- Gillespie, R. W., Ryno, J. Epidemiology of leptospirosis. American Journal of Public Health and Nation’s Health. 53 (6), 950-955 (1963).
- Bierque, E., et al. Leptospira interrogans retains direct virulence after long starvation in water. Current Microbiology. 77 (10), 3035-3043 (2020).
- Zhang, Y., Dai, B. Marking and detection of DNA of leptospires in the dot-blot and situ hybridization with digoxigenin-labeled probes. Journal of West China University of Medical Sciences. 23 (4), 353-435 (1992).
- Mérien, F., Amouriaux, P., Perolat, P., Baranton, G., Saint Girons, I. Polymerase chain reaction for detection of Leptospira spp. in clinical samples. Journal of Clinical Microbiology. 30 (9), 2219-2224 (1992).
- Veerapandian, R., et al. Silver enhanced nano-gold dot-blot immunoassay for leptospirosis. Journal of Microbiological Methods. 156, 20-22 (2019).
- Junpen, S., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Ishmael, F. T., Stellato, C. Principles and applications of polymerase chain reaction: basic science for the practicing physician. Annals of Allergy, Asthma & Immunology: Official Publication of the American College of Allergy, Asthma, & Immunology. 101 (4), 437-443 (2008).
- Boerner, B., Weigelt, W., Buhk, H. J., Castrucci, G., Ludwig, H. A sensitive and specific PCR/Southern blot assay for detection of bovine herpesvirus 4 in calves infected experimentally. Journal of Virological Methods. 83 (1-2), 169-180 (1999).
- Curry, E., Pratt, S. L., Kelley, D. E., Lapin, D. R., Gibbons, J. R. Use of a Combined duplex PCR/Dot-blot assay for more sensitive genetic characterization. Biochemistry Insights. 1, 35-39 (2008).
- Pilatti, M. M., Ferreira, S. d. e. A., de Melo, M. N., de Andrade, A. S. Comparison of PCR methods for diagnosis of canine visceral leishmaniasis in conjunctival swab samples. Research in Veterinary Science. 87 (2), 255-257 (2009).
- Conrads, G., et al. PCR reaction and dot-blot hybridization to monitor the distribution of oral pathogens within plaque samples of periodontally healthy individuals. Journal of Periodontology. 67 (10), 994-1003 (1996).
- Langa, S., et al. Differentiation of Enterococcus faecium from Lactobacillus delbrueckii subsp. bulgaricus and Streptococcus thermophilus strains by PCR and dot-blot hybridisation. International Journal of Food Microbiology. 88 (2-3), 197-200 (2003).
- Francesca, C., Lucilla, I., Marco, F., Giuseppe, C., Marisa, M. Identification of the unculturable bacteria Candidatus arthromitus in the intestinal content of trouts using dot-blot and Southern blot techniques. Veterinary Microbiology. 156 (3-4), 389-394 (2012).
- Arent, Z., Pardyak, L., Dubniewicz, K., Plachno, B. J., Kotula-Balak, M. Leptospira taxonomy: then and now. Medycyna Weterynaryjna. 78 (10), 489-496 (2022).
- Thibeaux, R., et al. Biodiversity of environmental Leptospira: Improving identification and revisiting the diagnosis. Frontiers in Microbiology. 9, 816 (2018).
- Carrillo-Casas, E. M., Hernández-Castro, R., Suárez-Güemes, F., de la Peña-Moctezuma, A. Selection of the internal control gene for real-time quantitative RT-PCR assays in temperature treated Leptospira. Current Microbiology. 56 (6), 539-546 (2008).
- Azali, M. A., Yean Yean, C., Harun, A., Aminuddin Baki A, N. N., Ismail, N. Molecular characterization of Leptospira spp. in environmental samples from North-Eastern Malaysia revealed a pathogenic strain, Leptospira alstonii. Journal of Tropical Medicine. 2016, 2060241 (2016).
- Ahmed, N., et al. Multilocus sequence typing method for identification and genotypic classification of pathogenic Leptospira species. Annals of Clinical Microbiology and Antimicrobials. 5, 28 (2006).
- Bourhy, P., Collet, L., Brisse, S., Picardeau, M. Leptospira mayottensis sp. nov., a pathogenic species of the genus Leptospira isolated from humans. International Journal of Systematic and Evolutionary Microbiology. 64, 4061-4067 (2014).
- Weiss, S., et al. An extended Multilocus Sequence Typing (MLST) scheme for rapid direct typing of Leptospira from clinical samples. PLOS Neglected Tropical Diseases. 10 (9), e0004996 (2016).
- Branger, C., et al. Polymerase chain reaction assay specific for pathogenic Leptospira based on the gene hap1 encoding the hemolysis-associated protein-1. FEMS Microbiology Letters. 243 (2), 437-445 (2005).
- Ren, S. X., et al. Unique physiological and pathogenic features of Leptospira interrogans revealed by whole-genome sequencing. Nature. 422 (6934), 888-893 (2003).
- Picardeau, M., et al. Genome sequence of the saprophyte Leptospira biflexa provides insights into the evolution of Leptospira and the pathogenesis of leptospirosis. PLOS One. 3 (2), e1607 (2008).
- Kafatos, F. C., Jones, C. W., Efstratiadis, A. Determination of nucleic acid sequence homologies and relative concentrations by a dot hybridization procedure. Nucleic Acids Research. 7 (6), 1541-1552 (1979).
- Bhat, A. I., Rao, G. P. Dot-blot hybridization technique. Characterization of Plant Viruses. , 303-321 (2020).
- Yadav, J. P., Batra, K., Singh, Y., Singh, M. Comparative evaluation of indirect-ELISA and Dot-blot assay for serodetection of Mycoplasma gallisepticum and Mycoplasma synoviae antibodies in poultry. Journal of Microbiological Methods. 189, 106317 (2021).
- Malinen, E., Kassinen, A., Rinttilä, T., Palva, A. Comparison of real-time PCR with SYBR Green I or 5′-nuclease assays and dot-blot hybridization with rDNA-targeted oligonucleotide probes in quantification of selected faecal bacteria. Microbiology. 149, 269-277 (2003).
- Wyss, C., et al. Treponema lecithinolyticum sp. nov., a small saccharolytic spirochaete with phospholipase A and C activities associated with periodontal diseases. International Journal of Systematic Bacteriology. 49, 1329-1339 (1999).
- Shah, J. S., I, D. C., Ward, S., Harris, N. S., Ramasamy, R. Development of a sensitive PCR-dot-blot assay to supplement serological tests for diagnosing Lyme disease. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 37 (4), 701-709 (2018).
- Niu, C., Wang, S., Lu, C. Development and evaluation of a dot-blot assay for rapid determination of invasion-associated gene ibeA directly in fresh bacteria cultures of E. coli. Folia microbiologica. 57 (6), 557-561 (2012).
- Wetherall, B. L., McDonald, P. J., Johnson, A. M. Detection of Campylobacter pylori DNA by hybridization with non-radioactive probes in comparison with a 32P-labeled probe. Journal of Medical Microbiology. 26 (4), 257-263 (1988).
- Kolk, A. H., et al. Detection of Mycobacterium tuberculosis in clinical samples by using polymerase chain reaction and a nonradioactive detection system. Journal of Clinical Microbiology. 30 (10), 2567-2575 (1992).
- Scherer, L. C., et al. PCR colorimetric dot-blot assay and clinical pretest probability for diagnosis of Pulmonary Tuberculosis in smear-negative patients. BMC Public Health. 7, 356 (2007).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. The Clinical Biochemist Reviews. 29, S49-S52 (2008).
- Zhang, Y., Dai, B. Detection of Leptospira by dot-blot hybridization with photobiotin- and 32P-labeled DNA. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 130-132 (1992).
- Terpstra, W. J., Schoone, G. J., ter Schegget, J. Detection of leptospiral DNA by nucleic acid hybridization with 32P- and biotin-labeled probes. Journal of Medical Microbiology. 22 (1), 23-28 (1986).
- Shukla, J., Tuteja, U., Batra, H. V. DNA probes for identification of leptospires and disease diagnosis. The Southeast Asian Journal of Tropical Medicine and Public Health. 35 (2), 346-352 (2004).
- Jiang, N., Jin, B., Dai, B., Zhang, Y. Identification of pathogenic and nonpathogenic leptospires by recombinant probes. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 26 (1), 1-5 (1995).
- Fach, P., Trap, D., Guillou, J. P. Biotinylated probes to detect Leptospira interrogans on dot-blot hybridization or by in situ hybridization. Letters in Applied Microbiology. 12 (5), 171-176 (1991).
- Huang, N., Dai, B. Assay of genomic DNA homology among strains of different virulent leptospira by DNA hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (2), 122-125 (1992).
- Dong, X., Dai, B., Chai, J. Homology study of leptospires by molecular hybridization. Journal of West China University of Medical Sciences = Huaxi like daxue xuebao. 23 (1), 1-4 (1992).
- Komminoth, P. Digoxigenin as an alternative probe labeling for in situ hybridization. Diagnostic Molecular Pathology: The American Journal of Surgical Pathology, part B. 1 (2), 142-150 (1992).
- Saengjaruk, P., et al. Diagnosis of human leptospirosis by monoclonal antibody-based antigen detection in urine. Journal of Clinical Microbiology. 40 (2), 480-489 (2002).
- Okuda, M., et al. Enzyme-linked immunosorbent assay for the detection of canine Leptospira antibodies using recombinant OmpL1 protein. The Journal of Veterinary Medical Science. 67 (3), 249-254 (2005).
- Suwimonteerabutr, J., et al. Evaluation of a monoclonal antibody-based dot-blot ELISA for detection of Leptospira spp in bovine urine samples. American Journal of Veterinary Research. 66 (5), 762-766 (2005).
- Kanagavel, M., et al. Peptide-specific monoclonal antibodies of Leptospiral LigA for acute diagnosis of leptospirosis. Scientific reports. 7 (1), 3250 (2017).
- Levett, P. N. Leptospirosis. Clinical Microbiology Reviews. 14 (2), 296-326 (2001).
- Monahan, A. M., Callanan, J. J., Nally, J. E. Proteomic analysis of Leptospira interrogans shed in urine of chronically infected hosts. Infection and Immunity. 76 (11), 4952-4958 (2008).
- Rojas, P., et al. Detection and quantification of leptospires in urine of dogs: a maintenance host for the zoonotic disease leptospirosis. European Journal of Clinical Microbiology & Infectious Diseases: Official Publication of the European Society of Clinical Microbiology. 29 (10), 1305-1309 (2010).

