RIBO-seq em Bactérias: um Protocolo de Coleta de Amostras e Preparação de Bibliotecas para Sequenciamento NGS
Summary
Aqui descrevemos os estágios de coleta e preparação da amostra para RIBO-seq em bactérias. O sequenciamento das bibliotecas preparadas de acordo com essas diretrizes resulta em dados suficientes para análise bioinformática abrangente. O protocolo que apresentamos é simples, utiliza equipamentos de laboratório padrão e leva sete dias desde a lise até a obtenção de bibliotecas.
Abstract
A técnica de criação de perfil ribossomo (RIBO-seq) é atualmente a ferramenta mais eficaz para estudar o processo de síntese proteica in vivo. A vantagem deste método, em comparação com outras abordagens, é sua capacidade de monitorar a tradução mapeando precisamente a posição e o número de ribossomos em uma transcrição mRNA.
Neste artigo, descrevemos as etapas consecutivas de coleta e preparação da amostra para o método RIBO-seq em bactérias, destacando os detalhes relevantes para o planejamento e execução do experimento.
Uma vez que o RIBO-seq conta com ribossomos intactos e mRNAs relacionados, o passo chave é a rápida inibição da tradução e a desintegração adequada das células. Assim, sugerimos filtração e congelamento de flash em nitrogênio líquido para colheita celular com um pré-tratamento opcional com clororamfenícol para prender a tradução em bactérias. Para a desintegração, propomos moer células congeladas com argamassa e pilão na presença de óxido de alumínio para interromper mecanicamente a parede celular. Neste protocolo, não é necessária uma almofada de sacarose ou uma ultracentrifugação gradiente de sacarose para purificação monossótil. Em vez disso, a separação mRNA usando eletroforese de gel de poliacrilamida (PAGE) seguida pela excisão da pegada ribossômica (banda 28-30 nt) é aplicada e fornece resultados satisfatórios. Isso simplifica em grande parte o método, bem como reduz os requisitos de tempo e equipamento para o procedimento. Para a preparação da biblioteca, recomendamos usar o kit RNA pequeno disponível comercialmente para sequenciamento de illumina da New England Biolabs, seguindo as diretrizes do fabricante com algum grau de otimização.
As bibliotecas cDNA resultantes apresentam quantidade e qualidade adequadas para o sequenciamento de próxima geração (NGS). O sequenciamento das bibliotecas preparadas de acordo com os resultados do protocolo descrito em leituras mapeadas exclusivamente por amostra fornece dados suficientes para análise bioinformática abrangente. O protocolo que apresentamos é rápido e relativamente fácil e pode ser realizado com equipamentos de laboratório padrão.
Introduction
A técnica de perfil ribossomo (RIBO-seq) foi desenvolvida no laboratório de Jonathan Weissman na Universidade da Califórnia, São Francisco1. Em comparação com outros métodos usados para estudar a expressão genética no nível translacional, RIBO-seq concentra-se em cada ligação ribossosome ao mRNA e fornece informações sobre sua localização e o número relativo de ribossomos em uma transcrição. Permite monitorar o processo de síntese proteica in vivo e pode fornecer resolução e precisão de códon único permitindo a medição da densidade ribossomo em ambos, o mRNA individual e ao longo de todo o transcriptome na célula. Na base da técnica RIBO-seq está o fato de que durante a tradução o ribossomo liga a molécula de mRNA e, assim, protege o fragmento enterrado da transcrição de uma digestão ribonuclease. Após a adição da ribonuclease, o mRNA desprotegido é digerido e os fragmentos fechados por ribossomos – tipicamente de ~28-30 nt de comprimento – permanecem intactos. Esses fragmentos, chamados pegadas ribossômicas (RF), podem então ser isolados, sequenciados e mapeados na transcrição de que se originaram resultando na detecção da posição exata dos ribossomos. De fato, a capacidade ribossômica de proteger fragmentos de mRNA tem sido usada desde a década de 1960 para estudar os locais de iniciação de ligação ribossômica e tradução (TIS)2,3,4. No entanto, com o avanço da tecnologia de sequenciamento profundo, o RIBO-seq tornou-se um padrão ouro para o monitoramento de tradução5 que, através do engajamento ribossomo, pode fornecer uma informação em todo o genoma sobre asíntese deproteínas 6 . O perfil ribossomo preencheu a lacuna tecnológica existente entre quantificar o transcriptome e o proteome6.
Para realizar o perfil ribossomo precisamos obter lisecelulares do organismo que cresceu sob as condições investigadas. Interromper essas condições durante a coleta e lise celular pode fornecer dados não confiáveis. Para evitar isso, inibidores de tradução, colheita rápida e congelamento de flash em nitrogênio líquido são comumente usados. As células podem ser lístidas pela moagem criogênica em um homogeneizador mecânico como um moinho de batedeira7,8 ou um batedor de contas9, e por trituração através de uma pipeta10 ou com uma agulha11. O tampão de lise pode ser adicionado pouco antes ou pouco depois da pulverização das células. Em nosso protocolo usamos nitrogênio líquido para argamassa pré-colono e pilão, bem como óxido de alumínio como uma abordagem mais suave para a interrupção da parede celular bacteriana, o que impede a tesoura de RNA frequentemente encontrada quando métodos como a sonificação são aplicados. Após a pulverização, adicionamos um tampão de lise gelada no conteúdo resfriado da argamassa. A seleção de um tampão de lise apropriado é importante para obter a melhor resolução de pegadas ribossômicas. Uma vez que a força iônica afeta tanto o tamanho do RF quanto a precisão do quadro de leitura, recomenda-se atualmente o uso de tampões de lise com baixa resistência iônica e capacidade de tampão, mesmo que pareça que a composição do buffer não afeta a ocupação ribossômica em mRNAs11,12. Componentes importantes do tampão de lise são íons de magnésio, a presença que impede a dissociação das subunidades ribossômicas e inibe alterações conformais espontâneas nos ribossomos bacterianos11,13. Os íons de cálcio também desempenham um papel significativo e são essenciais para a atividade da nuclease microcócica (MNase) utilizada no método de perfil ribossomo bacteriano14. Adição de guanosina 5′-[β,γ-imido]triphosfato (GMP-PNP), um análogo não hidroglizável do GTP, juntamente com o clorofenicol inibe a tradução durante a lise15.
Quando o lisato é obtido, ele é esclarecido por centrifugação e dividido em duas porções, cada uma para um RIBO-seq e um sequenciamento de mRNA total de alta produtividade (RNA-seq) uma vez que são realizados simultaneamente(Figura 1). O RNA-seq fornece um ponto de referência que permite a comparação de dados tanto do RIBO-seq quanto do RNA-seq durante a análise dos dados. O translatome investigado é definido pela normalização das pegadas ribossômicas à abundância de mRNA16. Os dados do RNA-seq também podem ajudar a identificar a clonagem ou sequenciamento de artefatos17.
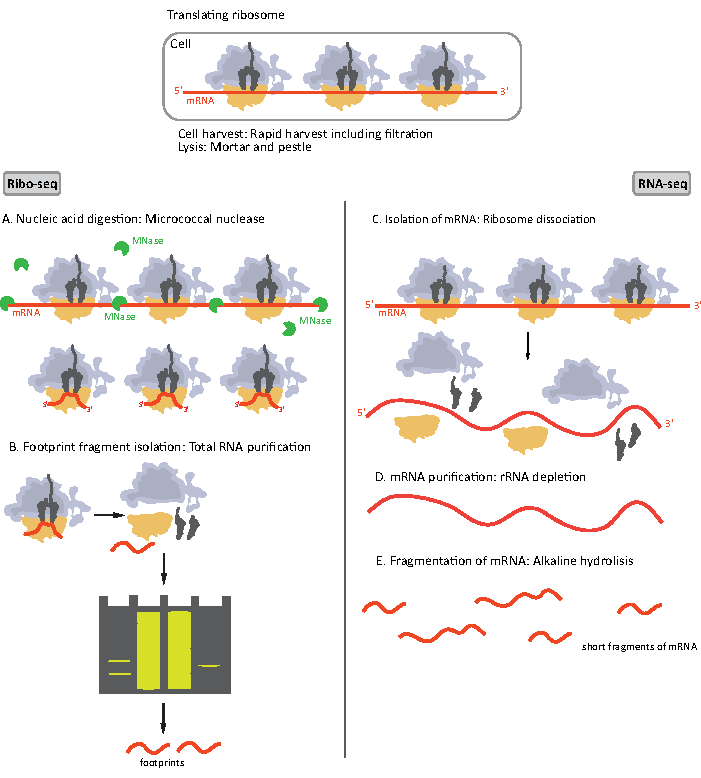
Figura 1. Esquemas de preparação de amostras de mRNA para RIBO-seq e RNA-seq. Para a preparação da biblioteca RIBO-seq, o RNA é digerido com MNase (A), seguido pela seleção de tamanho de RF de ~28-30 nt de comprimento (B); para RNA-seq RNA é isolado (C), esgotado de rRNA (D), e o mRNA resultante é aleatoriamente fragmentado em fragmentos de comprimentos variados (E). Clique aqui para ver uma versão maior desta figura.
As etapas iniciais do procedimento de preparação da amostra para RIBO-seq e RNA-seq diferem ligeiramente(Figura 1). Para o perfil ribossômico, o liseto precisa ser digerido por uma endonuclease específica para degradar as moléculas de mRNA não protegidas pelos ribossomos. Nos protocolos padrão, os monossomos obtidos são recuperados por uma ultracentrifugação de almofada de sacarose ou uma ultracentrifugação de gradiente de sacarose8,14. Neste artigo, mostramos que esta etapa não é necessária para isolar a RF necessária para o RIBO-seq em bactérias, da mesma forma para as células eucarióticas18, e que a seleção de tamanho dos fragmentos de comprimento apropriado do gel de poliacrilamida é suficiente.
Para RNA-seq, o mRNA é obtido pelo esgotamento do rRNA do total de moléculas de RNA – rRNA hibridizem-se às sondas oligonucleotidas biotinínas que se ligam às contas magnéticas revestidas de streptavidin. Os complexos rRNA-oligonucleotídeo-contas são então removidos da amostra com um ímã resultando em uma amostra esgotada de rRNA19,20. As moléculas de mRNA purificadas são então fragmentadas aleatoriamente por hidrólise alcalina. Os fragmentos obtidos de mRNA, bem como as pegadas ribossômicas são convertidos em bibliotecas cDNA e preparados para sequenciamento profundo(Figura 2). Isso envolve o reparo necessário após a hidrólise alcalina (para mRNA) e digestão enzimática (para RF): desfosforilação de extremidades de 3′ seguida de fosforilação de extremidades de 5′. Os próximos passos são a ligadura dos adaptadores e a transcrição reversa para criar inserções cDNA emolduradas por sequências necessárias para o sequenciamento de próxima geração (NGS) usando a plataforma Illumina. A última fase da preparação da biblioteca é uma reação pcr na qual os construtos são amplificados e rotulados com códigos de barras específicos de amostra para permitir multiplexagem e sequenciamento de várias amostras em um único canal. Antes do sequenciamento, a qualidade e a quantidade das bibliotecas são avaliadas pelo DNA de alta sensibilidade na eletroforese do chip. bibliotecas cDNA com parâmetros apropriados podem então ser agrupadas e sequenciadas. O sequenciamento pode ser realizado em diferentes plataformas de Illumina, como MiSeq, NextSeq ou HighSeq, dependendo do número de bibliotecas, comprimento de leitura necessário e profundidade de sequenciamento. Após o sequenciamento, a análise bioinformática é realizada.

Figura 2. Preparação da biblioteca. A preparação da biblioteca inclui o reparo de extremidades, ligadura dos adaptadores, transcrição reversa e amplificação com codificação de barras. Clique aqui para ver uma versão maior desta figura.
O perfil ribossomo é um método universal que pode ser facilmente modificado e ajustado de acordo com a questão científica. Originalmente foi usado na levedura1, mas logo depois foi aplicado às células bacterianas21, bem como organismos modelo eucarióticos, incluindo camundongos10,zebrafish22, mosca de frutas23 e Arabidopsis thaliana24. Também foi utilizado para o estudo de diferentes tipos ribossósmicos: citoplasmásmico, mitocondrial25,26 e cloroplasto27,28. Em eucariotes RIBO-seq é comumente adaptado e refinado para investigar aspectos específicos da tradução, incluindo iniciação10,11,29,30,31,32, alongamento1,10,11,31,33, ribossomo parando33 e alteração de conformação33. A maioria das modificações envolve o uso de diferentes inibidores de tradução. No entanto, nas bactérias, estudos análogos têm sido difíceis de realizar devido à escassez de inibidores com o mecanismo de ação necessário34. O inibidor de tradução mais usado em bactérias é o clorofífenicol (CAM), que se liga ao centro de transferência de peptidyl (PTC) e impede o posicionamento correto do aminoacílico-tRNA no local A. Como resultado, cam impede a formação de um vínculo peptídeo que leva à prisão dos ribossomosalongamentos 35. Outros exemplos de inibidores de tradução em bactérias são a tetraciclina (TET)36, retapamulina (RET)34 e Onc11237 que foram usados para investigar sites de iniciação de tradução. O TET, que impede a entrega de tRNA ao ribossomo, sobrepondo-se diretamente com o loop anticodon de tRNA no local A, foi originalmente aplicado para verificar os resultados obtidos do tratamento CAM, uma vez que ambos são antibióticos inibindo a alongamento da tradução38. Foi encontrado tet para detectar TIS primário, no entanto foi incapaz de revelar TIS36interno . RET se liga no PTC do ribossomo bacteriano, e impede a formação da primeira ligação de peptídeo interferindo com um alongador aminoacyl-tRNA no site A. A aplicação de RET resulta em prisão de ribossomos tanto nas TISs primárias quanto internas34. Onc112, um peptídeo antimicrobiano rico em proline, liga-se no túnel de saída e bloqueia a ligação aminoacíl-tRNA no local ribossômico A. Como resultado, o Onc112 impede que os complexos de iniciação entrem na fase de alongamento37.
A principal informação que o perfil ribossomo fornece é a densidade de ribossomos e sua posição no mRNA. Foi aplicado com sucesso para investigar a expressão genética diferencial no nível da tradução em várias condições de crescimento1,6, medir a eficiência translacional1,38,39 e detectar eventos de regulação de tradução, como a pausa ribossômica10. RIBO-seq também permite descobrir a tradução de ncRNA anotado, pseudogenes e pequenos quadros de leitura abertos não anotados (ORF) levando à identificação de genes de codificação de proteínas novos e/ou muito curtos10,12,22,30,37. Nesses casos, ribo-seq pode ajustar e melhorar a anotação do genoma. Com sua alta sensibilidade para a identificação de ORFs traduzidos e sua natureza quantitativa, o perfil ribossomo também pode servir como proxy para a determinação proteome ou para auxiliar estudos de proteômica31,34,39. Ao mapear o TIS, o perfil ribossomo revela isóformes n-terminalmente estendidos e truncados de proteínas conhecidas10,32. RIBO-seq também foi adaptado para estudar o dobrável co-translacional das proteínas14,21,24. Este método permite medir as taxas de alongamento1,10,39 ou velocidades de decodificação de códons individuais6 e ajuda no desenvolvimento de modelos quantitativos de tradução17. O método de criação de perfil ribossomo também é capaz de fornecer insights mecanicistas sobre a pausa ribossa em bactérias7,15,17, frameshifting40, stop-codon readthrough21, defeitos de terminação/reciclagem41,42 e alterações de conformação ribossômica33 em eucariotos. RIBO-seq também foi adaptado para examinar o impacto de fatores trans-acionados específicos na tradução, como miRNAs6 e proteínas de ligação de RNA em eucariotes16,43. No entanto, é importante reconhecer que o projeto experimental e a resolução obtida do RIBO-seq determinam a quantidade de informações que podem ser extraídas dos dados de sequenciamentoresultantes 12.
Protocol
Representative Results
Discussion
O principal desafio técnico do perfil ribossomo é a necessidade de inibir rapidamente a tradução para capturar um instantâneo de ribossomos em mRNAs em um determinado estado fisiológico de interesse. Para isso, inibidores de tradução, colheita rápida e congelamento de flash em nitrogênio líquido são comumente usados. A aplicação de antibióticos é opcional, pois podem causar artefatos. Chloramphenicol é uma droga comumente usada para prender ribossomos alongamentos em RIBO-seq bacteriana. No entanto, não…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
A ALS gostaria de reconhecer o apoio financeiro dos subsídios de instalação da EMBO IG 3914 e da POIR. 04.04.00-00-3E9C/17-00 realizado no programa First TEAM da Fundação para a Ciência Polonesa co-financiado pela União Europeia no âmbito do Fundo Europeu de Desenvolvimento Regional.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Riferimenti
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

