Bakterilerde RIBO-seq: NGS Dizilimi için Örnek Toplama ve Kütüphane Hazırlama Protokolü
Summary
Burada bakterilerde RIBO-seq için numune toplama ve hazırlama aşamalarını açıklıyoruz. Bu yönergelere göre hazırlanan kütüphanelerin sıraya dizilimi, kapsamlı biyoinformatik analiz için yeterli veri ile sonuçlanır. Sunduğumuz protokol basittir, standart laboratuvar ekipmanlarını kullanır ve lizizden kütüphanelerin eldeine kadar yedi gün sürer.
Abstract
Ribozom profilleme tekniği (RIBO-seq) şu anda protein sentezi sürecini incelemek için en etkili araçtır in vivo. Bu yöntemin avantajı, diğer yaklaşımlara kıyasla, bir mRNA transkriptinde ribozomların konumunu ve sayısını tam olarak haritalayarak çeviriyi izleme yeteneğidir.
Bu yazıda, bakterilerde RIBO-seq yöntemi için numune toplama ve hazırlamanın ardışık aşamalarını açıklar, deneyin planlanması ve yürütülmesi ile ilgili ayrıntıları vurgularız.
RIBO-seq bozulmamış ribozomlara ve ilgili mRNA’lara dayandığından, anahtar adım çevirinin hızlı inhibisyonu ve hücrelerin yeterli parçalanmasıdır. Bu nedenle, bakterilerde çeviriyi tutuklamak için kloramfenikol ile isteğe bağlı bir ön işlemle hücre hasadı için sıvı nitrojende filtrasyon ve flaş dondurma öneriyoruz. Parçalanma için, hücre duvarını mekanik olarak bozmak için alüminyum oksit varlığında donmuş hücrelerin harç ve pestle ile öğütülmeyi öneriyoruz. Bu protokolde, monozomlu saflaştırma için sakkaroz yastığı veya sakkaroz gradyan ultrasantrifüj gereklidir. Bunun yerine poliakrilamid jel elektroforez (PAGE) kullanılarak mRNA ayrımı ve ardından ribozomal ayak izi eksizyonu (28-30 nt bandı) uygulanır ve tatmin edici sonuçlar sağlar. Bu, yöntemi büyük ölçüde basitleştirir ve prosedür için zaman ve ekipman gereksinimlerini azaltır. Kütüphane hazırlığı için, New England Biolabs’tan Illumina dizilimi için piyasada bulunan küçük RNA kitini kullanmanızı öneririz, üreticinin yönergelerini bir dereceye kadar optimizasyonla takip edin.
Elde edilen cDNA kitaplıkları, yeni nesil sıralama (NGS) için gereken uygun miktar ve kaliteyi sunar. Açıklanan protokole göre hazırlanan kütüphanelerin sıraya dizilimi, kapsamlı biyoinformatik analiz için yeterli veri sağlayan örnek başına 2 ila 10 mln benzersiz haritalı okuma ile sonuçlanır. Sunduğumuz protokol hızlı ve nispeten kolaydır ve standart laboratuvar ekipmanları ile gerçekleştirilebilir.
Introduction
Ribozom profilleme tekniği (RIBO-seq), Kaliforniya Üniversitesi, San Francisco1’dekiJonathan Weissman’ın laboratuvarında geliştirilmiştir. Ribo-seq, gen ekspresyonunu çeviri düzeyinde incelemek için kullanılan diğer yöntemlere kıyasla, mRNA’ya her ribozom bağlamasına odaklanır ve konumu ve transkriptteki ribozomların göreceli sayısı hakkında bilgi sağlar. Protein sentezi sürecinin in vivo olarak izlenmesini sağlar ve hem bireysel mRNA’da hem de hücredeki tüm transkriptom boyunca ribozom yoğunluğunun ölçülmesine izin veren tek kodon çözünürlüğü ve doğruluğu sağlayabilir. RIBO-seq tekniğinin temelinde, çeviri sırasında ribozomların mRNA molekülünü bağlaması ve böylece transkriptin gömülü parçasını bir ribonükleaz sindiriminden koruması yatmaktadır. Ribonokleaz ek olarak, korunmasız mRNA sindirilir ve ribozomlar tarafından çevrelenmiş parçalar – tipik olarak ~ 28-30 nt uzunluğunda – bozulmadan kalır. Ribozomal ayak izleri (RF) olarak adlandırılan bu parçalar daha sonra izole edilebilir, sıralanabilir ve ribozomların tam konumunun tespit edilmesiyle ortaya çıktıkları transkript üzerine eşlenebilir. Aslında, ribozom yeteneği mRNA parçalarını korumak için ribozom yeteneği 1960’lardan beri ribozomal bağlama ve çeviri başlatma alanlarını (Tİs) incelemek içinkullanılmaktadır 2,3,4. Bununla birlikte, derin sıralama teknolojisindeki ilerleme ile RIBO-seq, ribozom katılımı yoluyla protein sentezi 6 hakkında genom çapında bilgi sağlayabilen çeviri izleme5 için altın bir standarthalinegelmiştir. Ribozom profilleme, transkriptom ve proteom6’nınölçülmesi arasında var olan teknolojik boşluğu doldurdu.
Ribozom profilleme yapmak için, araştırılan koşullar altında büyüyen organizmanın hücre lizatını elde etmeliyiz. Hücre toplama ve lizis sırasında bu koşulların bozulması güvenilmez veriler sağlayabilir. Bunu önlemek için, çeviri inhibitörleri, hızlı hasat ve sıvı azot içinde flaş dondurma yaygın olarak kullanılır. Hücreler, mikser değirmeni7,8 veya boncuk çırpıcı9gibi mekanik bir homojenizatörde kriyojenik taşlama ve pipet10 veya iğne ile tritürasyon ile11ile yutabilir. Lizis tamponu, hücrelerin pulverizasyondan hemen önce veya kısa bir süre sonra eklenebilir. Protokolümüzde, harç ve pestle’yi ön soğutmak için sıvı nitrojenin yanı sıra, sonifikasyon gibi yöntemler uygulandığında sıklıkla karşılaşılan RNA makasını önleyen bakteri hücre duvarının bozulmasına daha nazik bir yaklaşım olarak alüminyum oksit kullanıyoruz. Pulverizasyondan sonra harcın soğutulmuş içeriğine buz gibi bir lizis tamponu ekliyoruz. Uygun bir lizis tamponunun seçilmesi ribozomal ayak izlerinin en iyi çözünürlüğünü elde etmek için önemlidir. İyonik mukavemet hem RF boyutunu hem de okuma çerçevesi hassasiyetini etkilediğinden, tampon bileşiminin mRNA’lar11,12’deribozomal doluluğu etkilemediği görünse bile, düşük iyonik mukavemet ve tampon kapasitesine sahip lizis tamponlarının kullanılması önerilir. Lizis tamponunun önemli bileşenleri, varlığı ribozomal alt ünüllerin ayrışmasını önleyen ve bakteriyel ribozomlardaki spontan konformasyonel değişiklikleri inhibe eden magnezyum iyonlarıdır11,13. Kalsiyum iyonları da önemli bir rol oynar ve bakteriyel ribozom profilleme yönteminde kullanılan mikrokoksal çekirdeğin (MNaze) aktivitesi için gereklidir14. Guanosine ilavesi 5′-[β,γ-imido]trifosfat (GMP-PNP), GTP’nin hidrolize edilemez bir analogu, kloramfenikol ile birlikte liziz sırasında çeviriyi engeller15.
Lysate elde edildiğinde, santrifüjleme ile netleştirilir ve her biri bir RIBO-seq ve aynı anda gerçekleştirildikleri için yüksek verimli toplam mRNA dizilimi (RNA-seq) için iki bölüme ayrılır (Şekil 1). RNA-seq, veri analizi sırasında hem RIBO-seq hem de RNA-seq’ten gelen verilerin karşılaştırılmasına olanak sağlayan bir referans noktası sağlar. Araştırılan translatom, ribozomal ayak izlerinin mRNA bolluğuna normalleştirilmesi ile tanımlanır16. RNA-seq’ten gelen veriler,17kopyalama veya sıralama yapıtlarını tanımlamaya da yardımcı olabilir.
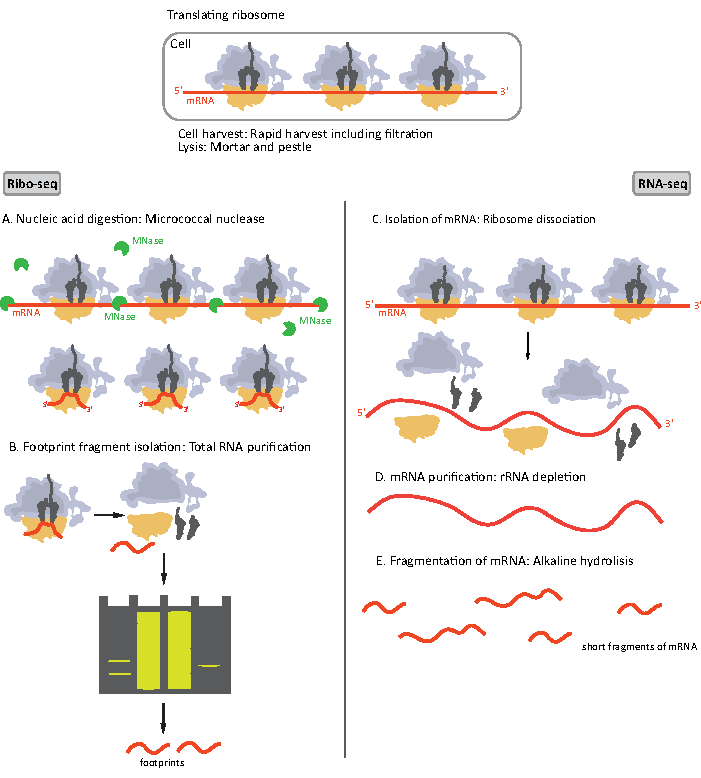
Şekil 1. RIBO-seq ve RNA-seq için mRNA örnek hazırlama şemaları. RIBO-seq kitaplığı hazırlanması için RNA, MNase (A) ile sindirilir ve ardından ~28-30 nt uzunluğunda (B) RF boyut seçimi; RNA-seq RNA yalıtılır (C), rRNA (D) tükenmiş ve elde edilen mRNA rastgele değişen uzunluklarda (E) parçalara bölünmüş. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
RIBO-seq ve RNA-seq için numune hazırlama prosedürünün ilk adımları biraz farklıdır (Şekil 1). Ribozomal profilleme için, ribozomlar tarafından korunmayan mRNA moleküllerini bozmak için lizatın belirli bir endonokleaz tarafından sindirilmesi gerekir. Standart protokollerde, elde edilen monozomlar bir sakkaroz yastığı ultrasantrifüjasyonu veya sakkaroz gradyan ultracentrifugation8,14ile kurtarılır. Bu yazıda, bu adımın bakterilerde RIBO-seq için gerekli RF’yi izole etmek için gerekli olmadığını, aynı şekilde ökaryotik hücreler için18, ve poliakrilamid jelden uygun uzunlukta mRNA parçalarının boyut seçiminin yeterli olduğunu gösteriyoruz.
RNA-seq için mRNA, rRNA’nın toplam RNA’dan tükenmesi ile elde edilir – rRNA molekülleri, streptavidin kaplı manyetik boncuklara bağlanan biyotinillenmiş oligonükleotid problarına melezleşir. RRNA-oligonükleotid-boncuk kompleksleri daha sonra bir mıknatıs ile numuneden çıkarılır ve rRNA tükenmiş bir örnek19,20ile sonuçlanır. Saflaştırılmış mRNA molekülleri daha sonra alkali hidroliz ile rastgele parçalanır. Elde edilen mRNA parçalarının yanı sıra ribozomal ayak izleri cDNA kütüphanelerine dönüştürülür ve derin sıralama için hazırlanır (Şekil 2). Bu, alkali hidroliz (mRNA için) ve enzimatik sindirimden (RF için) sonra ihtiyaç duyulan uç onarımını içerir: 3′ uçların defosforilasyonu ve ardından 5′ uçların fosforilasyonu. Sonraki adımlar adaptör ligasyonu ve Illumina platformunu kullanarak yeni nesil sıralama (NGS) için gereken sıralarla çerçevelenmiş cDNA eklemeleri oluşturmak için ters transkripsiyondur. Kütüphane hazırlığının son aşaması, yapıların bir kanalda çeşitli örneklerin çoklayıcı ve sıralı olmasına izin vermek için örnek özel barkodlarla yükseltildiği ve etiket edildiği bir PCR reaksiyondur. Sıralamadan önce, kütüphanelerin kalitesi ve miktarı çip üzerindeki yüksek hassasiyetli DNA elektroforez ile değerlendirilir. Uygun parametrelere sahip cDNA kitaplıkları daha sonra havuza alınıp sıralanabilir. Sıralama, kitaplık sayısına, gerekli okuma uzunluğuna ve sıralama derinliğine bağlı olarak MiSeq, NextSeq veya HighSeq gibi farklı Illumina platformlarında gerçekleştirilebilir. Sıralamadan sonra biyoinformatik analiz yapılır.

Şekil 2. Kütüphane hazırlığı. Kütüphane hazırlığı, uç onarımını, adaptör ligasyonunu, ters transkripsiyonu ve barkodlama ile amplifikasyonu içerir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ribozom profilleme, bilimsel soruya göre kolayca değiştirilebilen ve ayarlanabilen evrensel bir yöntemdir. Başlangıçta maya1’dekullanıldı, ancak bakteri hücrelerine uygulandıktan kısa bir süre sonra21 ve fare10, zebra balığı22,meyve sineği 23 ve Arabidopsis thaliana24dahil olmak üzere ökaryotik model organizmalara uygulandı. Ayrıca farklı ribozom türlerini incelemek için de kullanılmıştır: sitoplazmik, mitokondriyal25,26 ve kloroplast27,28. Ökaryotlarda RIBO-seq, başlatma 10 , 11 , 29 , 30 ,31,32,uzama1,10 , 11,31,33,ribozom oyalama 33 ve uygunlukdeğişikliği33 dahil olmak üzere çevirinin belirli yönlerini araştırmak için yaygın olarak uyarlanmış ve rafine edilmiştir. Değişikliklerin çoğu farklı çeviri inhibitörlerinin kullanımını içerir. Bununla birlikte, bakterilerde, gerekli etki mekanizmasına sahip inhibitörlerin azlığı nedeniyle benzer çalışmaların yapılması zor olmuştur34. Bakterilerde en sık kullanılan çeviri inhibitörü, peptidyl transferaz merkezine (PTC) bağlanan ve A bölgesinde aminoasil-tRNA’nın doğru konumlandırılmasını önleyen kloramfenikoldür (CAM). Sonuç olarak, CAM, uzun ribozomların tutuklanmasına yol açan bir peptit bağı oluşumunu önler35. Bakterilerdeki diğer çeviri inhibitörleri tetrasiklin (TET)36, retapamulin (RET)34 ve Onc11237’dir. A-sitedeki tRNA’nın antikon kök döngüsü ile doğrudan örtüşerek ribozom için tRNA doğumunu önleyen TET, başlangıçta CAM tedavisinden elde edilen sonuçları doğrulamak için uygulanmıştır, çünkü her ikisi de çeviri uzamasını engelleyen antibiyotiklerdir38. TET’nin birincil Tİs’i tespit etmesi bulundu, ancak dahili Tİs36’yıortaya koyamadı. RET, bakteriyel ribozom PTC’sinde bağlanır ve A bölgesinde bir uzay aminoasil-tRNA ile müdahale ederek ilk peptit bağının oluşumunu önler. RET uygulamak, hem birincil hem de dahili TISs34’teribozomların tutuklanmasıyla sonuçlanır. Prolin bakımından zengin bir antimikrobiyal peptit olan Onc112, çıkış tüneline bağlanır ve ribozomal A bölgesinde aminoasil-tRNA bağlamasını engeller. Sonuç olarak, Onc112 başlatma komplekslerinin uzama aşaması37’yegirmesini önler.
Ribozom profillemenin sağladığı ana bilgi ribozom yoğunluğu ve mRNA üzerindeki konumlarıdır. Çeşitli büyüme koşullarında çeviri düzeyinde diferansiyel gen ekspresyonunun araştırılması 1,6 , çeviri verimliliğininölçülması 1,38,39 ve ribozomal duraklama10gibi çeviri düzenleme olaylarını tespit etmek için başarıyla uygulanmıştır. RIBO-seq ayrıca, yeni ve / veya çok kısa protein kodlama genlerinin tanımlanmasına yol açan açıklamalı ncRNA, psödojenler ve açıklamalanmamış küçük açık okuma çerçevelerinin (ORF) çevirisini ortaya çıkarmaya izin verir10,12,22,30,37. Bu gibi durumlarda RIBO-seq genom ek açıklamalarını ince ayar yapabilir ve iyileştirebilir. Çevrilmiş ORF’lerin tanımlanmasına ve nicel doğasına yönelik yüksek hassasiyeti ile ribozom profilleme, proteom tayini için veya proteomik çalışmalara yardımcı olmak için bir vekil görevi de edebilir31,34,39. Ribozom profilleme, Tİs’i haritalayarak bilinen proteinlerin N-terminal genişletilmiş ve kesilmiş izoformlarını ortaya koymaktadır10,32. RIBO-seq ayrıca14 , 21,24proteinlerinin ortak çevirisel katlamasını incelemek için uyarlanmıştır. Buyöntem,1 ,10,39 uzama hızlarının veya bireysel kodonların kod çözme hızlarının ölçülmesine olanak tanır 6 veçevirinin nicel modellerinin geliştirilmesine yardımcı olur17. Ribozom profilleme yöntemi ayrıca7, 15,17,frameshifting 40,stop-codon readthrough 21 , sonlandırma/geri dönüşüm kusurları 41,42ve ribozomal konformasyon değişiklikleri33 ökaryotlarda ribozom duraklaması hakkında mekanistik içgörüler sağlayabilir. RIBO-seq ayrıca 16,43ökaryotlarda miRNAs6 ve RNA bağlayıcı proteinler gibi spesifik trans-etkili faktörlerin çeviri üzerindeki etkisini incelemek için uyarlanmıştır. Bununla birlikte, deneysel tasarımın ve RIBO-seq’in elde edilen çözünürlüğünün, elde edilen sıralama verilerinden çıkarabilecek bilgi miktarını belirlediğini kabul etmek önemlidir12.
Protocol
Representative Results
Discussion
Ribozom profillemenin temel teknik zorluğu, belirli bir fizyolojik ilgi durumunda mRNA’larda ribozomların bir anlık görüntüsünü yakalamak için çeviriyi hızla engelleme ihtiyacıdır. Bunu başarmak için, çeviri inhibitörleri, sıvı nitrojende hızlı hasat ve flaş dondurma yaygın olarak kullanılır. Antibiyotik uygulamak isteğe bağlıdır, çünkü eserlere neden olabilirler. Kloramfenikol, bakteriyel RIBO-seq’te uzun ribozomları tutuklamak için yaygın olarak kullanılan bir ilaçtır. Bununla birl…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
ALS, EMBO Kurulum Hibeleri IG 3914 ve POIR’in finansal desteğini kabul etmek istiyor. 04.04.00-00-3E9C/17-00 Avrupa Birliği tarafından Avrupa Bölgesel Kalkınma Fonu kapsamında ortaklaşa finanse edilen Polonya Bilim Vakfı’nın Birinci TAKIM programı dahilinde gerçekleştirildi.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Riferimenti
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

