RIBO-seq in Bakterien: ein Probensammlungs- und Bibliotheksvorbereitungsprotokoll für die NGS-Sequenzierung
Summary
Hier beschreiben wir die Phasen der Probenentnahme und Vorbereitung für RIBO-seq in Bakterien. Die Sequenzierung der nach diesen Richtlinien erstellten Bibliotheken liefert ausreichende Daten für eine umfassende bioinformatische Analyse. Das Protokoll, das wir präsentieren, ist einfach, verwendet Standard-Laborgeräte und dauert sieben Tage von der Lyse bis zur Beschaffung von Bibliotheken.
Abstract
Die Ribosomenprofilierungstechnik (RIBO-seq) ist derzeit das effektivste Werkzeug, um den Prozess der Proteinsynthese in vivo zuuntersuchen. Der Vorteil dieser Methode im Vergleich zu anderen Ansätzen ist ihre Fähigkeit, die Translation zu überwachen, indem die Position und Anzahl der Ribosomen auf einem mRNA-Transkript genau abgebildet wird.
In diesem Artikel beschreiben wir die aufeinanderfolgenden Phasen der Probenentnahme und Vorbereitung für die RIBO-seq-Methode bei Bakterien und heben die Details hervor, die für die Planung und Durchführung des Experiments relevant sind.
Da das RIBO-seq auf intakte Ribosomen und verwandte mRNAs angewiesen ist, ist der Schlüsselschritt eine schnelle Hemmung der Translation und ein adäquater Zerfall der Zellen. Daher empfehlen wir die Filtration und das Schockgefrieren in flüssigem Stickstoff für die Zellernte mit einer optionalen Vorbehandlung mit Chloramphenicol, um die Translation in Bakterien zu unterbrechen. Für den Zerfall schlagen wir vor, gefrorene Zellen mit Mörtel und Stößel in Gegenwart von Aluminiumoxid zu mahlen, um die Zellwand mechanisch zu stören. In diesem Protokoll ist kein Saccharosekissen oder eine Saccharosegradienten-Ultrazentrifugation zur Monosomenreinigung erforderlich. Stattdessen wird die mRNA-Trennung mittels Polyacrylamid-Gelelektrophorese (PAGE) gefolgt von der ribosomalen Fußabdruckexzision (28-30 nt-Band) angewendet und liefert zufriedenstellende Ergebnisse. Dies vereinfacht die Methode weitgehend und reduziert den Zeit- und Geräteaufwand für das Verfahren. Für die Bibliotheksvorbereitung empfehlen wir die Verwendung des kommerziell erhältlichen kleinen RNA-Kits für die Illumina-Sequenzierung von New England Biolabs, das den Richtlinien des Herstellers mit einem gewissen Grad an Optimierung folgt.
Die resultierenden cDNA-Bibliotheken stellen eine angemessene Quantität und Qualität dar, die für Next Generation Sequencing (NGS) erforderlich ist. Die Sequenzierung der nach dem beschriebenen Protokoll erstellten Bibliotheken führt zu 2 bis 10 Mio. eindeutig abgebildeten Lesevorgängen pro Probe, die ausreichende Daten für eine umfassende bioinformatische Analyse liefern. Das Protokoll, das wir präsentieren, ist schnell und relativ einfach und kann mit Standard-Laborgeräten durchgeführt werden.
Introduction
Die Ribosomenprofilierungstechnik (RIBO-seq) wurde im Labor von Jonathan Weissman an der University of California, San Francisco1entwickelt. Im Vergleich zu anderen Methoden zur Untersuchung der Genexpression auf translationaler Ebene konzentriert sich RIBO-seq auf jede Ribosombindung an mRNA und liefert Informationen über seine Position und die relative Anzahl der Ribosomen auf einem Transkript. Es ermöglicht die Überwachung des Prozesses der Proteinsynthese in vivo und kann eine einzelne Codonauflösung und -genauigkeit liefern, die die Messung der Ribosomendichte sowohl auf der einzelnen mRNA als auch entlang des gesamten Transkriptoms in der Zelle ermöglicht. Grundlage der RIBO-seq-Technik ist die Tatsache, dass das Ribosom während der Translation das mRNA-Molekül bindet und so das vergrabene Fragment des Transkripts vor einer Ribonuklease-Verdauung schützt. Nach Zugabe der Ribonuklease wird die ungeschützte mRNA verdaut und die von Ribosomen eingeschlossenen Fragmente – typischerweise von ~28-30 nt lang – bleiben intakt. Diese Fragmente, die als ribosomale Fußabdrücke (RF) bezeichnet werden, können dann isoliert, sequenziert und auf das Transkript abgebildet werden, aus dem sie stammen, was zur Erkennung der genauen Position der Ribosomen führt. Tatsächlich wird die Ribosomenfähigkeit zum Schutz von mRNA-Fragmenten seit den 1960er Jahren verwendet, um ribosomale Bindungs- und Translationsinitiierungsstellen (TIS)2,3,4zu untersuchen. Mit dem Fortschritt in der Tiefensequenzierungstechnologie ist RIBO-seq jedoch zu einem Goldstandard für die Translationsüberwachunggeworden 5, der durch das Ribosomengagement genomweite Informationen über die Proteinsynthese liefern kann6. Ribosomenprofilierung füllte die technologische Lücke, die zwischen der Quantifizierung des Transkriptoms und des Proteoms bestand6.
Um ein Ribosomen-Profiling durchzuführen, müssen wir Zelllysat des Organismus erhalten, der unter den untersuchten Bedingungen gewachsen war. Die Störung dieser Bedingungen während der Zellentnahme und -lyse kann unzuverlässige Daten liefern. Um dies zu verhindern, werden häufig Translationsinhibitoren, schnelle Ernte und Schockgefrieren in flüssigem Stickstoff verwendet. Zellen können durch kryogenes Mahlen in einem mechanischen Homogenisator wie einer Mischermühle7,8 odereinem Perlenschläger9und durch Trituration durch eine Pipette10 oder mit einer Nadel11lysiert werden. Der Lysepuffer kann kurz vor oder kurz nach der Pulverisierung der Zellen zugegeben werden. In unserem Protokoll verwenden wir flüssigen Stickstoff zur Vorkühlung von Mörtel und Stößel sowie Aluminiumoxid als schonenderen Ansatz zur Störung der bakteriellen Zellwand, was eine RNA-Scherung verhindert, die häufig bei Methoden wie sonifikation auftritt. Nach der Pulverisierung fügen wir dem gekühlten Inhalt des Mörtels einen eiskalten Lysepuffer hinzu. Die Auswahl eines geeigneten Lysepuffers ist wichtig, um die beste Auflösung der ribosomalen Fußabdrücke zu erhalten. Da die Ionenstärke sowohl die HF-Größe als auch die Leserahmengenauigkeit beeinflusst, wird derzeit empfohlen, Lysepuffer mit geringer Ionenstärke und Pufferkapazität zu verwenden, auch wenn es den Anschein hat, dass die Pufferzusammensetzung die ribosomale Belegung auf mRNAs11,12nicht beeinflusst. Wichtige Bestandteile des Lysepuffers sind Magnesiumionen, deren Vorhandensein eine Dissoziation der ribosomalen Untereinheiten verhindert und spontane Konformationsveränderungen in den bakteriellen Ribosomen hemmt11,13. Calciumionen spielen ebenfalls eine bedeutende Rolle und sind essentiell für die Aktivität der Mikrokokkennuklease (MNase), die in der bakteriellen Ribosomenprofilierungsmethode verwendet wird14. Die Zugabe von Guanosin 5′-[β,γ-imido]triphosphat (GMP-PNP), einem nicht hydrolysierbaren Analogon von GTP, zusammen mit Chloramphenicol hemmt die Translation während der Lyse15.
Wenn das Lysat erhalten wird, wird es durch Zentrifugation geklärt und in zwei Portionen unterteilt, jeweils für eine RIBO-seq- und eine Hochdurchsatz-Gesamt-mRNA-Sequenzierung (RNA-seq), da sie gleichzeitig durchgeführt werden (Abbildung 1). RNA-seq bietet einen Bezugspunkt, der den Vergleich von Daten sowohl von RIBO-seq als auch von RNA-seq während der Datenanalyse ermöglicht. Das untersuchte Translatom wird durch Normalisierung ribosomaler Fußabdrücke zur mRNA-Häufigkeitdefiniert 16. Daten von RNA-seq können auch helfen, Klon- oder Sequenzierungsartefakte zu identifizieren17.
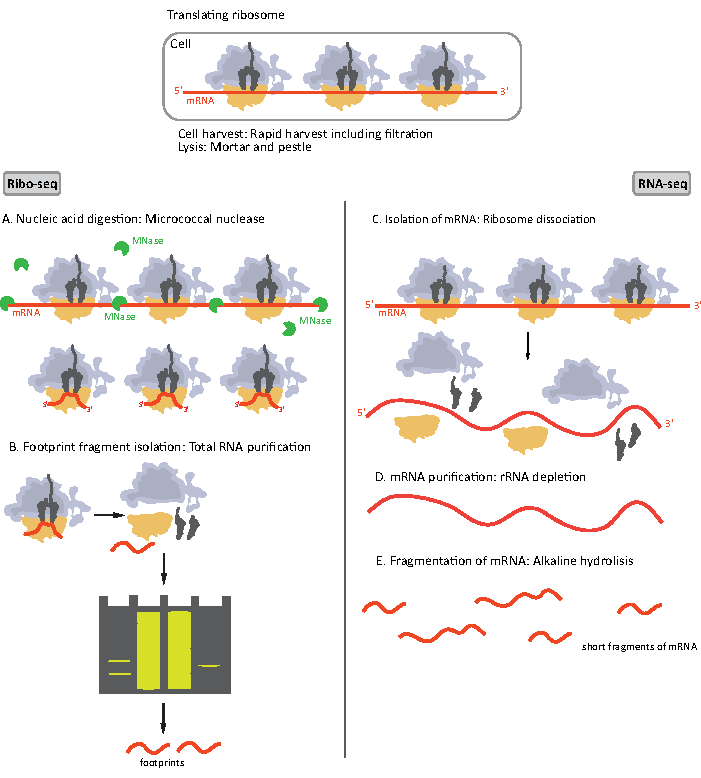
Abbildung 1. Schematische Darstellung der mRNA-Probenvorbereitung für RIBO-seq und RNA-seq. Für die RIBO-seq-Bibliotheksvorbereitung wird RNA mit MNase (A) verdaut, gefolgt von der Größenauswahl von RF von ~ 28-30 nt Länge (B); für RNA-seq wird RNA isoliert (C), von rRNA (D) erschöpft und die resultierende mRNA wird zufällig in Fragmente unterschiedlicher Länge (E) fragmentiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die ersten Schritte des Verfahrens der Probenvorbereitung für RIBO-seq und RNA-seq unterscheiden sich geringfügig (Abbildung 1). Für das ribosomale Profiling muss das Lysat durch eine spezifische Endonuklease verdaut werden, um die mRNA-Moleküle abzubauen, die nicht durch die Ribosomen geschützt sind. In Standardprotokollen werden die erhaltenen Monosomen durch eine Saccharosekissen-Ultrazentrifugation oder eine Saccharosegradienten-Ultrazentrifugation8,14zurückgewonnen. In diesem Artikel zeigen wir, dass dieser Schritt nicht notwendig ist, um RF zu isolieren, die für das RIBO-seq in Bakterien erforderlich sind, ebenso für eukaryotische Zellen18, und dass die Größenauswahl der geeigneten mRNA-Fragmente aus dem Polyacrylamid-Gel ausreichend ist.
Für RNA-seq wird mRNA durch die Erschöpfung von rRNA aus der gesamten RNA erhalten – rRNA-Moleküle hybridisieren zu den biotinylierten Oligonukleotidsonden, die an die Streptavidin-beschichteten magnetischen Perlen binden. Die rRNA-Oligonukleotid-Perlenkomplexe werden dann mit einem Magneten aus der Probeentfernt,was zu einer rRNA-erschöpften Probe19,20führt. Die gereinigten mRNA-Moleküle werden dann durch alkalische Hydrolyse zufällig fragmentiert. Die erhaltenen Fragmente der mRNA sowie die ribosomalen Fußabdrücke werden in cDNA-Bibliotheken umgewandelt und für die Tiefensequenzierung vorbereitet (Abbildung 2). Dies beinhaltet die Reparatur der Enden, die nach der alkalischen Hydrolyse (für mRNA) und der enzymatischen Verdauung (für RF) erforderlich ist: Dephosphorylierung von 3′-Enden, gefolgt von Phosphorylierung von 5′-Enden. Die nächsten Schritte sind die Adapterligatur und die umgekehrte Transkription, um cDNA-Inserts zu erstellen, die von Sequenzen umrahmt werden, die für die Next Generation Sequencing (NGS) mit der Illumina-Plattform erforderlich sind. Die letzte Phase der Bibliotheksvorbereitung ist eine PCR-Reaktion, bei der die Konstrukte amplifiziert und mit probenspezifischen Barcodes markiert werden, um multiplexen und sequenzieren zu können, verschiedene Proben auf einem Kanal zu sequenzieren. Vor der Sequenzierung werden Qualität und Quantität der Bibliotheken durch die hochempfindliche DNA-On-Chip-Elektrophorese bewertet. cDNA-Bibliotheken mit entsprechenden Parametern können dann gepoolt und sequenziert werden. Die Sequenzierung kann auf verschiedenen Illumina-Plattformen wie MiSeq, NextSeq oder HighSeq durchgeführt werden, abhängig von der Anzahl der Bibliotheken, der erforderlichen Leselänge und der Sequenzierungstiefe. Nach der Sequenzierung wird die bioinformatische Analyse durchgeführt.

Abbildung 2. Bibliotheksvorbereitung. Die Bibliotheksvorbereitung umfasst die Reparatur der Enden, die Ligatur der Adapter, die umgekehrte Transkription und die Verstärkung mit Barcoding. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die Ribosomenprofilierung ist eine universelle Methode, die sich leicht modifizieren und entsprechend der wissenschaftlichen Fragestellung anpassen lässt. Ursprünglich wurde es in Hefe1verwendet, aber kurz darauf wurde es auf Bakterienzellen21 sowie eukaryotische Modellorganismen wie Maus10,Zebrafisch22,Fruchtfliege23 und Arabidopsis thaliana24aufgetragen. Es wurde auch für die Untersuchung verschiedener Ribosomentypen verwendet: zytoplasmatische, mitochondriale25,26 und Chloroplast27,28. Bei Eukaryoten wird RIBO-seq üblicherweise angepasst und verfeinert, um spezifische Aspekte der Translation zu untersuchen, einschließlich Initiation10,11,29,30,31,32, Dehnung1,10,11,31,33, Ribosomenabwürgen33 und Konformationsänderung33. Die meisten Modifikationen beinhalten die Verwendung verschiedener Translationsinhibitoren. Bei Bakterien waren analoge Studien jedoch aufgrund des Mangels an Inhibitoren mit dem erforderlichen Wirkmechanismus schwierigdurchzuführen 34. Der am häufigsten verwendete Translationsinhibitor in Bakterien ist Chloramphenicol (CAM), das an das Peptidyltransferase-Zentrum (PTC) bindet und eine korrekte Positionierung der Aminoacyl-tRNA in der A-Stelle verhindert. Infolgedessen verhindert CAM die Bildung einer Peptidbindung, die dazu führt, dass die länglichen Ribosomen35blockiert werden. Weitere Beispiele für Translationsinhibitoren in Bakterien sind Tetracyclin (TET)36, Retapamulin (RET)34 und Onc11237, die zur Untersuchung von Translationsinitiierungsstellen verwendet wurden. TET, das die tRNA-Abgabe an das Ribosom verhindert, indem es sich direkt mit der Anticodon-Stammschleife der tRNA an der A-Stelle überschneidet, wurde ursprünglich angewendet, um die Ergebnisse der CAM-Behandlung zu überprüfen, da beide Antibiotika sind, die die Translationsdehnung hemmen38. Es wurde festgestellt, dass TET primäre TIS erkennt, konnte jedoch kein internes TIS36aufdecken. RET bindet im PTC des bakteriellen Ribosoms und verhindert die Bildung der ersten Peptidbindung, indem es mit einer Elongator-Aminoacyl-tRNA an der A-Stelle interferiert. Die Anwendung von RET führt zu einem Ribosomen-Arrest sowohl bei primären als auch bei internen TISs34. Onc112, ein prolinreiches antimikrobielles Peptid, bindet im Austrittstunnel und blockiert die Aminoacyl-tRNA-Bindung an der ribosomalen A-Stelle. Dadurch verhindert Onc112, dass Initiationskomplexe in die Dehnungsphase37eintreten.
Die Hauptinformation, die das Ribosomen-Profiling liefert, ist die Ribosomendichte und ihre Position auf der mRNA. Es wurde erfolgreich angewendet, um die differentielle Genexpression auf der Ebene der Translation in verschiedenen Wachstumsbedingungen1,6zu untersuchen, die translationale Effizienz1,38,39 zu messen und Translationsregulationsereignisse wie ribosomale Pausierung10zu erkennen. RIBO-seq ermöglicht auch die Aufdeckung der Translation von annotierter ncRNA, Pseudogenen und unangekündigten kleinen offenen Leserahmen (ORF), die zur Identifizierung neuer und/oder sehr kurzer proteinkodierender Gene10,12,22,30,37führen. In solchen Fällen kann RIBO-seq die Genomannotation verfeinern und verbessern. Mit seiner hohen Sensitivität für die Identifizierung übersetzter ORFs und seiner quantitativen Natur kann das Ribosomen-Profiling auch als Proxy für die Proteombestimmung oder zur Unterstützung von Proteomik-Studiendienen 31,34,39. Durch die Kartierung von TIS zeigt das Ribosomen-Profiling N-terminal ausgedehnte und abgeschnittene Isoformen bekannter Proteine10,32. RIBO-seq wurde auch angepasst, um die co-translationale Faltung der Proteine14,21,24zu untersuchen. Diese Methode ermöglicht die Messung von Dehnungsraten1,10,39 oder Dekodierungsgeschwindigkeiten einzelner Codons6 und hilft bei der Entwicklung quantitativer Modelle der Übersetzung17. Die Ribosomen-Profiling-Methode ist auch in der Lage, mechanistische Einblicke in die Ribosomen-Pause bei Bakterien7,15,17, Frameshifting40, Stop-Codon-Readthrough21, Abschluss- / Recyclingdefekte41,42 und ribosomale Konformationsänderungen33 in Eukaryoten zu liefern. RIBO-seq wurde auch angepasst, um den Einfluss spezifischer trans-wirkender Faktoren auf die Translation wie miRNAs6 und RNA-bindende Proteine in Eukaryoten16,43zuuntersuchen. Es ist jedoch wichtig anzuerkennen, dass das experimentelle Design und die erhaltene Auflösung von RIBO-seq die Menge an Informationen bestimmen, die aus den resultierenden Sequenzierungsdaten extrahiert werden können12.
Protocol
Representative Results
Discussion
Die wichtigste technische Herausforderung des Ribosomen-Profilings besteht in der Notwendigkeit, die Translation schnell zu hemmen, um eine Momentaufnahme von Ribosomen auf mRNAs in einem bestimmten physiologischen Zustand von Interesse zu erfassen. Um dies zu erreichen, werden häufig Translationsinhibitoren, schnelle Ernte und Schockgefrieren in flüssigem Stickstoff verwendet. Die Anwendung von Antibiotika ist optional, da sie Artefakte verursachen können. Chloramphenicol ist ein häufig verwendetes Medikament, um l?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
ALS möchte die finanzielle Unterstützung der EMBO Installation Grants IG 3914 und POIR anerkennen. 04.04.00-00-3E9C/17-00 durchgeführt im Rahmen des Programms First TEAM der Stiftung für polnische Wissenschaft, das von der Europäischen Union im Rahmen des Europäischen Fonds für regionale Entwicklung kofinanziert wird.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Riferimenti
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

