RIBO-seq בחיידקים: אוסף לדוגמה ופרוטוקול הכנת ספריה לריצוף NGS
Summary
כאן אנו מתארים את שלבי איסוף המדגם והכנת RIBO-seq בחיידקים. רצף הספריות שהוכנו על פי הנחיות אלה מביא לנתונים מספיקים לניתוח ביואינפורמטי מקיף. הפרוטוקול שאנו מציגים הוא פשוט, משתמש בציוד מעבדה סטנדרטי ולוקח שבעה ימים מן התזה להשגת ספריות.
Abstract
טכניקת פרופיל הריבוזום (RIBO-seq) היא כיום הכלי היעיל ביותר לחקר תהליך סינתזת החלבון ב vivo. היתרון של שיטה זו, בהשוואה לגישות אחרות, הוא היכולת שלה לפקח על התרגום על ידי מיפוי מדויק של המיקום ומספר הריבוזומים בתעתיק mRNA.
במאמר זה אנו מתארים את השלבים העוקבים של איסוף והכנה לדוגמה לשיטת RIBO-seq בחיידקים, ומדגישים את הפרטים הרלוונטיים לתכנון וביצוע הניסוי.
מאז RIBO-seq מסתמך על ריבוזומים שלמים mRNAs הקשורים, הצעד העיקרי הוא עיכוב מהיר של תרגום התפוררות נאותה של תאים. לכן, אנו מציעים סינון והקפאת פלאש בחנקן נוזלי לקצירת תאים עם טיפול מקדים אופציונלי עם כלוראמפניקול כדי לעצור תרגום בחיידקים. להתפוררות, אנו מציעים שחיקה תאים קפואים עם מרגמה ועלי בנוכחות תחמוצת אלומיניום כדי לשבש מכנית את דופן התא. בפרוטוקול זה, כרית סוכרוז או שיפוע סוכרוז ultracentrifugation עבור טיהור מונוזום אינו נדרש. במקום זאת, הפרדת mRNA באמצעות אלקטרופורזה ג’ל פוליאקרילמיד (PAGE) ואחריו כריתת טביעת הרגל ריבוזומלית (28-30 nt הלהקה) מוחל ומספק תוצאות משביעות רצון. פעולה זו מפשטת במידה רבה את השיטה וכן מפחיתה את דרישות הזמן והציוד עבור ההליך. להכנת הספרייה, אנו ממליצים להשתמש בערכת RNA קטנה הזמינה מסחרית לריצוף Illumina מניו אינגלנד Biolabs, בהתאם להנחיות היצרן עם מידה מסוימת של אופטימיזציה.
ספריות ה- cDNA המתקבלות מציגות כמות ואיכות מתאימות הנדרשות לריצוף הדור הבא (NGS). רצף הספריות שהוכנו על פי תוצאות הפרוטוקול המתוארות ב 2 עד 10 mln קריאות ממופה באופן ייחודי לכל מדגם מתן נתונים מספיקים לניתוח ביואינפורמטי מקיף. הפרוטוקול שאנו מציגים מהיר וקל יחסית וניתן לבצעו באמצעות ציוד מעבדה סטנדרטי.
Introduction
טכניקת פרופיל הריבוזום (RIBO-seq) פותחה במעבדתו של ג’ונתן וייסמן באוניברסיטת קליפורניה בסן פרנסיסקו1. בהשוואה לשיטות אחרות המשמשות לחקר ביטוי גנים ברמה התרגומית, RIBO-seq מתמקד בכל מחייב ריבוזום ל- mRNA ומספק מידע על מיקומו ועל המספר היחסי של ריבוזומים בתעתיק. זה מאפשר ניטור התהליך של סינתזת חלבון vivo והוא יכול לספק רזולוציה קודון יחיד ודיוק המאפשר מדידה של צפיפות ריבוזום על שניהם, mRNA הפרט לאורך התמלול כולו בתא. בבסיס טכניקת RIBO-seq טמונה העובדה כי במהלך התרגום הריבוזום קושר את מולקולת mRNA ובכך מגן על החלק הקבור של התמליל מעיכול ריבונוקלאאז. עם תוספת של ribonuclease, mRNA לא מוגן מתעכל ואת שברים מוקף ריבוזומים – בדרך כלל של ~ 28-30 nt ארוך – להישאר שלם. שברים אלה, הנקראים עקבות ריבוזומליות (RF), יכולים להיות מבודדים, רציפים וממופים לתעתיק שמקורו בזיהוי המיקום המדויק של הריבוזומים. למעשה, היכולת הריבוזום להגן על שברי mRNA שימש מאז 1960 ללמוד אתרי ייזום מחייב ותרגום ריבוזומלי (TIS)2,3,4. עם זאת, עם ההתקדמות בטכנולוגיית רצף עמוק, RIBO-seq הפך תקן זהב לניטור תרגום5 אשר, באמצעות מעורבות ריבוזום, יכול לספק מידע גנומי רחב על סינתזת חלבונים6. פרופיל ריבוזום מילא את הפער הטכנולוגי שהיה קיים בין כימות התמלול לבין פרוטאום6.
כדי לנהל פרופיל ריבוזום אנחנו צריכים להשיג ליזאט התא של האורגניזם שגדל בתנאים שנחקרו. שיבוש תנאים אלה במהלך איסוף תאים ותמוגה עשוי לספק נתונים לא אמינים. כדי למנוע זאת, מעכבי תרגום, קציר מהיר והקפאת פלאש בחנקן נוזלי משמשים בדרך כלל. תאים יכולים להיות lysed על ידי שחיקה קריוגנית homogenizer מכני כמו טחנת מערבל7,8 או מקצף חרוזים9, ועל ידי טריטורציה באמצעות פיפטה10 או עם מחט11. מאגר התמוגה ניתן להוסיף רק לפני או זמן קצר לאחר ריסוק של התאים. בפרוטוקול שלנו אנו משתמשים חנקן נוזלי כדי מרגמה precool ועלי, כמו גם תחמוצת אלומיניום כגישה עדינה יותר להפרעה של דופן התא החיידקי, אשר מונע גזירה RNA לעתים קרובות נתקל כאשר שיטות כגון sonification מוחלים. לאחר ריסוק, אנו מוסיפים חיץ תמוגה קר כקרח לתכולת המרגמה מקוררת. בחירה של מאגר תמוגה מתאים חשוב להשגת הרזולוציה הטובה ביותר של עקבות ריבוזומליות. מאז חוזק יוני משפיע הן על גודל RF ואת דיוק מסגרת הקריאה, מומלץ כיום להשתמש מאגרי תמוגה עם חוזק יוני נמוך קיבולת חיץ, גם אם נראה כי הרכב המאגר אינו משפיע על תפוסה ריבוזואלית על mRNAs11,12. מרכיבים חשובים של מאגר התמוגה הם יוני מגנזיום, שנוכחותם מונעת ניתוק של יחידות המשנה הריבוזומליות ומעכבת שינויים קונפורמיים ספונטניים בריבוזומים החיידקיים11,13. יוני סידן גם לשחק תפקיד משמעותי והם חיוניים לפעילות של גרעין מיקרוקוקאלי (MNase) המשמש בשיטת פרופיל ריבוזום חיידקי14. תוספת של גואנוסין 5′-[β,γ-imido]טריפוספט (GMP-PNP), אנלוגי שאינו הידרוליזה של GTP, יחד עם כלוראמפניקול מעכב תרגום במהלך תמוגה15.
כאשר מתקבל הליסנט, מובהר על ידי צנטריפוגה ומחולק לשני חלקים, כל אחד עבור RIBO-seq ורצף mRNA כולל בעל תפוקה גבוהה (RNA-seq) שכן הם מבוצעים בו זמנית (איור 1). RNA-seq מספק נקודת התייחסות המאפשרת השוואה של נתונים הן RIBO-seq והן RNA-seq במהלך ניתוח נתונים. התרגום שנחקר מוגדר על ידי נורמליזציה של עקבות ריבוזומליות לשפע mRNA16. נתונים מ-RNA-seq יכולים גם לסייע בזיהוי שיבוט או רצף של חפצים17.
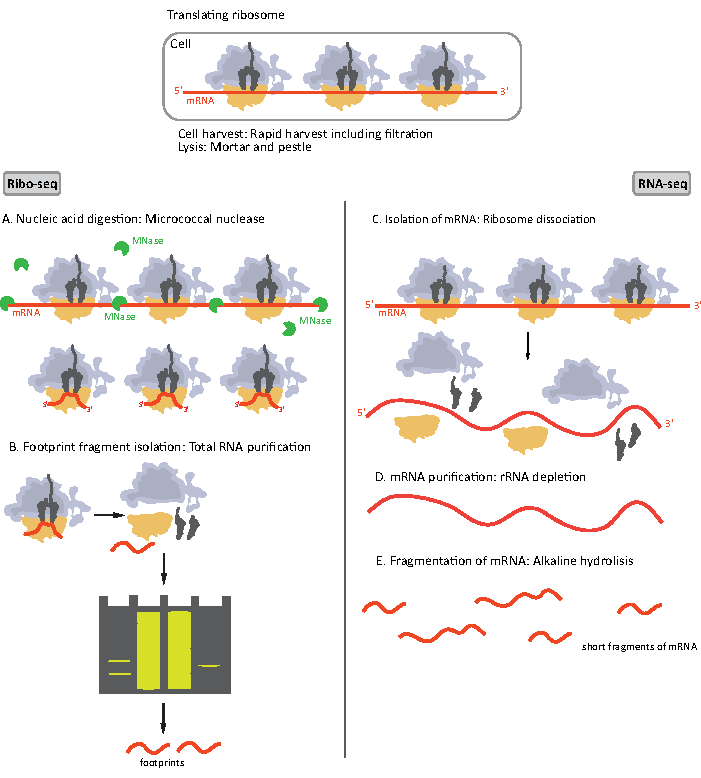
איור 1. שרטוטים של הכנת דגימת mRNA עבור ריבו-seq ו- RNA-seq. להכנת ספריית RIBO-seq, RNA מתעכל עם MNase (A), ואחריו מבחר הגודל של RF של ~ 28-30 nt אורך (B); עבור RNA-seq RNA מבודד (C), מדולדל של rRNA (D), ואת mRNA וכתוצאה מכך הוא מקוטע באופן אקראי לרסיסים באורכים משתנים (E). לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
השלבים הראשוניים של הליך ההכנה לדוגמה עבור RIBO-seq ו- RNA-seq שונים במקצת (איור 1). עבור פרופיל ריבוזומלי, lysate צריך להיות מתעכל על ידי אנדונוקלאאז מסוים כדי להשפיל את מולקולות mRNA לא מוגן על ידי ריבוזומים. בפרוטוקולים סטנדרטיים, מונוזומים המתקבלים הם התאוששו על ידי אולטרה צנטריפוגציה כרית סוכרוז או אולטרה צנטריפוגציה שיפוע סוכרוז8,14. במאמר זה, אנו מראים כי שלב זה אינו הכרחי כדי לבודד RF הנדרש עבור RIBO-seq בחיידקים, כמו כן עבור תאים אוקריוטיים18, וכי מבחר גודל של שברי mRNA אורך המתאים מן ג’ל polyacrylamide מספיק.
עבור RNA-seq, mRNA מתקבל על ידי דלדול של rRNA מן RNA הכולל – מולקולות rRNA הכלאה לבדיקות אוליגונוקלאוטיד biotinylated אשר נקשרים חרוזים מגנטיים מצופים סטרפטבידין. קומפלקסים rRNA-oligonucleotide-חרוזים מוסרים לאחר מכן מן המדגם עם מגנט וכתוצאה מכך מדגם מדולדל rRNA19,20. מולקולות mRNA מטוהרים לאחר מכן מפוצלים באופן אקראי על ידי הידרוליזה אלקליין. שברי ה-mRNA המתקבלים, כמו גם עקבות הריבוזומליות, מומרים לספריות cDNA ומוכנים לריצוף עמוק(איור 2). זה כרוך בתיקון מסתיים הדרוש לאחר הידרוליזה אלקליין (עבור mRNA) ועיכול אנזימטי (עבור RF): dephosphorylation של 3 ‘ קצוות ואחריו זרחון של קצוות 5 ‘. השלבים הבאים הם קשירת מתאמים ואת התמלול ההפוך כדי ליצור מוסיף cDNA ממוסגר על ידי רצפים הנדרשים עבור ריצוף הדור הבא (NGS) באמצעות פלטפורמת Illumina. השלב האחרון של הכנת הספרייה הוא תגובת PCR שבה המבנים מוגברים ומתויגים עם ברקודים ספציפיים לדוגמה כדי לאפשר multiplexing רצף דגימות שונות בערוץ אחד. לפני הרצף, האיכות והכמות של הספריות מוערכים על ידי אלקטרופורזה DNA רגישות גבוהה על השבב. לאחר מכן ניתן לאגד ולרצף ספריות cDNA עם פרמטרים מתאימים. ניתן לבצע רצף בפלטפורמות שונות של Illumina, כגון MiSeq, NextSeq או HighSeq, בהתאם למספר הספריות, אורך הקריאה הנדרש ועומק הרצף. לאחר הרצף מבוצע הניתוח הביואינפורמטי.

איור 2. הכנת ספרייה. הכנת הספרייה כוללת את תיקון הקצוות, קשירת מתאמים, שעתוק הפוך והגברה עם ברקודינג. לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
פרופיל הריבוזום הוא שיטה אוניברסלית אשר ניתן לשנות ולהתאים בקלות על פי השאלה המדעית. במקור הוא שימש שמרים1, אבל זמן קצר לאחר מכן הוחל על תאים חיידקיים21, כמו גם אורגניזמים מודל אוקריוטי כולל עכבר10, זברה דג22, זבוב פירות23 ו Arabidopsis thaliana24. הוא שימש גם לחקר סוגים שונים של ריבוזומים: ציטופלסמי, מיטוכונדריאלי25,26 וכלורופלסט27,28. באיקריוטים RIBO-seq מותאם ומעודן בדרך כלל כדי לחקור היבטים ספציפיים של התרגום, כולל חניכה10,11,29,30,31,32, התארכות 1,10,11,31,33, ריבוזום השתהות33 ושינוי קונפורמציה33. רוב השינויים כרוכים בשימוש במעכבי תרגום שונים. בחיידקים עם זאת, מחקרים מקבילים היו קשים לביצוע בגלל מחסור במעכבים עם מנגנון הפעולה הנדרש34. מעכב התרגום הנפוץ ביותר בחיידקים הוא כלוראמפניקול (CAM) הנקשר למרכז הפפטידיל טרנספראז (PTC) ומונע מיקום נכון של האמינואציל-tRNA באתר A. כתוצאה מכך, CAM מונע היווצרות של קשר פפטיד אשר מוביל למעצר ריבוזומים מוארכים35. דוגמאות אחרות של מעכבי תרגום בחיידקים הם טטרציקלין (TET)36, retapamulin (RET)34 ו Onc11237 אשר שימשו כדי לחקור אתרי ייזום תרגום. TET, אשר מונע משלוח tRNA לריבוזום על ידי חפיפה ישירה עם לולאת גזע אנטיקודון של tRNA באתר A, הוחל במקור כדי לאמת את התוצאות שהתקבלו מטיפול CAM שכן שניהם אנטיביוטיקה מעכבת התארכות תרגום38. TET נמצא לזהות TIS ראשי, עם זאת לא הצליח לחשוף פנימי TIS36. RET נקשר PTC של ריבוזום חיידקי, ומונע היווצרות של הקשר פפטיד הראשון על ידי הפרעה עם aminoacyl-tRNA מוארך באתר. החלת תוצאות RET במעצר ריבוזומים הן ראשי, כמו גם פנימי TISS34. Onc112, פפטיד אנטי מיקרוביאלי עשיר בפרולין, נקשר במנהרת היציאה וחוסם את כריכת aminoacyl-tRNA באתר ריבוזומלי A. כתוצאה מכך, Onc112 מונע ממתחמי חניכה להיכנס לשלב התארכות37.
המידע העיקרי פרופיל ריבוזום מספק הוא צפיפות ריבוזומים ואת מיקומם על mRNA. זה יושם בהצלחה לחקור ביטוי גנים דיפרנציאליים ברמת התרגום בתנאי צמיחה שונים1,6, למדוד יעילות תרגום1,38,39 ולזהות אירועי ויסות תרגום כגון השהייה ריבוזומלית10. RIBO-seq מאפשר גם לחשוף את התרגום של ncRNA ביאורים, פסאודוגנים ומסגרות קריאה פתוחות קטנות ללא ביאורים (ORF) המוביל לזיהוי של גנים קידוד חלבון הרומן ו / או קצר מאוד10,12,22,30,37. במקרים כאלה, RIBO-seq יכול לכוונן ולשפר ביאור הגנום. עם הרגישות הגבוהה שלה לזיהוי של ORFs מתורגם ואופיו הכמותי, פרופיל ריבוזום יכול לשמש גם פרוקסי לקביעת פרוטאום או בסיוע מחקרים פרוטאומיקים31,34,39. על ידי מיפוי TIS, פרופיל ריבוזום חושף N-סופני מורחב וקצץ isoforms של חלבונים ידועים10,32. RIBO-seq הותאם גם ללמוד קיפול תרגום משותף של חלבונים14,21,24. שיטה זו מאפשרת מדידה של שיעורי התארכות1,10,39 או מהירויות פענוח של קודונים בודדים6 ומסייעת בפיתוח מודלים כמותיים של תרגום17. שיטת פרופיל ריבוזום הוא גם מסוגל לספק תובנות מכניות לתוך הריבוזום השהיה בחיידקים7,15,17, frameshifting40, עצור קודון readthrough21, סיום / מיחזור פגמים41,42 ו שינויים קונפורמציה ריבוזומלית33 באיקריוטים. RIBO-seq הותאם גם כדי לבחון את ההשפעה של גורמים טרנס-משחקים ספציפיים על תרגום כגון miRNAs6 וחלבונים מחייבי RNA באיקריוטים16,43. עם זאת, חשוב להכיר בכך שהתכנון הניסיוני והרזולוציה המתקבלת של RIBO-seq קובעים את כמות המידע שניתן לחלץ מנתוני הרצףהמתקבלים 12.
Protocol
Representative Results
Discussion
האתגר הטכני העיקרי של פרופיל הריבוזום הוא הצורך לעכב במהירות את התרגום על מנת ללכוד תמונת מצב של ריבוזומים על mRNAs במצב פיזיולוגי מסוים של עניין. כדי להשיג זאת, מעכבי תרגום, קציר מהיר והקפאת פלאש בחנקן נוזלי משמשים בדרך כלל. החלת אנטיביוטיקה היא אופציונלית שכן הם יכולים לגרום חפצים. כלוראמפ?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
ALS רוצה להכיר בתמיכה הכספית של EMBO התקנה מענקים IG 3914, ו POIR. 04.04.00-00-3E9C/17-00 שבוצע במסגרת תוכנית TEAM הראשונה של הקרן למדע פולני במימון משותף של האיחוד האירופי תחת הקרן האירופית לפיתוח אזורי.
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Riferimenti
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

