बैक्टीरिया में RIBO-seq: NGS अनुक्रमण के लिए एक नमूना संग्रह और पुस्तकालय तैयारी प्रोटोकॉल
Summary
यहां हम बैक्टीरिया में रिबो-सेक्यू के लिए नमूना संग्रह और तैयारी के चरणों का वर्णन करते हैं। इन दिशा-निर्देशों के अनुसार तैयार पुस्तकालयों के अनुक्रमण के परिणामस्वरूप व्यापक जैव सूचना विश्लेषण के लिए पर्याप्त डेटा होता है । प्रोटोकॉल हम मौजूद सरल है, मानक प्रयोगशाला उपकरण का उपयोग करता है और पुस्तकालयों को प्राप्त करने के लिए लाइसिस से सात दिन लगते हैं ।
Abstract
राइबोसोम प्रोफाइलिंग तकनीक (RIBO-seq) वर्तमान में वीवो मेंप्रोटीन संश्लेषण की प्रक्रिया का अध्ययन करने के लिए सबसे प्रभावी उपकरण है। इस विधि का लाभ, अन्य दृष्टिकोणों की तुलना में, एमआरएनए ट्रांसक्रिप्ट पर राइबोसोम्स की स्थिति और संख्या को ठीक से मैप करके अनुवाद की निगरानी करने की क्षमता है।
इस लेख में, हम नमूना संग्रह के लगातार चरणों और बैक्टीरिया में RIBO-seq विधि के लिए तैयारी का वर्णन करते हैं, जो प्रयोग की योजना और निष्पादन के लिए प्रासंगिक विवरणों को उजागर करते हैं।
चूंकि RIBO-seq बरकरार राइबोसोम्स और संबंधित mRNAs पर निर्भर करता है, महत्वपूर्ण कदम अनुवाद और कोशिकाओं के पर्याप्त विघटन के तेजी से अवरोध है । इस प्रकार, हम बैक्टीरिया में अनुवाद को रोकने के लिए क्लोरम्फेनिकोल के साथ एक वैकल्पिक पूर्वउपचार के साथ सेल हार्वेस्टिंग के लिए तरल नाइट्रोजन में छानने और फ्लैश-फ्रीजिंग का सुझाव देते हैं। विघटन के लिए, हम यांत्रिक रूप से सेल की दीवार को बाधित करने के लिए एल्यूमीनियम ऑक्साइड की उपस्थिति में मोर्टार और मूसल के साथ जमे हुए कोशिकाओं को पीसने का प्रस्ताव करते हैं। इस प्रोटोकॉल में, मोनोसोम शुद्धिकरण के लिए सुक्रोज कुशन या सुक्रोज रेडिएंट अल्ट्रासेंट्रफ्यूजन की आवश्यकता नहीं है। इसके बजाय, पॉलीएक्रीलैमाइड जेल इलेक्ट्रोफोरेसिस (पेज) का उपयोग करके एमआरएनए सेपरेशन लागू किया जाता है और इसके बाद राइबोसोमल पदचिह्न एक्ससेशन (28-30 एनटी बैंड) लागू होता है और संतोषजनक परिणाम प्रदान करता है। यह काफी हद तक विधि को सरल बनाता है और साथ ही प्रक्रिया के लिए समय और उपकरण आवश्यकताओं को कम करता है। पुस्तकालय की तैयारी के लिए, हम कुछ हद तक अनुकूलन के साथ निर्माता के दिशानिर्देशों का पालन करते हुए न्यू इंग्लैंड बायोलैब्स से इलुमिना अनुक्रमण के लिए व्यावसायिक रूप से उपलब्ध छोटे आरएनए किट का उपयोग करने की सलाह देते हैं।
परिणामस्वरूप सीडीएनए पुस्तकालयों उचित मात्रा और अगली पीढ़ी अनुक्रमण (NGS) के लिए आवश्यक गुणवत्ता मौजूद हैं । 2 से 10 एमएलएन विशिष्ट रूप से मैप किए गए नमूने में वर्णित प्रोटोकॉल परिणामों के अनुसार तैयार पुस्तकालयों की अनुक्रमण व्यापक जैव सूचनात्मक विश्लेषण के लिए पर्याप्त डेटा प्रदान करने वाले प्रति नमूने को पढ़ता है। प्रोटोकॉल हम मौजूद है जल्दी और अपेक्षाकृत आसान है और मानक प्रयोगशाला उपकरणों के साथ किया जा सकता है ।
Introduction
राइबोसोम प्रोफाइलिंग तकनीक (RIBO-seq) कैलिफोर्निया विश्वविद्यालय, सैन फ्रांसिस्को1में जोनाथन Weissman की प्रयोगशाला में विकसित किया गया था । ट्रांसलेशनल स्तर पर जीन अभिव्यक्ति का अध्ययन करने के लिए उपयोग किए जाने वाले अन्य तरीकों की तुलना में, RIBO-seq एमआरएनए के लिए प्रत्येक राइबोसोम बाध्यकारी पर केंद्रित है और इसके स्थान और ट्रांसक्रिप्ट पर राइबोसोम की सापेक्ष संख्या के बारे में जानकारी प्रदान करता है। यह वीवो में प्रोटीन संश्लेषण की प्रक्रिया की निगरानी करने में सक्षम बनाता है और एकल कोडन रिज़ॉल्यूशन और सटीकता प्रदान कर सकता है जो दोनों पर राइबोसोम घनत्व के माप की अनुमति देता है, व्यक्तिगत एमआरएनए और सेल में पूरे ट्रांसक्रिप्टोम के साथ। RIBO-seq तकनीक की नींव पर तथ्य यह है कि अनुवाद के दौरान राइबोसोम एमआरएनए अणु को बांधता है और इस प्रकार एक राइबोन्यूलीज पाचन से ट्रांसक्रिप्ट के दफन टुकड़े की रक्षा करता है। राइबोन्यूलेज के अलावा, असुरक्षित एमआरएनए पचा जाता है और राइबोसोम्स से घिरे टुकड़े – आमतौर पर ~ 28-30 एनटी लंबे समय तक – बरकरार रहते हैं। इन टुकड़ों, जिसे राइबोसोमल पैरों के निशान (आरएफ) कहा जाता है, को तब अलग किया जा सकता है, अनुक्रमित किया जा सकता है और ट्रांसक्रिप्ट पर मैप किया जा सकता है जिसके परिणामस्वरूप राइबोसोम्स की सटीक स्थिति का पता लगाया जा सकता है। वास्तव में, 1960 के दशक से राइबोसोम के टुकड़ों की रक्षा करने की क्षमता का उपयोग रिबोसोमल बाइंडिंग और अनुवाद दीक्षा स्थलों (टीआई)2,3,4का अध्ययन करने के लिए किया गया है। हालांकि, गहरी अनुक्रमण प्रौद्योगिकी में उन्नति के साथ, RIBO-seq अनुवाद निगरानी5 के लिए एक स्वर्ण मानक बन गया है, जो राइबोसोम सगाई के माध्यम से, प्रोटीन संश्लेषण6पर जीनोम-व्यापी जानकारी प्रदान कर सकता है। राइबोसोम प्रोफाइलिंग ने ट्रांसक्रिप्टोम और प्रोटेम6की मात्रा निर्धारित करने के बीच मौजूद तकनीकी अंतर को भर दिया ।
राइबोसोम प्रोफाइलिंग का संचालन करने के लिए हमें उस जीव की कोशिका लाइसेट प्राप्त करने की आवश्यकता होती है जो जांच की गई परिस्थितियों में बड़ा हो गया था। सेल संग्रह और लाइसिस के दौरान इन स्थितियों को बाधित करने से अविश्वसनीय डेटा प्रदान किया जा सकता है। इसे रोकने के लिए, अनुवाद अवरोधक, तेजी से कटाई और तरल नाइट्रोजन में फ्लैश फ्रीजिंग का आमतौर पर उपयोग किया जाता है। कोशिकाओं को मिक्सर मिल 7,8 या मनका बीटर 9 जैसे यांत्रिक समरूप में क्रायोजेनिक पीसकर और पिपेट10के माध्यम से या सुई11 के माध्यम से त्रयी द्वारा किया जा सकता है। कोशिकाओं के पल्वराइजेशन से ठीक पहले या कुछ ही समय बाद लाइसिस बफर जोड़ा जा सकता है। हमारे प्रोटोकॉल में हम प्रीकूल मोर्टार और मूसल के लिए तरल नाइट्रोजन का उपयोग करते हैं, साथ ही एल्यूमीनियम ऑक्साइड बैक्टीरियल सेल दीवार के व्यवधान के लिए एक सज्जन दृष्टिकोण के रूप में, जो आरएनए कतरनी को अक्सर रोकता है जब सोनिफिकेशन जैसे तरीके लागू होते हैं। पल्वराइजेशन के बाद, हम मोर्टार की ठंडी सामग्री में एक बर्फ-ठंडी लाइसिस बफर जोड़ते हैं। राइबोसोमल पैरों के निशान का सबसे अच्छा संकल्प प्राप्त करने के लिए एक उपयुक्त लाइसिस बफर का चयन महत्वपूर्ण है। चूंकि आयनिक ताकत आरएफ आकार और पढ़ने के फ्रेम परिशुद्धता दोनों को प्रभावित करती है, इसलिए वर्तमान में कम आयनिक शक्ति और बफर क्षमता के साथ लाइसिस बफर का उपयोग करने की सिफारिश की जाती है, भले ही ऐसा लगता है कि बफर संरचना mRNAs11, 12पर रिबोसोमल अधिभोग को प्रभावित नहीं करती है। लाइसिस बफर के महत्वपूर्ण घटक मैग्नीशियम आयन हैं, जिनकी उपस्थिति राइबोसोमल सबयूनिट के वियोजन को रोकती है और बैक्टीरियल राइबोसोम्स11, 13में सहज अनुरूप परिवर्तनों को रोकती है। कैल्शियम आयन भी महत्वपूर्ण भूमिका निभाते हैं और बैक्टीरियल राइबोसोम प्रोफाइलिंग विधि में उपयोग किए जाने वाले माइक्रोकोकल न्यूक्लियज़ (एमएनएज) की गतिविधि के लिए आवश्यक हैं14। गुआनोसिन 5 के अलावा-[β,γ-इमिडो] ट्रिप्होस्फेट (जीएमपी-पीएनपी), जीटीपी का एक गैर-हाइड्रोलिजेबल एनालॉग, क्लोरम्फेनिकोल के साथ-साथ लाइसिस15के दौरान अनुवाद को रोकता है ।
जब लिस्नेट प्राप्त किया जाता है, तो इसे अपकेंद्रित्र द्वारा स्पष्ट किया जाता है और दो भागों में विभाजित किया जाता है, प्रत्येक को रीबो-सेक्यू और एक उच्च-थ्रूपुट कुल एमआरएनए अनुक्रमण (आरएनए-एसईक्यू) के लिए क्योंकि वे एक साथ किए जाते हैं(चित्र 1)। आरएनए-एसईक्यू संदर्भ का एक बिंदु प्रदान करता है जो डेटा विश्लेषण के दौरान रिबो-सेक्यू और आरएनए-सेक्यू दोनों से डेटा की तुलना में सक्षम बनाता है। जांच किए गए अनुवाद को राइबोसोमल पैरों के निशान को सामान्य बनाकर एमआरएनए बहुतायतमें परिभाषितकिया गया है । आरएनए-एसईक्यू के डेटा से कलाकृतियों17की क्लोनिंग या अनुक्रमण की पहचान करने में भी मदद मिल सकती है ।
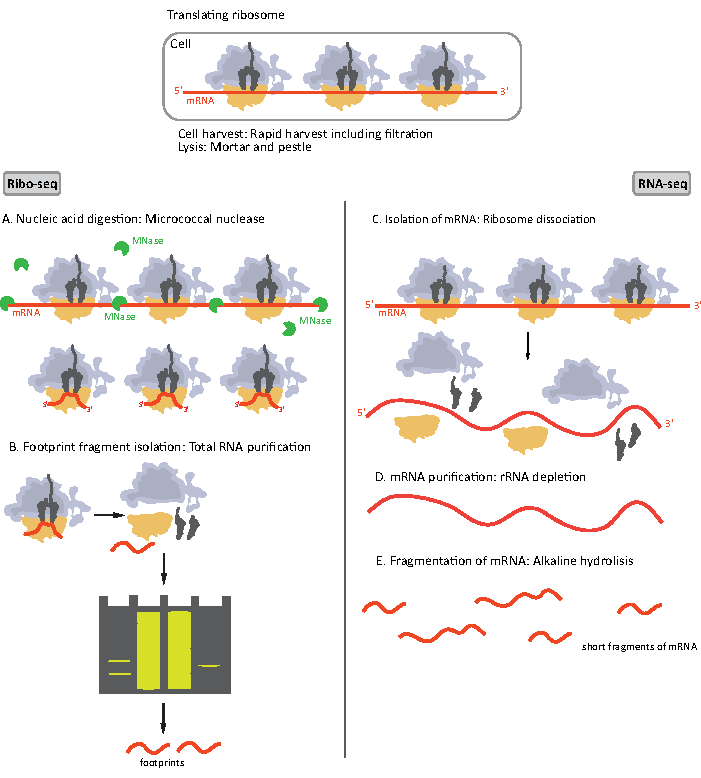
चित्रा 1। RIBO-seq और आरएनए-seq के लिए mRNA नमूना तैयारी की योजनाबद्ध। RIBO-seq पुस्तकालय की तैयारी के लिए, आरएनए को एमएनएई (ए) के साथ पचाया जाता है, जिसके बाद ~ 28-30 एनटी लंबाई (बी) के आरएफ का आकार चयन होता है; आरएनए-एसईक्यू आरएनए के लिए अलग-थलग (सी), आरएनए (डी) से समाप्त हो गया है, और परिणामस्वरूप एमआरएनए बेतरतीब ढंग से अलग-अलग लंबाई (ई) के टुकड़ों में खंडित हो जाता है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
RIBO-seq और आरएनए-seq के लिए नमूना तैयार करने की प्रक्रिया के प्रारंभिक चरण थोड़ा(चित्रा 1)भिन्न हैं। राइबोसोम्स प्रोफाइलिंग के लिए, लाइसेट को राइबोसोम्स द्वारा संरक्षित नहीं एमआरएनए अणुओं को नीचा दिखाने के लिए एक विशिष्ट एंडोन्यूक्लियस द्वारा पचाने की आवश्यकता होती है। मानक प्रोटोकॉल में, प्राप्त मोनोसोम्स को सुक्रोज कुशन अल्ट्रासेंट्रफ्यूगेशन या सुक्रोज रेडिएंट अल्ट्रासेंट्रफ्यूजेशन8,14द्वारा बरामद किया जाता है। इस लेख में, हम बताते हैं कि यह कदम बैक्टीरिया में RIBO-seq के लिए आवश्यक आरएफ को अलग करने के लिए आवश्यक नहीं है, इसी तरह यूकेरियोटिक कोशिकाओं18के लिए, और पॉलीएक्रीलामाइड जेल से उचित लंबाई एमआरएनए टुकड़ों का आकार चयन पर्याप्त है।
आरएनए-सेक्यू के लिए, एमआरएनए को कुल आरएनए से आरएनए की कमी से प्राप्त किया जाता है – आरएनए अणु बायोटिनाइलेटेड ओलिगोन्यूक्लियोटाइड प्रोब्स को संकरित करते हैं जो स्ट्रेप्टाविडिन-लेपित चुंबकीय मोतियों से बांधते हैं। इसके बाद आरआरएनए-ओलिगोन्यूक्लियोटाइड-मनका परिसरों को एक चुंबक के साथ नमूने से हटा दिया जाता है जिसके परिणामस्वरूप आरआरएनए का नमूना19,20समाप्त हो जाता है । शुद्ध mRNA अणुओं तो बेतरतीब ढंग से क्षारीय हाइड्रोलिसिस द्वारा खंडित कर रहे हैं । एमआरएनए के प्राप्त टुकड़ों के साथ-साथ रिबोसोमल पैरों के निशान सीडीएनए पुस्तकालयों में परिवर्तित हो जाते हैं और गहरे अनुक्रमण(चित्रा 2)के लिए तैयार होते हैं। इसमें क्षारीय हाइड्रोलिसिस (एमआरएनए के लिए) और एंजाइमेटिक पाचन (आरएफ के लिए) के बाद आवश्यक मरम्मत समाप्त होती है: 3 के डीफोस्फोरिलेशन 5 ‘ सिरों के फॉस्फोरिलेशन के बाद समाप्त होता है। अगले कदम इलुमिना प्लेटफॉर्म का उपयोग करके अगली पीढ़ी के अनुक्रमण (एनजीएस) के लिए आवश्यक दृश्यों द्वारा तैयार सीडीएनए आवेषण बनाने के लिए एडाप्टर लिगेशन और रिवर्स ट्रांसक्रिप्शन हैं। पुस्तकालय तैयार करने का अंतिम चरण एक पीसीआर प्रतिक्रिया है जिसमें निर्माणों को एक चैनल पर मल्टीप्लेक्सिंग और अनुक्रमण की अनुमति देने के लिए नमूना विशिष्ट बारकोड के साथ परिलक्षित और लेबल किया जाता है। अनुक्रमण से पहले, पुस्तकालयों की गुणवत्ता और मात्रा का मूल्यांकन उच्च संवेदनशीलता वाले डीएनए ऑन-चिप इलेक्ट्रोफोरेसिस द्वारा किया जाता है। उचित मापदंडों के साथ सीडीएनए पुस्तकालयों को फिर पूल और अनुक्रमित किया जा सकता है। विभिन्न इलुमिना प्लेटफार्मों पर अनुक्रमण किया जा सकता है, जैसे कि मिसेक़, नेक्स्टसेक़ या हाईसेक़, पुस्तकालयों की संख्या के आधार पर, आवश्यक अवधि और अनुक्रमण गहराई। अनुक्रमण के बाद, जैव सूचना विश्लेषण किया जाता है।

चित्रा 2। पुस्तकालय की तैयारी। पुस्तकालय की तैयारी में बार्कोडिंग के साथ सिरों की मरम्मत, एडाप्टर लिगेशन, रिवर्स ट्रांसक्रिप्शन और प्रवर्धन शामिल है। कृपया इस आंकड़े का एक बड़ा संस्करण देखने के लिए यहां क्लिक करें ।
राइबोसोम प्रोफाइलिंग एक सार्वभौमिक विधि है जिसे वैज्ञानिक प्रश्न के अनुसार आसानी से संशोधित और समायोजित किया जा सकता है। मूल रूप से इसका उपयोग खमीर1में किया जाता था , लेकिन कुछ ही समय बाद इसे बैक्टीरियल कोशिकाओं21 के साथ – साथ यूकेरियोटिक मॉडल जीवों पर लागू किया गया, जिसमें माउस10, जेब्राफिश22, फ्रूट फ्लाई23 और अरबीडोप्सिस थैलियाना24शामिल हैं । इसका उपयोग विभिन्न राइबोसोम प्रकारों का अध्ययन करने के लिए भी किया जाता था: साइटोप्लाज्मिक, माइटोकॉन्ड्रियल25,26 और क्लोरोप्लास्ट27,28। यूकेरियोट्स आरआईबीओ-सेक़ में अनुवाद के विशिष्ट पहलुओं की जांच करने के लिए आमतौर पर अनुकूलित और परिष्कृत किया जाता है, जिसमें दीक्षा10,11,29,30,32,विस्तार1,10,11,31,33,रिबोसोम स्टालिंग33 और अनुरूपता परिवर्तन33शामिल हैं। अधिकांश संशोधनों में विभिन्न अनुवाद अवरोधकों का उपयोग शामिल है। बैक्टीरिया में हालांकि, कार्रवाई34के आवश्यक तंत्र के साथ अवरोधकों की कमी के कारण अनुरूप अध्ययन ों का संचालन करना मुश्किल हो गया है। बैक्टीरिया में सबसे अधिक उपयोग किया जाने वाला अनुवाद अवरोधक क्लोरम्फेनिकोल (सीएएम) है जो पेप्टिडिल ट्रांसफरेस सेंटर (पीटीसी) से बांधता है और ए-साइट में अमीनोएसिल-टर्ना की सही स्थिति को रोकता है। नतीजतन, सीएएम पेप्टाइड बॉन्ड के गठन को रोकता है जो लम्बी राइबोसोम्स35को गिरफ्तार करता है। बैक्टीरिया में अनुवाद अवरोधकों के अन्य उदाहरण टेट्रासाइक्लिन (टीईटी)36,रेटापमुलिन (आरईटी)34 और ओन्क 11237 हैं जिनका उपयोग अनुवाद दीक्षा स्थलों की जांच के लिए किया गया है। टीईटी, जो ए-साइट पर टीएनएआर के एंटीकोडन स्टेम-लूप के साथ सीधे ओवरलैपिंग करके राइबोसोम को टीआरएनए डिलीवरी को रोकता है, मूल रूप से सीएएम उपचार से प्राप्त परिणामों को सत्यापित करने के लिए लागू किया गया था क्योंकि वे दोनों एंटीबायोटिक्स अनुवाद विस्तार38को रोकते हैं। टीईटी प्राथमिक TIS का पता लगाने के लिए पाया गया था, लेकिन आंतरिक TIS३६प्रकट करने में असमर्थ था । आरईटी बैक्टीरियल राइबोसोम के पीटीसी में बांधता है, और एक साइट में एक elongator अमीनोएसिल-tRNA के साथ हस्तक्षेप करके पहले पेप्टाइड बांड के गठन को रोकता है । आरईटी लागू करने से प्राथमिक और आंतरिक तीएसएस34दोनों में राइबोसोम्स की गिरफ्तारी होती है . Onc112, एक प्रोलाइन-समृद्ध एंटीमाइक्रोबियल पेप्टाइड, निकास सुरंग में बांधता है और राइबोसोमल ए साइट में अमीनोएसिल-टर्ना बाध्यकारी को अवरुद्ध करता है। नतीजतन, Onc112 दीक्षा परिसरों को विस्तार चरण37में प्रवेश करने से रोकता है।
मुख्य जानकारी राइबोसोम प्रोफाइलिंग प्रदान करता है राइबोसोम घनत्व और एमएनए पर उनकी स्थिति है। इसे विभिन्न विकास स्थितियों 1 , 6 में अनुवाद के स्तर पर अंतर जीन अभिव्यक्ति की जांच करने , अनुवादात्मक दक्षता1,38,39 को मापने और रिबोसोमल रुकेहुए 10जैसे अनुवाद विनियमन कार्यक्रमों का पता लगाने के लिए सफलतापूर्वक लागू किया गया था। RIBO-seq एनोटेटेड एनसीआरएनए, छद्मजीन और बिना एनएनएनोट किए गए छोटे खुले पठन फ्रेम (ओआरएफ) के अनुवाद को उजागर करने की भी अनुमति देता है जिससे उपन्यास और/या बहुत कम प्रोटीन कोडिंगजीन10,12, 22,30, ३७की पहचान होती है । ऐसे मामलों में, RIBO-seq ठीक धुन और जीनोम एनोटेशन में सुधार कर सकते हैं । अनुवादित ओआरएफ और इसकी मात्रात्मक प्रकृति की पहचान के लिए अपनी उच्च संवेदनशीलता के साथ, राइबोसोम प्रोफाइलिंग प्रोटेम निर्धारण के लिए या प्रोटेओमिक्स अध्ययन 31 ,34,39के लिए प्रॉक्सी के रूप में भी काम कर सकती है। टीआई मैप करके, राइबोसोम प्रोफाइलिंग से पता चलता है कि एन-मरणासन्न विस्तारित और ज्ञात प्रोटीन10,32के कटा हुआ आइसोफॉर्म। रीबो-सेक को प्रोटीन14 , 21,24के सह-अनुवादीय तह का अध्ययन करने के लिए भी अनुकूलित किया गयाथा। यह विधि विस्तार दर 1,10,39 या अलग – अलग कोडन 6 की डिकोडिंग गति को मापने में सक्षम बनाती है और अनुवाद17के मात्रात्मक मॉडल विकसित करने में मदद करती है । राइबोसोमप्रोफाइलिंग विधि भी बैक्टीरिया 7,15,17,फ्रेमशिफ्टिंग40,स्टॉप-कोडन रीडथ्रू के माध्यम से21,टर्मिनेशन/रीसाइक्लिंग दोष41, 42 और रिबोसोमल संरचना परिवर्तन33 में रिकेरियोट में मेकेनिस्टिक अंतर्दृष्टि प्रदान करने में सक्षम है। यूकेरियोट्स16,43में मिरनास6 और आरएनए-बाइंडिंग प्रोटीन जैसे अनुवाद पर विशिष्ट ट्रांस-एक्टिंग कारकों के प्रभाव की जांच करने के लिए RIBO-seq को भी अनुकूलित किया गया था। हालांकि, यह स्वीकार करना महत्वपूर्ण है कि प्रायोगिक डिजाइन और RIBO-seq का प्राप्त संकल्प जानकारी की मात्रा निर्धारित करता है जो परिणामी अनुक्रमण डेटा12से निकाला जा सकता है।
Protocol
Representative Results
Discussion
राइबोसोम प्रोफाइलिंग की प्रमुख तकनीकी चुनौती एक विशेष शारीरिक स्थिति में mRNAs पर राइबोसोम्स के स्नैपशॉट पर कब्जा करने के लिए तेजी से अनुवाद को बाधित करने की आवश्यकता है। इसे पूरा करने के लिए, अनुवाद अवर?…
Divulgazioni
The authors have nothing to disclose.
Acknowledgements
एएलएस एम्बो इंस्टॉलेशन ग्रांट आईजी 3914 और पीओआईआर की वित्तीय सहायता को स्वीकार करना चाहेगा। 04.04.00-00-3E9C/17-00 यूरोपीय क्षेत्रीय विकास कोष के तहत यूरोपीय संघ द्वारा सह-वित्तपोषित पोलिश विज्ञान के लिए फाउंडेशन की पहली टीम कार्यक्रम के भीतर किया गया ।
Materials
| 10X TBE (powder) | Invitrogen | AM9864 | |
| 2-Mercaptoethanol, 99%, pure | Acros Organics | 125472500 | |
| Adenosine 5'-Triphosphate (ATP) | New England Biolabs | P0756S | |
| Aluminium oxide calcinated pure p.a. | Chempur | 114560600 | |
| Calcium chloride dihydrate | Sigma-Aldrich | C3881-500G | |
| Chloramphenicol | MP Biomedicals | 190321 | |
| DNA Clean & Concentrator -5 | Zymo Research | D4004 | |
| Dnase I recombinant, Rnase-free | Roche | 4716728001 | |
| EDTA disodium salt | Fisher Scientific | E/P140/48 | |
| Ethyl Alcohol Absolut 99,8% Pure-P.A.-Basic | POCH Avantor Performance Materials Poland S.A | BA6480111 | |
| Filtration apparatus | VWR Collection | 511-0265 | all-glass filtration apparatus, with funnel, fritted base, cap, 47 mm Ø spring clamp and ground joint flask |
| Gel 40 (19:1) | Rotiphorese | 3030.1 | |
| Gel Loading Dye, Blue, 6X | New England Biolabs | E6138G | |
| Guanosine 5′-[β,γ-imido]triphosphate trisodium salt hydrate | Sigma-Aldrich | G0635-25MG | |
| labZAP | A&A Biotechnology | 040-500 | |
| Magnesium acetate tetrahydrate | Sigma-Aldrich | M5661-250G | |
| MCE membrane fiter | Alfatec Technology | M47MCE45GWS | pore size: 0.45um |
| MICROBExpress Bacterial mRNA Purification | Invitrogen | AM1905 | |
| Multiplex Small RNA Library Prep Set for Illumina | New England Biolabs | E7300S | |
| Nuclease-Free Water | Ambion | AM9937 | |
| Potassium Acetate Anhydrous Pure P.A. | POCH Avantor Performance Materials Poland S.A | 744330113 | |
| Quick-Load pBR322 DNA-MspI Digest | New England Biolabs | E7323A | |
| RNA Clean & Concentrator -25 | Zymo Research | R1018 | |
| Sodium acetate | Sigma-Aldrich | S2889-250G | |
| Sodium carbonate | Sigma-Aldrich | 223530-500G | |
| Sodium hydrogen carbonate pure p.a. | POCH Avantor Performance Materials Poland S.A | 810530115 | |
| SYBR Gold nucleic acid gel stain | Life Technologies | S11494 | |
| T4 Polynucleotide Kinase | New England Biolabs | M0201L | |
| T4 Polynucleotide Kinase Reaction Buffer | New England Biolabs | B0201S | |
| TBE-Urea Sample Buffer (2x) | Invitrogen | LC6876 | |
| Tris(hydroxymethyl)amino-methane, ultrapure, 99,9% | AlfaAesar | J65594 | |
| Triton X-100, 98% | Acros Organics | 327371000 | |
| Urea G.R. | lach:ner | 40096-AP0 |
Riferimenti
- Ingolia, N. T., Ghaemmaghami, S., Newman, J. R., Weissman, J. S. Genome-wide analysis in vivo of translation with nucleotide resolution using ribosome profiling. Science. 324 (5924), 218-223 (2009).
- Takanami, M., Yan, Y., Jukes, T. H. Studies on the site of ribosomal binding of f2 bacteriophage RNA. Journal of Molecular Biology. 12 (3), 761-773 (1965).
- Steitz, J. A. Polypeptide Chain Initiation: Nucleotide Sequences of the Three Ribosomal Binding Sites in Bacteriophage R17 RNA. Nature. 224 (5223), 957-964 (1969).
- Wolin, S. L., Walter, P. Ribosome pausing and stacking during translation of a eukaryotic mRNA. The EMBO journal. 7 (11), 3559-3569 (1988).
- Argüello, R. J., et al. SunRiSE – measuring translation elongation at single-cell resolution by means of flow cytometry. Journal of Cell Science. 131 (10), 214346 (2018).
- Michel, A. M., Baranov, P. V. Ribosome profiling: a Hi-Def monitor for protein synthesis at the genome-wide scale. Wiley Interdisciplinary Reviews: RNA. 4 (5), 473-490 (2013).
- Woolstenhulme, C. J., Guydosh, N. R., Green, R., Buskirk, A. R. High-precision analysis of translational pausing by ribosome profiling in bacteria lacking EFP. Cell reports. 11 (1), 13-21 (2015).
- McGlincy, N. J., Ingolia, N. T. Transcriptome-wide measurement of translation by ribosome profiling. Methods (San Diego, Calif). 126, 112-129 (2017).
- Gerashchenko, M. V., Gladyshev, V. N. Translation inhibitors cause abnormalities in ribosome profiling experiments. Nucleic acids research. 42 (17), 134 (2014).
- Ingolia, N. T., Lareau, L. F., Weissman, J. S. Ribosome Profiling of Mouse Embryonic Stem Cells Reveals the Complexity of Mammalian Proteomes. Cell. 147 (4), 789-802 (2011).
- Ingolia, N. T., Brar, G. A., Rouskin, S., McGeachy, A. M., Weissman, J. S. The ribosome profiling strategy for monitoring translation in vivo by deep sequencing of ribosome-protected mRNA fragments. Nature protocols. 7 (8), 1534-1550 (2012).
- Hsu, P. Y., et al. Super-resolution ribosome profiling reveals unannotated translation events in Arabidopsis. Proceedings of the National Academy of Sciences of the United States of America. 113 (45), 7126-7135 (2016).
- Blanchard, S. C., Kim, H. D., Gonzalez, R. L., Puglisi, J. D., Chu, S. tRNA dynamics on the ribosome during translation. Proceedings of the National Academy of Sciences of the United States of America. 101 (35), 12893-12898 (2004).
- Becker, A. H., Oh, E., Weissman, J. S., Kramer, G., Bukau, B. Selective ribosome profiling as a tool for studying the interaction of chaperones and targeting factors with nascent polypeptide chains and ribosomes. Nature protocols. 8 (11), 2212-2239 (2013).
- Li, G. W., Oh, E., Weissman, J. S. The anti-Shine-Dalgarno sequence drives translational pausing and codon choice in bacteria. Nature. 484 (7395), 538-541 (2012).
- King, H. A., Gerber, A. P. Translatome profiling: methods for genome-scale analysis of mRNA translation. Briefings in functional genomics. 15 (1), 22-31 (2014).
- Mohammad, F., Woolstenhulme, C. J., Green, R., Buskirk, A. R. Clarifying the Translational Pausing Landscape in Bacteria by Ribosome Profiling. Cell reports. 14 (4), 686-694 (2016).
- Reid, D. W., Shenolikar, S., Nicchitta, C. V. Simple and inexpensive ribosome profiling analysis of mRNA translation. Methods (San Diego, Calif). 91, 69-74 (2015).
- Sanz, E., Yang, L., Su, T., Morris, D. R., McKnight, G. S., Amieux, P. S. Cell-type-specific isolation of ribosome-associated mRNA from complex tissues. Proceedings of the National Academy of Sciences of the United States of America. 106 (33), 13939-13944 (2009).
- Petrova, O. E., Garcia-Alcalde, F., Zampaloni, C., Sauer, K. Comparative evaluation of rRNA depletion procedures for the improved analysis of bacterialbiofilm and mixed pathogen culture transcriptomes. Scientific Reports. 7, 41114 (2017).
- Oh, E. Selective ribosome profiling reveals the cotranslational chaperone action of trigger factor in vivo. Cell. 147 (6), 1295-1308 (2011).
- Chew, G. L. Ribosome profiling reveals resemblance between long non-coding RNAs and 5′ leaders of coding RNAs. Development. 140 (13), 2828-2834 (2013).
- Dunn, J. G., Foo, C. K., Belletier, N. G., Gavis, E. R., Weissman, J. S. Ribosome profiling reveals pervasive and regulated stop codon readthrough in Drosophila melanogaster. eLife. 2, 01179 (2013).
- Liu, M. J., et al. Translational landscape of photomorphogenic Arabidopsis. The Plant cell. 25 (10), 3699-3710 (2013).
- Rooijers, K., Loayza-Puch, F., Nijtmans, L. G., Agami, R. Ribosome profiling reveals features of normal and disease-associated mitochondrial translation. Nature Communications. 4, 2886 (2013).
- Danielle, L., et al. Fidelity of translation initiation is required for coordinated respiratory complex assembly. Science Advances. 5 (12), 2118 (2019).
- Zoschke, R., Watkins, K. P., Barkan, A. A rapid ribosome profiling method elucidates chloroplast ribosome behavior in vivo. The Plant cell. 25 (6), 2265-2275 (2013).
- Chotewutmontri, P., Barkan, A. Dynamics of chloroplast translation during chloroplast differentiation in maize. PLoS genetics. 12 (7), 1006106 (2016).
- Lee, S., Liu, B., Huang, S. X., Shen, B., Qian, S. B. Global mapping of translation initiation sites in mammalian cells at single-nucleotide resolution. Proceedings of the National Academy of Sciences of the United States of America. 109 (37), 2424-2432 (2012).
- Martinez, T. F., Chu, Q., Donaldson, C., Tan, D., Shokhirev, M. N., Saghatelian, A. Accurate annotation of human protein-coding small open reading frames. Nature chemical biology. 16 (4), 458-468 (2020).
- Simsek, D., et al. The Mammalian Ribo-interactome Reveals Ribosome Functional Diversity and Heterogeneity. Cell. 169 (6), 1051-1065 (2017).
- Fritsch, C., et al. Genome-wide search for novel human uORFs and N-terminal protein extensions using ribosomal footprinting. Genome research. 22 (11), 2208-2218 (2012).
- Lareau, L. F., Hite, D. H., Hogan, G. J., Brown, P. O. Distinct stages of the translation elongation cycle revealed by sequencing ribosome-protected mRNA fragments. eLife. 3, 01257 (2014).
- Meydan, S., et al. Retapamulin-Assisted Ribosome Profiling Reveals the Alternative Bacterial Proteome. Molecular Cell. 74 (3), 481-493 (2019).
- Wilson, D. N. The A-Z of bacterial translation inhibitors. Critical Reviews in Biochemistry and Molecular Biology. 44, 393-433 (2009).
- Nakahigashi, K. Comprehensive identification of translation start sites by tetracycline-inhibited ribosome profiling. DNA research : an international journal for rapid publication of reports on genes and genomes. 23 (3), 193-201 (2016).
- Weaver, J., Mohammad, F., Buskirk, A. R., Storz, G. Identifying Small Proteins by Ribosome Profiling with Stalled Initiation Complexes. mBio. 10 (2), 02819 (2019).
- Nakahigashi, K. Effect of codon adaptation on codon-level and gene-level translation efficiency in vivo. BMC genomics. 15 (1), 1115 (2014).
- Li, G. W., Burkhardt, D., Gross, C., Weissman, J. S. Quantifying absolute protein synthesis rates reveals principles underlying allocation of cellular resources. Cell. 157 (3), 624-635 (2014).
- Michel, A. M., et al. Observation of dually decoded regions of the human genome using ribosome profiling data. Genome research. 22 (11), 2219-2229 (2012).
- Guydosh, N. R., Green, R. Dom34 rescues ribosomes in 3′ untranslated regions. Cell. 156 (5), 950-962 (2014).
- Young, D. J., Guydosh, N. R., Zhang, F., Hinnebusch, A. G., Green, R. Rli1/ABCE1 Recycles Terminating Ribosomes and Controls Translation Reinitiation in 3’UTRs In Vivo. Cell. 162 (4), 872-884 (2015).
- Ingolia, N. T. Ribosome profiling: new views of translation, from single codons to genome scale. Nature Reviews Genetics. 15 (3), 205-213 (2014).
- Dobin, A., et al. ultrafast universal RNA seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- . . Babraham Bioinformatics. Fast QC. , (2021).
- Gerashchenko, M. V., Gladyshev, V. N. Ribonuclease selection for ribosome profiling. Nucleic acids research. 45 (2), 6 (2017).
- Verbruggen, S., Menschaert, G. mQC: A post-mapping data exploration tool for ribosome profiling. Computer Methods and Programs in Biomedicine. 181, 104806 (2019).
- Cui, H., Hu, H., Zeng, J., Chen, T. DeepShape: estimating isoform-level ribosome abundance and distribution with Ribo-seq data. BMC bioinformatics. 20, 678 (2019).
- Choi, J. . RiboToolkit: an integrated platform for analysis and annotation of ribosome profiling data to decode RNA translation at codon resolution. , (2021).
- Choi, J. Dynamics of the context-specific translation arrest by chloramphenicol and linezolid. Nature chemical biology. 16 (3), 310-317 (2020).
- Tompson, J., O’Connor, M., Mills, J. A., Dahlberg, A. E. The protein synthesis inhibitors, oxazolidinones and chloramphenicol, cause extensive translational inaccuracy in vivo. Journal of Molecular Biology. 322 (2), 273-279 (2002).
- Brar, G. A., Weissman, J. S. Ribosome profiling reveals the what, when, where and how of protein synthesis. Nature reviews. Molecular cell biology. 16 (11), 651-664 (2015).
- Glaub, A., Huptas, C., Neuhaus, K., Ardern, Z. Recommendations for bacterial ribosome profiling experiments based on bioinformatic evaluation of published data. Journal of Biological Chemistry. 295, 8999-9011 (2020).
- Sorour, M. H., Hani, H. A., Shaalan, H. F., El-Sayed, M. M. H. Experimental screening of some chelating agents for calcium and magnesium removal from saline solutions. Desalination and Water Treatment. 57 (48-49), 22799-22808 (2015).
- Mohammad, F., Green, R., Buskirk, A. R. A systematically-revised ribosome profiling method for bacteria reveals pauses at single-codon resolution. Elife. 8, 42591 (2019).
- O’Connor, P. B., Li, G. W., Weissman, J. S., Atkins, J. F., Baranov, P. V. rRNA:mRNA pairing alters the length and the symmetry of mRNA-protected fragments in ribosome profiling experiments. Bioinformatics. 29 (12), 1488-1491 (2013).
- Protocol for RNA Clean & Concentrator -25. Zymo Research Available from: https://files.zymoresearch.com/protocols/_r1017_r1018_rna_clean_concentrator-25.pdf (2021)
- Protocol for use with NEBNext Small RNA Library Prep Set for Illumina (E7300, E7580, E7560, E7330). New England Biolabs Available from: https://www.international.neb.com/protocols/2018/03/27/protocol-for-use-with-nebnext-small-rna-library-prep-set-for-illumina-e7300-e7580-e7560-e7330 (2021)

